抗生素抗性基因(antibiotic resistant genes, ARGs)在环境中的广泛传播、迁移和扩散已成为全球最大的环境和卫生健康问题之一[1]。如果微生物耐药性传播得不到有效控制,预计到2050年将有1000万人死于抗生素耐药性引起的感染[2]。仅2019年抗生素耐药性就导致了127万人的直接死亡以及495万人的间接死亡,成为仅次于心脏病和中风的全球第三大死亡病因[3]。世界卫生组织(World Health Organization, WHO)确认抗生素耐药性问题是21世纪经济发展和生态安全的最大威胁之一,为遏制细菌耐药发展与蔓延,多个国家和全球公共卫生组织相继制定相应防控计划,并一致认为追踪ARGs的出现和流行对于最大限度地减少对人类健康的威胁至关重要[4-7]。
2006年,Pruden等[8]正式提出ARGs是一类新型环境污染物,环境中的ARGs没有得到普遍检测,在细菌耐药方面发挥了重要作用。在环境中,ARGs具有持久性、可移动性和可传导性,ARGs从非致病菌到病原体的传播可能比抗生素本身产生更大的生态毒理效应和环境健康风险[9]。过去,研究人员关注的重点是ARGs在临床领域的影响,ARGs在污水处理厂和土壤中的污染和环境风险等[10-12]。随着研究的不断深入,探究ARGs对海洋环境的影响也是至关重要的,海洋环境不仅是ARGs的储存库,也是ARGs增殖、迁移和扩散的重要介质[13-14]。
研究表明,环境中含有ARGs的抗生素抗性细菌(antibiotic resistant bacteria, ARB)的增殖将增加ARGs在环境微生物中的迁移、转化和扩散,最终导致环境成为各种ARGs的天然储存库[15]。近年来在近海养殖系统水体及沉积物、水生生物以及病原体、水产品批发市场和零售市场等介质中普遍检出ARGs,包括四环素类抗性基因、喹诺酮类抗性基因、大环内酯类抗性基因、氨基糖苷类抗性基因、氯霉素类抗性基因、β-内酰胺类抗性基因和磺胺类抗性基因等[16-18]。这些研究初步探明了水环境中ARGs的存在类型、多样性和污染分布状况。然而,有关近岸海洋环境中ARGs的分布特征、来源以及环境健康风险仍缺乏系统和深入的研究。
面对区域性和全球性的抗生素耐药性挑战,本文结合国内外最新研究成果,综述了近岸海洋环境中ARGs的分布特征、来源、传播扩散途径以及影响因素和潜在健康风险,为评估近岸海域ARGs的潜在危害和健康风险提供基本参考,如图1所示。
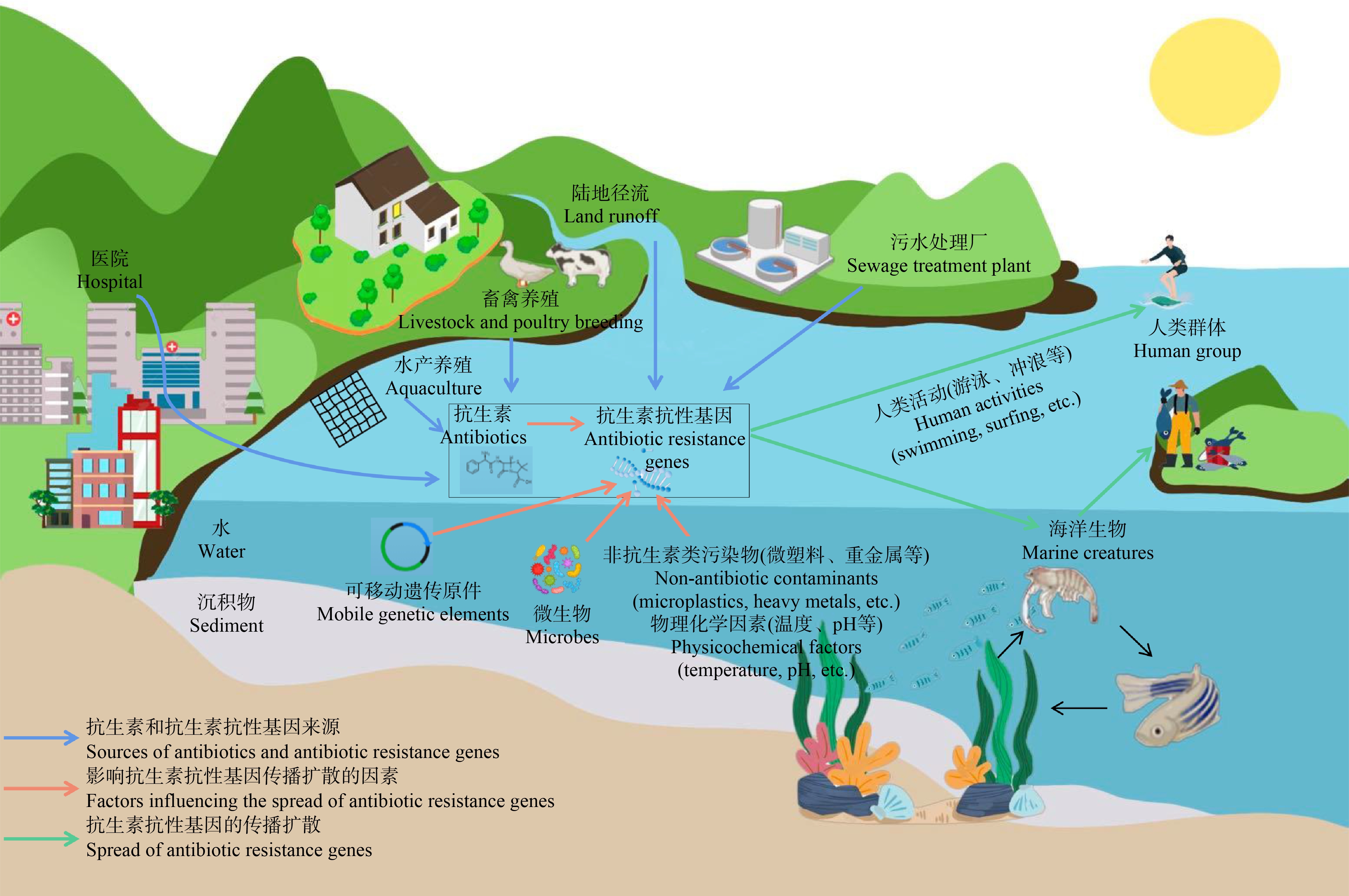
图1 海洋环境中抗生素抗性基因(ARGs)的主要来源和传播扩散
Fig. 1 Major sources and spread of antibiotic resistance genes(ARGs)in the marine environment
1 海洋环境中ARGs的分布特征(Distribution characteristics of ARGs in marine environment)
1.1 近岸海洋环境中ARGs分布基本概况
近年来,随着细菌分离培养、荧光定量PCR和宏基因组测序等技术在海洋调查中的广泛应用,在国内外典型河口、沿海地区以及周围海洋等不同环境介质中广泛检出多种类型的ARGs,其中,磺胺类、四环素类和喹诺酮类抗性基因的检出率和检测丰度较高,磺胺类抗性基因检出频率最高。国内外近岸海域ARGs的分布和丰度如表1所示。我国长江口水体中磺胺类抗性基因(sul1、sul2)、四环素类抗性基因(tetA、tetQ和tetW)和氟喹诺酮类抗性基因(aac(6’)-Ib)的检出率均为100%,磺胺类抗性基因sul1是优势ARGs,丰度介于2.26×104~3.19×105copies·mL-1之间,四环素类抗性基因中tetQ丰度最高(2.23×104copies·mL-1),aac(6’)-Ib丰度范围是2.68×104~3.05×104copies·mL-1[19]。我国珠江口水体中磺胺类抗性基因含量最丰富的是sul1和sul2,丰度范围是104~106copies·mL-1[20]。Niu等[21]在我国渤海湾海域调查了10种ARGs的分布,发现磺胺类抗性基因sul1和sul2在海水和沉积物中的检出率最高,且丰度高于其他ARGs,水体中丰度为10-5~10-3copies(相对于标记基因16S rRNA);沉积物中丰度为10-4~10-2copies(相对于标记基因16S rRNA),tetM是四环素类抗性基因中最常见、相对丰度最高的ARGs,平均丰度为5.15×10-5copies(相对于标记基因16S rRNA)。Suzuki等[22]发现磺胺类抗性基因sul1、sul2和sul3在菲律宾马尼拉湾海洋细菌中广泛存在。Lu等[23]在我国福建省沿海循环水养殖系统中共检出10种ARGs,磺胺类抗性基因(sul1和sul2)为主,其次是四环素类抗性基因(tetX)和质粒介导的喹诺酮类抗性基因(qnrA),sul1、sul2、tetX和qnrA的绝对丰度分别为6.50×103~3.31×104copies·mL-1、1.26×105~5.17×105copies·mL-1、9.17×104~2.09×105copies·mL-1和1.67×104~9.69×104copies·mL-1。韩国沿海地区四环素类抗性基因tetM含量丰富,其丰度高达4.34×102~1.10×108copies·mL-1[24]。Lu等[25]在我国黄海和渤海海域共检出10种类型的ARGs,其中磺胺类抗性基因在黄海和渤海的丰度最高(sul1最高丰度达2.13×103copies·mL-1;sul2最高丰度达6.23×103copies·mL-1),其次是四环素和喹诺酮类抗性基因。以上结果表明近岸海域已成为ARGs的大型储存库。
表1 近岸海洋环境和极地海域主要ARGs丰度
Table 1 Major ARGs abundance in nearshore marine environments and Arctic and Antarctic polar marine environments
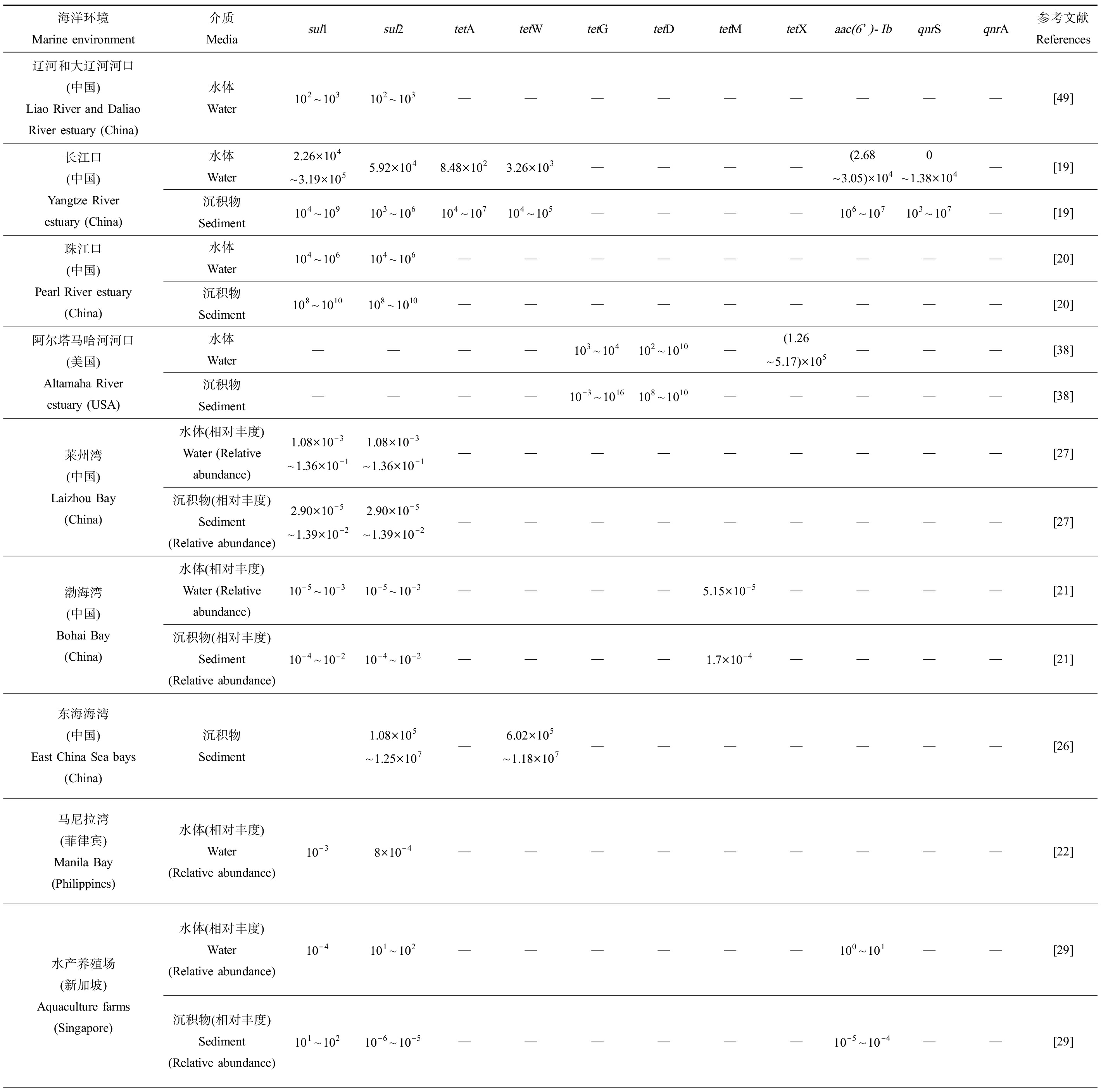
海洋环境Marine environment介质Mediasul1sul2tetAtetWtetGtetDtetMtetXaac(6’)-IbqnrSqnrA参考文献References辽河和大辽河河口(中国)Liao River and Daliao River estuary (China)水体Water102~103102~103—————————[49]长江口(中国)Yangtze River estuary (China)水体Water2.26×104~3.19×1055.92×1048.48×1023.26×103————(2.68~3.05)×1040~1.38×104—[19]沉积物Sediment104~109103~106104~107104~105————106~107103~107—[19]珠江口(中国)Pearl River estuary (China)水体Water104~106104~106—————————[20]沉积物Sediment108~1010108~1010—————————[20]阿尔塔马哈河河口(美国)Altamaha River estuary (USA)水体Water————103~104102~1010—(1.26~5.17)×105———[38]沉积物Sediment————10-3~1016108~1010—————[38]莱州湾(中国) Laizhou Bay (China)水体(相对丰度)Water (Relative abundance)1.08×10-3~1.36×10-11.08×10-3~1.36×10-1—————————[27]沉积物(相对丰度)Sediment (Relative abundance)2.90×10-5~1.39×10-22.90×10-5~1.39×10-2—————————[27]渤海湾(中国) Bohai Bay (China)水体(相对丰度)Water (Relative abundance)10-5~10-310-5~10-3————5.15×10-5————[21]沉积物(相对丰度)Sediment (Relative abundance)10-4~10-210-4~10-2————1.7×10-4————[21]东海海湾(中国)East China Sea bays (China)沉积物Sediment1.08×105~1.25×107—6.02×105~1.18×107———————[26]马尼拉湾(菲律宾)Manila Bay (Philippines) 水体(相对丰度)Water (Relative abundance)10-38×10-4—————————[22]水产养殖场(新加坡) Aquaculture farms (Singapore)水体(相对丰度)Water (Relative abundance)10-4101~102——————100~101——[29]沉积物(相对丰度)Sediment (Relative abundance)101~10210-6~10-5——————10-5~10-4——[29]
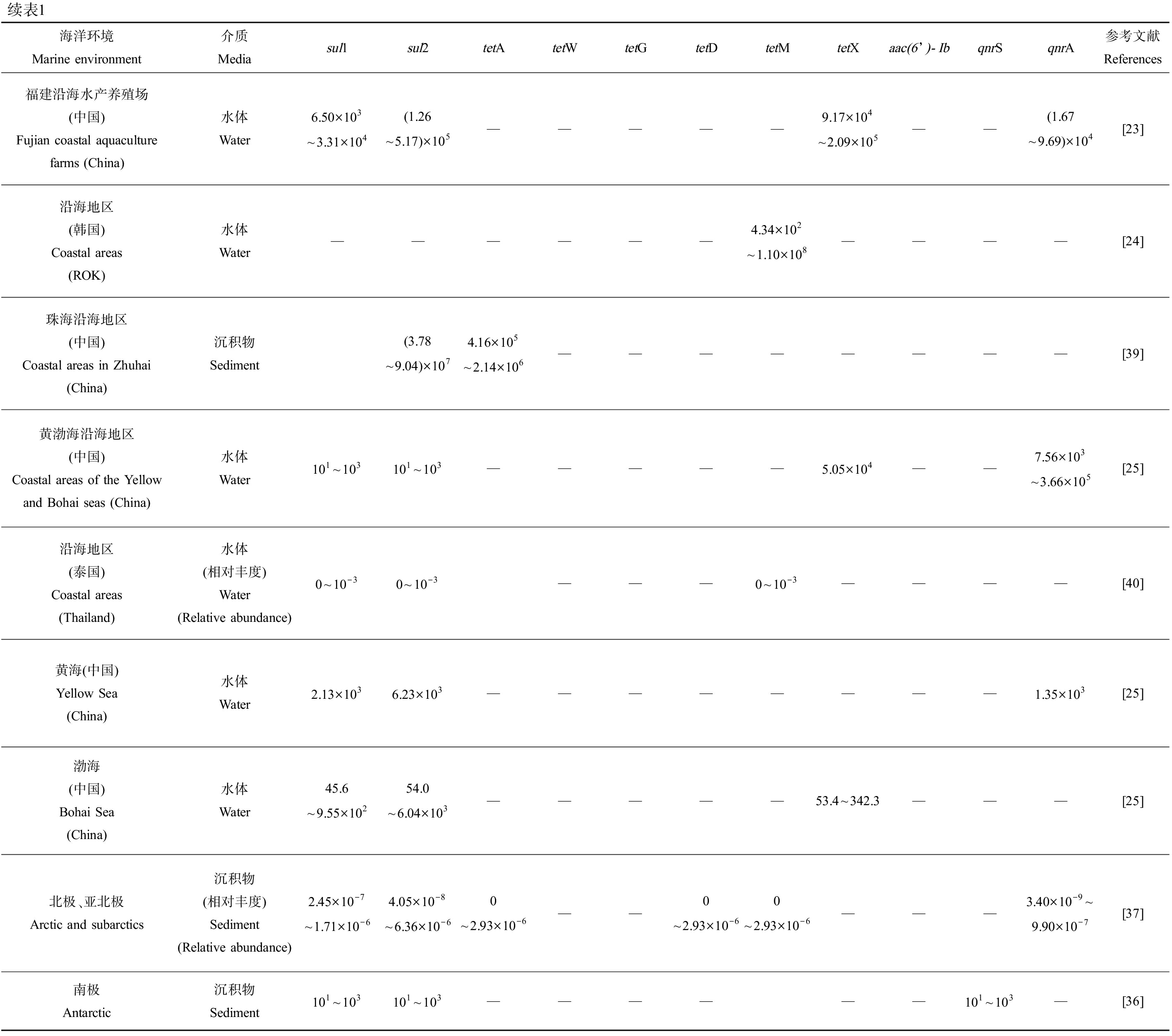
续表1海洋环境Marine environment介质Mediasul1sul2tetAtetWtetGtetDtetMtetXaac(6’)-IbqnrSqnrA参考文献References福建沿海水产养殖场(中国)Fujian coastal aquaculture farms (China)水体Water6.50×103~3.31×104(1.26~5.17)×105—————9.17×104~2.09×105——(1.67~9.69)×104[23]沿海地区(韩国)Coastal areas(ROK)水体Water——————4.34×102~1.10×108————[24]珠海沿海地区(中国)Coastal areas in Zhuhai(China)沉积物Sediment(3.78~9.04)×1074.16×105~2.14×106————————[39]黄渤海沿海地区(中国)Coastal areas of the Yellow and Bohai seas (China)水体Water101~103101~103—————5.05×104——7.56×103~3.66×105[25]沿海地区(泰国) Coastal areas(Thailand)水体(相对丰度)Water (Relative abundance)0~10-30~10-3———0~10-3————[40]黄海(中国)Yellow Sea (China)水体Water2.13×1036.23×103————————1.35×103[25]渤海(中国)Bohai Sea (China)水体Water45.6~9.55×10254.0~6.04×103—————53.4~342.3———[25]北极、亚北极Arctic and subarctics沉积物(相对丰度)Sediment (Relative abundance)2.45×10-7~1.71×10-64.05×10-8~6.36×10-60~2.93×10-6——0~2.93×10-60~2.93×10-6———3.40×10-9~9.90×10-7[37]南极Antarctic沉积物Sediment101~103101~103——————101~103—[36]
注:—表示文献中没有相关数据;水体浓度单位为copies·mL-1;沉积物浓度单位为copies·g-1;表中标明相对丰度的单位为copies(相对于标记基因16S rRNA)。
Note:—indicates that there is no relevant data in the article; the unit of water concentration is copies·mL-1; the unit of sediment facies concentration is copies·g-1; the unit of relative abundance is copies(relative to the marker gene 16S rRNA).
1.2 从沿海地区到周边海洋ARGs丰度总体呈下降趋势
Chen等[26]调查了台州湾海域沉积物中11种检测频率最高的ARGs的空间分布,发现ARGs的总绝对丰度在离大陆最近的取样点最高,总绝对丰度由沿岸(1.14×108copies·g-1)向海洋(2.33×107copies·g-1)逐渐减少,其中sul1(绝对丰度从8.05×107copies·g-1降为1.53×106copies·g-1)和sul2(绝对丰度从1.25×107copies·g-1降为1.53×106copies·g-1)丰度变化最大。Lu等[25]检测发现我国黄海、渤海沿海地区及周围海域共10种ARGs的总绝对丰度分别为1.23×104~3.94×105copies·mL-1和21.1~8.00×103copies·mL-1,沿海地区ARGs总丰度比海洋高出1~4个数量级。莱州湾海域水和沉积物中磺胺类抗性基因(sul1和sul2)和喹诺酮类抗性基因(qnrS和aac(6’)-Ib)的丰度从河口到海洋呈现明显的下降趋势[27]。Niu等[21]对天津沿岸海域水和沉积物进行ARGs污染调查,发现ARGs浓度最高的是距离海岸最近的取样点,离海岸最远的点检测到最低的ARGs浓度。Ullah等[28]发现红海沿岸样品中ARGs丰度显著高于周边海域。从沿海地区到周边海洋,人为影响逐渐减弱,这可能是导致ARGs浓度呈明显下降趋势的主要原因。
1.3 近岸海洋环境沉积物中ARGs丰度普遍高于表层水
我国渤海海水样品中测定ARGs总绝对丰度为2.05×102~7.25×103copies·mL-1,沉积物中ARGs总绝对丰度范围为4.67×103~5.41×105copies·g-1,比海水高出1~2个数量级[25]。我国珠江口沉积物中磺胺类抗性基因丰度为108~1010copies·g-1,比海水(104~106copies·g-1)高出2~4个数量级[20]。我国黄海海水样品中测定ARGs总绝对丰度为21.1~8.00×103copies·mL-1,沉积物中ARGs丰度范围为3.88×105~1.08×107copies·g-1,比海水高出4个数量级[25]。Lu等[25]的调查显示我国黄渤海沿岸沉积物中磺胺类抗性基因的丰度是水中的103倍。新加坡沿海水产养殖场沉积物中sul1的丰度为101~102copies(相对于标记基因16S rRNA),而在水体中的丰度仅为10-4copies(相对于标记基因16S rRNA)[29]。以上结果证实ARGs倾向于在沉积物中积累,主要原因可能是与流动性较强的海水相比,沉积物理化性质更稳定,从而成为海洋环境中ARGs的主要储集层。
1.4 生物膜是近岸海洋环境中ARGs的有效载体并增加ARGs的积累
生物膜更容易富集ARGs。Guo等[19]发现长江口天然生物膜中大部分ARGs的检出频率和丰度均高于沉积物和海水,其中sul1、sul2、aac(6’)-Ib、tetA、tetQ、tetW和intI1的检出率达100%,生物膜上最丰富的ARGs是磺胺类sul1,丰度范围为3.20×107~2.50×1011copies·g-1,其次是喹诺酮类aac(6’)-Ib,丰度范围为7.28×106~2.52×109copies·g-1,tetA是生物膜上最丰富的四环素类抗性基因,丰度在2.68×105~4.29×109copies·g-1之间。Guo等[30]的调查表明长江口塑料表面生物膜中sul1、aac(6’)-Ib、tetA、tetW、intI1和Chl等6种ARGs的平均绝对丰度均高于相应采样点的沉积物和水体中的丰度,磺胺类抗性基因sul1、sul2的平均丰度分别为3.68×106~1.14×109copies·g-1、1.01×106~2.75×107copies·g-1,sul1丰度比沉积物高出1~4个数量级,四环素类抗性基因中tetW丰度较高(平均丰度为3.44×108copies·g-1),比沉积物高出2个数量级,喹诺酮类抗性基因aac(6’)-Ib平均丰度高达1.29×109copies·g-1,比沉积物高出4个数量级。我国福建省水产养殖系统中微塑料生物膜上四环素类抗性基因的绝对丰度是水中的2 200倍~6 600倍[23]。Dong等[31]指出生物膜上能够吸附更多的细菌和病毒等微生物,从而积累更多的ARGs。
1.5 海洋生物是ARGs的重要载体
海洋生物中广泛存在ARGs。Hong等[32]对我国东南沿海32种海洋水生动物(收集自5个城市)中ARGs赋存情况进行调查,共检测到18种ARGs,检出频率在3.1%~43.8%之间,其中tetS、floR、aadA、sul1和sul2是主要的ARGs,研究人员发现即便没有靶标抗生素残留(没有选择压力),磺胺类和氯霉素类抗性基因(floR、sul1和sul2)在水生动物中依然持续存在。Muziasari等[33]检测了波罗的海养殖鱼场鱼类的肠道内容物和渔场沉积物的ARGs组成,在肠道内容物中共检测到28种ARGs,其中20种ARGs与农场沉积物中的相同,这表明ARGs可通过鱼类粪便进入渔场沉积物使其在环境中进一步传播扩散。Giatsis等[34]在没有抗生素使用历史的养殖场鱼类肠道中检测到ARGs,推测ARGs可能来自周围水环境中的ARB,ARB与肠道菌群之间发生了基因转移。Wang等[35]在分离的蓝藻菌株中检测出sulA、sul1、sul2、tetA、tetB、qnrB和blaTEM等多种ARGs,检测丰度高于106copies·g-1,蓝藻作为海洋环境中的优势细菌,可能已经成为ARGs在海洋环境中的重要储存库和来源。
1.6 极地海域ARGs的分布特征
在极地海洋环境中检测到ARGs广泛存在并且ARGs的丰度低于低纬度地区。对北极和南极的ARGs污染调查显示,磺胺类抗性基因是极地海洋环境中的优势基因。如表1所示,南极样品中磺胺类和喹诺酮类抗性基因的丰度范围在101~103copies·g-1之间,远低于受人类影响的低纬度地区(如北黄海沿岸磺胺类抗性基因的丰度范围为8.3×103~5.9×107copies·g-1,长江三角洲喹诺酮类抗性基因丰度范围为2.5×105~4.2×107copies·g-1)[36]。Tan等[37]调查了北极和亚北极海洋沉积物中30种ARGs的丰度和多样性,共检测出26种ARGs,磺胺类抗性基因sul1、sul2和sul3在极地地区样本中最普遍,与中国受人类活动影响较大的参考位点相比,如海河、天津水上公园等,ARGs丰度在10-8~10-2copies(相对于标记基因16S rRNA)之间,极地海洋沉积物中ARGs的丰度水平较低,丰度范围为10-9~10-5copies(相对于标记基因16S rRNA)。对极地海洋环境的有限研究证明,与受人类影响严重的低纬度海区相比,极地海洋环境中的ARGs丰度较低。此外,河口,沿海地区以及极地海洋环境中ARGs的广泛存在进一步证明了ARGs的全球化扩散现象[36-40]。
2 近岸海洋环境中ARGs的主要来源(Main sources of ARGs in nearshore marine environment)
2.1 医疗废水
据中国细菌耐药性调查(2021)统计,从中国52家医院临床采集的49 437株大肠埃希菌中,环丙沙星和甲氧苄啶-磺胺甲恶唑的耐药株比例超过50%[41],说明我国医院废水中ARB和ARGs广泛存在。中国7家医院废水中检测出的ARGs总丰度范围为4.62×107~5.70×1011copies·mL-1[42]。Wang等[43]在我国3家医院废水中分别检测到131、160和139种ARGs,有126种ARGs在3家医院的检出率为100%,sul1和tetO等优势基因的绝对丰度高达(7.24±1.49)×107copies·mL-1和(8.34±1.23)×107copies·mL-1。西班牙某医院废水中四环素类抗性基因tetW的绝对丰度为104copies·mL-1,磺胺类类抗性基因sul1的绝对丰度为104.4copies·mL-1,喹诺酮类抗性基因qnrS的绝对丰度为103.3copies·mL-1,大环内酯类抗性基因ermB的绝对丰度为103.2copies·mL-1[44]。制药废水中也含有高浓度的ARGs残留。Guo等[45]发现中国6家制药厂废水中检测到的ARGs总绝对丰度介于(1.47±10.02)×102~(2.96±0.88)×108copies·mL-1之间。医院和制药厂一般带有污水处理系统,但其对ARGs的去除效果较差。例如,Liu等[46]比较了10种ARGs在医院污水处理系统进水和出水中的相对丰度,发现除tetA,其他ARGs的丰度没有显著变化,甚至高于进水。我国某制药废水处理厂仅能将ARGs总绝对丰度降低1个数量级,出水中ARGs总丰度仍高达101~107copies·mL-1[45]。因此,医院和制药厂自带的废水处理系统并不能有效去除ARGs。现场处理后的医疗废水或集中到污水处理厂处理后排入海洋,中间没有针对ARGs的专门处理步骤,ARGs的生物安全评估也并不在废水排放标准之列[47]。由此可得,医疗废水可能是ARGs扩散到海洋环境中的重要来源。
2.2 陆地径流
陆地地区抗生素消耗量巨大,Zhang等[48]估计中国每年消耗92 700 t抗生素,最终超过58%的抗生素排放到全国58个流域,来源于人类活动和农业畜牧的大量残留抗生素、ARB和ARGs流入河流,最终排放到河口和沿海环境。我国辽河河口sul1、sul2和sul3的浓度从河流到海洋呈明显下降的趋势,说明陆地径流输入是辽河口磺胺类抗性基因的主要来源[49]。Sala-Comorera等[50]发现爱尔兰都柏林湾海水浴场的ARGs组成与流入海湾的2条主要河流和小溪的ARGs组成一致,并且河流和海洋中的噬菌体携带的ARGs丰度水平没有显著差异,说明河流输入会对海洋中的ARGs产生持久的影响。我国莱州湾河口上游喹诺酮类抗性基因aac(6’)-Ib的浓度(3.89×102copies·mL-1)是下游(0~2.17×102copies·mL-1)的2~14倍,表明河流输入是该区域ARGs污染的主要来源之一[27]。
2.3 污水处理厂尾水
现有污水处理工艺无法完全去除ARGs,污水处理厂出水中ARGs普遍存在且丰度较高。Mao等[51]在我国某污水处理厂出水中共检出磺胺类、四环素类和大环内酯类等10种ARGs类型,其中磺胺类抗性基因sul1丰度高达8.9×105copies·mL-1。Liu等[46]在中国3个污水处理厂尾水中检测到sul1、sul2和intI1的相对丰度普遍高于进水。Lee等[52]报道了韩国2个污水处理厂出水中ARGs的日负荷分别高达4.2×1018copies和5.4×1018copies。污水处理厂废水排放到周边海域,使接收海区ARGs丰度显著上升。sul1、sul2和tetM在波罗的海附近3个污水处理厂出水中均被检出,其平均丰度高达107copies·mL-1,富含ARGs的废水直接排入海洋,对波罗的海造成严重污染[53]。对泰晤士河流域的研究表明,环境ARB负荷与污水处理厂的距离、规模和类型有很强的相关性[54]。Chen等[55]观察到中国杭州湾的某污水处理厂废水接收海区ARGs丰度逐年升高(2017—2018),所有检测到的ARGs在2018年富集度更高,磺胺类和四环素类是最丰富的ARGs,其丰度由104~107copies·g-1(2017年)增加到107~109copies·g-1(2018年)。以上结果表明污水处理厂尾水已成为周边海域ARGs的主要来源。
2.4 养殖废水
养殖废水是抗生素和ARGs的重要来源。抗生素作为饲料添加剂和生长促进剂曾在养殖业中被广泛应用[56]。研究表明,抗生素在生物体内不能被有效代谢,超过70%的抗生素能够在环境中保持有效的功能[57]。残留的抗生素可在水体和沉积物中累积,并对当地细菌群落施加抗生素选择性压力[58]。Chen等[59]在中国12个畜禽养殖场排放废水中共检测到22种ARGs,其中sul1、sul2和tetM含量最高。我国某养猪场废水共检测出8种ARGs,其中2种磺胺类抗性基因丰度为1.4×106~7.1×108copies·mL-1,6种四环素类抗性基因丰度为9.5×103~5.0×109copies·mL-1[60]。Jang等[61]调查了韩国2个沿海水产养殖区排放污水(不经任何处理,直接排放进海洋)中ARGs的分布,发现所检测的ARGs相对丰度范围为4.24×10-3~1.46×10-2copies(相对于标记基因16S rRNA),磺胺类抗性基因中tetB和tetD丰度最高,分别为7.35×10-3copies(相对于标记基因16S rRNA)和6.97×10-3copies(相对于标记基因16S rRNA)。对整个中国沿海水产养殖区的调查显示,磺胺类抗性基因,尤其是sul1和sul2,是沉积物中最常见的ARGs,其丰度分别高达6.87×10-2copies(相对于标记基因16S rRNA)和8.99×10-2copies(相对于标记基因16S rRNA)。磺胺类药物是水产养殖中最常用的抗生素之一,说明水产养殖抗生素的广泛使用对沿海ARGs的组成带来严重影响[62]。Jo等[63]利用宏基因组分析技术研究了韩国沿海养鱼场养殖尾水中的ARGs,结果表明养殖尾水中的ARGs丰度是非养殖区海水的10倍,而且养殖尾水不经任何处理直接排放入海洋,这表明养殖尾水已成为周边海域ARGs的主要来源。
2.5 近岸海域ARGs来源分析与定量
海洋环境中的ARGs有多种来源(图1),但不同来源的量化评估以及ARGs的溯源分析仍然是一个挑战,准确追踪ARGs污染来源并分析其通量对于控制ARGs在环境中的传播至关重要。Zheng等[64]收集了40条关于近岸海洋环境ARGs污染来源的文献记录,发现有33.75%~55.00%的研究将河流径流确定为ARGs的来源,15.00%~47.50%的研究将污水处理厂废水确定为ARGs的来源,20.00%~48.00%的研究将畜禽养殖、医疗废水归为海洋环境ARGs的重要来源,15.00%~40.00%的研究将水产养殖废水归为ARGs的重要来源。但明确各来源对某一环境地点ARGs污染的具体贡献比仍面临许多问题。ARGs的环境本底值、污染物对ARGs的选择作用、ARGs的时空变化性等多重因素限制了对ARGs实现准确源追踪[65]。近年来,基于宏基因组测序和机器学习分类相结合的方法为海洋环境中ARGs来源量化提供了一个可行的方案,宏基因组测序可以获得环境中完整的ARGs数据,机器学习分类技术可以实现对数据的全面分析[65]。Li等[66]结合宏基因组测序和微生物来源分析工具SourceTracker,量化了不同污染源对珠江口和东海沉积物中ARGs污染的贡献水平,发现污水处理厂污泥对珠江口沉积物中ARGs的贡献比达44%~60%,而对大部分东海沉积物样品中ARGs的贡献比<8%,总体而言,粪便污染源对珠江口沉积物中ARGs的贡献比至少是南海沉积物中的5倍,反映了2个地区不同程度的人为干扰。Chen等[67]结合宏基因组测序与快速期望最大化微生物源跟踪(fast expectation-maximization microbial source tracking, FEAST)以及线性判别效应方法(linear discriminant analysis effect size method, LEfSe),结合最新发现的crAssphage噬菌体(人类粪便和污水中最丰富的病毒,用来追踪人类粪便ARGs污染对环境ARGs污染影响),发现白洋淀超过80%的ARGs污染与府河输入有关。无论采用哪种方法,获得来源环境和接收环境的完整ARGs信息(宏基因组测序完成)是构建ARGs源追踪量化框架的基础。宏基因组测序与不断优化的机器学习分类方法相结合将进一步提高ARGs源解析的准确性。
3 近岸海洋环境中ARGs的传播扩散(Propagation and diffusion of ARGs in nearshore marine environment)
除了细菌的内在耐药性外,细菌还可以获得或发展对抗生素的耐药性。细菌获得ARGs的途径主要有2种。第1种是在环境的选择性压力下(抗生素、重金属等污染物),为了继续生存,基因发生自发突变产生ARGs;第2种是ARGs在细菌之间转移,ARGs转移分为垂直转移和水平转移,前者是指亲代和子代遗传导致的ARGs传播,后者是ARGs通过可移动遗传元件(mobile genetic elements, MGEs)进行水平基因转移(horizontal gene transfer, HGT),并且能够在不同种和属的菌株间水平传播[68-69]。仅靠环境选择性压力导致的自发基因突变并不会造成ARGs如此广泛的传播,HGT是ARGs传播扩散的主要途径[70]。ARGs可以通过转化、转导和接合等多种机制进行水平转移。近年来的研究发现,整合子和质粒等多种MGEs以及噬菌体对海洋环境水体和沉积物中ARGs的水平传播发挥重要作用。
接合是指质粒和整合子等携带ARGs的MGEs通过菌毛接触从一种细菌转移到另一种细菌[71]。近年来,在近岸海域发现整合子尤其是一类整合子(intI1)的广泛存在。大量研究表明近岸海域ARGs的发生与分布与intI1显著相关。例如,Zhu等[72]对中国沿海18个河口沉积物中的MGEs进行了分析,发现所检测的9种转座子基因和1种整合子基因的总绝对丰度范围为5.66×104~4.23×107copies·g-1,并且MGEs与ARGs的总绝对丰度之间存在显著相关性(P<0.01)。Chen等[55]发现杭州湾沿海沉积物中磺胺类抗性基因(sul1和sul2)和二氨基嘧啶类抗性基因(dfrA1和dfrA13)的绝对丰度与intI1的绝对丰度显著相关(P<0.01)。Zhang等[73]在澳大利亚的河口和海岸沉积物样本中检测到8种MGEs,MGEs的相对丰度与总ARGs丰度呈显著正相关(P<0.01)。在大辽河河口、渤海和南极等环境中均发现磺胺抗性基因的相对丰度与intI1之间存在显著相关性(P<0.01)[21, 36, 49]。整合子基因序列可促进ARGs转移到细菌染色体中,从而促进细菌耐药性的传播[36]。Jun等[74]在韩国海水养殖场分离的爱德华氏菌(Edwardsiella tarda)中检测出4种四环素类抗性基因tetA、tetB、tetD和tetG,其中tetA和tetD广泛存在于可移动质粒上,推测海洋细菌之间可能通过可移动质粒进行水平基因转移从而导致韩国水产养殖环境中四环素类ARGs的广泛存在。Jo等[63]对沿海养殖废水中的ARGs遗传背景进行分析,发现携带ARGs的质粒比例显著高于染色体(P<0.05),而且磺胺类抗性基因仅在质粒上发现。综上可知,整合子和质粒等MGEs可能通过接合转移显著促进了近岸海域ARGs的传播。
转导是指通过中间体-噬菌体病毒,在细菌之间转移染色体和染色体外DNA[75]。Calero-Cáceres和Balcázar[76]在北太平洋环流、印度洋、马尾藻海、北冰洋、哥伦比亚湾和墨西哥湾的海洋噬菌体样本中均发现了与氨基香豆素、杆菌肽和多药耐药相关的ARGs,这一研究结果表明来自海洋的噬菌体是ARGs的潜在储存库。研究报道在都柏林海滩洗浴水域分离的噬菌体中检测到blaTEM、blaSHV、qnrS和sul1等4种ARGs,其中blaTEM丰度高达10~102copies·mL-1,通过比较发现淡水和海水中的噬菌体ARGs丰度水平没有显著差异。推测噬菌体介导的细菌间ARGs的水平转移可能普遍存在于海洋环境中[50]。
自然转化是指来自细胞外环境的裸露DNA(ARGs)被细菌主动吸收并整合到它们的基因组中。转化过程主要依赖于细胞外DNA,在海水环境中DNA浓度极易被稀释,因此,自然转化在海洋环境条件下可能很难发生。目前转化过程在海洋环境中研究较少[77]。Nonaka等[78]报告了一种新型MGE,它可以把从海洋弧菌分离株中获得的携带ARGs的质粒整合到大肠杆菌染色体中。综上,海洋环境中噬菌体和整合子等的广泛存在说明水平基因转移可能是海洋环境中ARGs传播扩散的主要途径。
近岸海域作为连接内陆环境和海洋环境的纽带区域,环境较为复杂,受人类活动影响较为严重,存在抗生素、重金属等多种污染物,容易引起ARB和ARGs的富集。此外,受水文动力学的影响沿岸海水能够与外海不断进行海水交换,容易引发ARGs的全球化扩散,因此,应重点监测近岸海域ARGs的赋存状态和传播分布。此外,有研究指出,当温度升高时,HGT的频率会加快[79-80]。目前,全球气候变化导致的海面温度上升为细菌提供了理想的生存条件,这种现象可能有利于促进ARGs在海洋环境微生物种群中的基因重组,从而促进ARGs在海洋细菌中的传播[81-82]。
4 近岸海洋环境中ARGs传播扩散的影响因素(Factors influencing propagation and diffusion of ARGs in nearshore marine environment)
自然环境中ARGs的大规模传播扩散是一个多维度且受多重因素影响的复杂过程。近年来的研究指出,环境中的ARGs会在抗生素和其他环境因子的选择压力下被选择和富集[58]。探究影响海洋环境中ARGs传播扩散的影响因素,有助于我们全面了解海洋环境中抗生素耐药性变化趋势,对于评估环境中ARGs的抗药性风险和控制阻断ARGs的传播具有重要意义。
4.1 抗生素
抗生素在促进ARGs的富集和传播中发挥了重要作用[83]。一些抗生素在亚致死剂量下能明显促进质粒介导的ARGs水平转移,比如常见的氟诺喹酮类抗生素可通过接合转移来促进ARGs在不同的细菌之间进行传播[84-85]。然而,抗生素的使用和ARGs的产生和传播之间的关联机制仍未明确。我国北黄海表层水中sul1、sul2的相对丰度与磺胺类抗生素总浓度显著相关(P=0.001),表明抗生素对ARGs施加了选择性压力[86]。但也有研究表明,抗生素与对应ARGs之间无直接相关性。Lu等[25]发现我国渤海表层海水中ARGs丰度与抗生素之间没有相关性。Wang等[87]发现沿海水产养殖系统中ARGs丰度与抗生素残留无关。推测可能是受海水稀释和交换的影响,海洋环境中抗生素的浓度较低且不稳定,抗生素耐药性与抗生素之间的相关性减弱[15]。
4.2 重金属
研究发现,金属纳米粒子和离子可以通过破坏细菌的细胞膜,加速细菌对ARGs的吸收[88]。实验室分析表明,重金属和抗生素表现出很强的协同效应,能够同时富集ARGs和重金属抗性基因[89-90]。我国长江口水体中sul1、sul2、sul3和aac(6’)-Ib的丰度和溶解铜浓度之间呈正相关(P<0.05),沉积物中tetW丰度与多种金属(铁、锰、铜、锌和镍)之间存在显著相关性(P<0.05),sul1、sul2和tetA丰度与铅和铬浓度显著相关(P<0.05)[91]。Lu等[49]发现在大辽河河口中,3种磺胺类抗性基因sul1、sul2和sul3的丰度与6种重金属铬、钴、镍、铜、锌和铅浓度呈正相关(P<0.05),其相关系数(r值范围为0.335~0.973)明显高于与抗生素的相关系数。抗生素易降解且易被海水稀释,而金属不会被降解,可以对细菌施加长期的选择压力,因此在海洋环境中,当抗生素选择压力较弱时,重金属可能在ARGs的维持和增殖中发挥重要作用[15]。
4.3 微塑料
水中高浓度的微塑料(microplastics, MPs)已经变成了ARB和ARGs的有效载体[92]。研究表明,MPs可以从周围的水中富集ARGs和微生物[93-94]。Yang等[94]报告称,在北太平洋环流海水中收集的MPs中总ARGs的平均相对丰度为5.15×10-3copies(相对于标记基因16S rRNA),比海水中高5.69倍,并且MPs颗粒中的微生物多样性也高于海水。中国福建省的水产养殖系统水样中MPs含量为58~72 个·m-3,MPs中的ARGs含量比沿海水域高出4个数量级[23]。ARGs在海洋环境MPs中富集的主要原因是MPs可以为微生物提供吸附力和漂浮载体,携带ARGs的MPs可以随洋流扩散和积累。此外,MPs的疏水表面容易形成一层生物膜,生物膜为携带ARGs的微生物提供了相对稳定的生存环境,成为ARB基因交换的热点区域[64]。
4.4 其他污染物和环境理化因子
研究发现草甘膦除草剂能够影响ARGs和MGEs的丰度,促进ARGs的HGT过程[95]。当含有ARGs的DNA与矿物质和腐殖质结合时,避免了核酸酶的降解,会导致携带ARGs的细菌迅速传播和繁殖[96]。Sui等[97]发现有机碳含量高的水环境相对更适合微生物生长,ARGs含量也较高。水体中的各种营养物质,如硝酸盐、磷酸盐和硅酸盐等,能够促进携带ARGs的细菌的生长,并促进沉积物对ARGs的吸附[98]。此外,温度和pH通过对细菌群落进化施加压力,进而影响水环境中ARGs结构和丰度[99-100]。Zhang等[73]发现盐度是影响澳大利亚海滩土壤ARGs分布的主要因素(P<0.05)。
4.5 微生物群落
Guo等[101]指出微生物群落结构是导致ARGs分布变化的重要因素。中国南海近岸沉积物中的ARGs与微生物群落显著相关(P<0.05),微生物群落可以解释11.5%的ARGs分布变化[98]。Jiang等[102]在中国海南岛红树林覆盖海域沉积物中发现133个细菌属与部分ARGs(如blaCMY、blaCMY2-01和lnuB-01)具有较高的相关性(P<0.01)。Zhu等[72]发现微生物群落解释了中国整个沿海河口2.61%的ARGs分布变化。Lu等[25]发现渤海和黄海海水中tetX、tetG和sul1等抗性基因与气微菌属(Aeromicrobium)、芽单胞菌属(Blastomonas)和苯基杆菌属(Phenylobacterium)等多种海洋优势菌属具有强烈相关性(P<0.01),进一步的共现分析揭示海洋细菌可能是ARGs的潜在宿主。
5 近岸海洋环境中ARGs的健康风险与评估(Health risks and assessment of ARGs in nearshore marine environment)
抗生素滥用乱用导致水产养殖环境中存在大量抗生素残留,对当地细菌群落构成选择性压力,加速了ARGs的产生、传播和转移,并可能导致ARGs在致病菌中出现[103]。许多细菌,特别是人类共生细菌和病原体,已被报道具有多重耐药性[104]。例如,Jo等[63]在韩国济州岛沿海养鱼场废水样本中分离出多种机会性病原体,如弧菌(Vibrio)、发光杆菌(Photobacterium)和黄杆菌属(Tenacibaculum)等,其中弧菌属是主要的耐药菌属,携带多种β-内酰胺类和四环素类抗性基因。ARGs随菌群传播,可以进入养殖生物体内,Kumaran等[105]从印度48份海产品中分离到80株大肠杆菌(条件致病菌),对氨苄西林耐药的菌株最高为56.25%。Hong等[32]从中国沿海三省的主要海产品(对虾、贝类、海参和半滑舌鳎等)中分离到90株副溶血弧菌(Vibrio parahaemolyticus),超过80%的菌株对氨苄西林和头孢唑林耐药,44.4%的菌株对至少3种抗生素具有多重耐药性,副溶血弧菌作为一种食源性致病菌,其多重抗药性对人类健康构成潜在的威胁。抗生素耐药性已成为对水产食品的最大威胁之一,并能通过食物链对人体健康造成威胁[106]。
人类可通过游泳或摄入海鲜等方式直接或间接地摄入ARGs(图1)。在智利病人的大肠杆菌中发现的ARGs序列qnrA1、qnrB1和qnrS1和智利海洋细菌中的相同,表明在海洋细菌和人类病原体之间可能发生了ARGs转移[107]。龙文芳等[108]对中国海水浴场中分离的革兰阴性菌进行抗药性检测,发现耐药比例高达45%。Leonard等[109]发现经常冲浪的人粪便中携带具有超广谱β-内酰胺酶基因大肠杆菌的可能性大约是不游泳人的3倍,而携带含有blaCTX-M的大肠杆菌的可能性则是不游泳人的4倍以上,进一步对人体中耐药性大肠杆菌进行量化研究,发现公开水域游泳选手耐药性大肠杆菌的携带率高于普通人群,但对于后续感染的风险还未知。
目前针对海洋环境中ARGs对人类健康造成的危害和风险尚缺乏定量评估模型和直接证据。此外,ARGs导致的抗药性风险与污染物的暴露水平、携带ARGs的宿主类型、暴露途径和ARGs的横向传播等多种因素有关,使得量化ARGs的健康风险变得困难[110]。如何确定各因素所占比例是构建ARGs健康风险定量评估模型的重点和难点。Hendriksen等[111]基于60个国家的污水宏基因组数据,精确地预测了全球ARGs的多样性和丰度,但丰度并不直接代表健康风险。Zhang等[112]基于宏基因组测序技术,通过计算ARGs的人类可及性(ARGs从环境传递到人类体内细菌的能力)、流动性和人类致病性(ARGs从环境细菌转移到致病菌的可能性)和临床相关性(ARGs对应的临床上使用的总的抗生素种类)等4个指标,构建了一个ARGs健康风险定量评估框架,将ARGs健康风险定义为ARGs会干扰对病原体的临床治疗的风险,并结合机器学习建立了海洋环境ARGs风险评估方法,发现太平洋和大西洋地区的抗生素耐药性威胁较高。南极附近海域的抗生素耐药性风险高于北极附近海域。但样本数量较少且分布不均以及宏基因组数据质量参差不一限制了对海洋环境进行更为全面和准确的ARGs风险评估。
6 展望(Prospect)
ARGs作为一种基因污染物,可以在生物之间进行复制和传播,同时具有传统化学污染物的特性,可以从陆源扩散到近岸海洋环境中,进而引发ARGs的全球化迁移和扩散,给海洋环境和人类健康造成严重威胁,作为ARGs的潜在储存库和风险来源,对海洋环境ARGs污染进行系统性、深入性的研究具有重要意义,未来研究应重点关注以下几点。
(1)构建海洋环境中ARGs污染的标准化研究方法。目前关于海洋环境中ARGs丰度和多样性的检测方法主要包括高通量qPCR和宏基因组测序。高通量qPCR检测灵敏度高、检测通量高,但不能检测未知ARGs,宏基因组测序可以检测未知ARGs、具有更高的检测通量,还可以对宏基因组数据集实现回溯分析和挖掘,但价格昂贵。此外,ARGs数据库是宏基因组检测ARGs的核心,数据库完整度决定了评估样本中ARGs丰度和多样性的准确性。未来应不断扩充数据库,并实时更新,提高数据库完整度。高通量qPCR和宏基因组测序相互补充,可以作为海洋环境样本ARGs污染检测的标准化方法。此外,还应建立系统化的采样方案,这是建立完善的海洋环境ARGs数据库的前提,有利于对不同海域进行ARGs污染水平比较。
(2)目前对海洋环境中ARGs的组成和分布规律以及影响因素的研究不够完善。许多新型ARGs和抗性机制在海洋环境中不断发现,不同的海洋环境中ARGs分布规律和影响因素复杂,目前的研究不足以解释海洋环境中ARGs的迁移和污染规律,未来需要建立一个全球海洋环境ARGs数据共享平台,对ARGs的相关变量(类型、丰度、机制和影响因素等)进行详细记录和统计,对于全面认识海洋ARGs污染水平、遏制耐药性发展和保护海洋生态环境具有重要意义。
(3)对海洋环境中ARGs和ARB的传播转移规律仍缺乏系统性研究。ARGs在海洋细菌与人类和动物致病菌间的水平转移机制以及ARGs和ARB在环境-动物-人之间的动态传播机制应成为未来关注的重点,对于阻断ARGs传播并降低ARGs对人类健康的威胁具有重要意义。
(4)定量评估海洋环境ARGs健康风险是未来研究的重难点。ARGs的风险主要包括在环境中传播转移的风险和导致临床上抗生素治疗失效的风险。评估重点为ARGs暴露水平、携带ARGs的宿主种类、暴露途径和环境中可构成ARGs选择压力的污染物因素影响等,此外,如何确定各因素所占比例有待深入研究,以期量化环境中的ARGs对人类健康的影响。
(5)关注ARGs引发的生态风险。ARGs在环境中的传播长久而持续,目前对ARGs的生态风险研究仅局限于特定时间段特定环境中的ARGs检测,需要在更长时间跨度和更大空间范围上对环境中的ARGs污染状况进行监测,以阐明其对生态环境的危害。
[1] Wu D, Jin L, Xie J W, et al.Inhalable antibiotic resistomes emitted from hospitals: Metagenomic insights into bacterial hosts, clinical relevance, and environmental risks[J].Microbiome, 2022, 10(1): 19
[2] World Bank Group.Drug resistant infections: A threat to our economic future: Final report[R].Washington DC: World Bank Group, 2017
[3] Shuai M L, Zhang G Q, Zeng F F, et al.Human gut antibiotic resistome and progression of diabetes[J].Advanced Science, 2022, 9(11): e2104965
[4] Cassini A, Högberg L D, Plachouras D, et al.Attributable deaths and disability-adjusted life-years caused by infections with antibiotic-resistant bacteria in the EU and the European Economic Area in 2015: A population-level modelling analysis[J].The Lancet Infectious Diseases, 2019, 19(1): 56-66
[5] World Health Organization.Global action plan on antimicrobial resistance 2015[R].Geneva: World Health Organization, 2015
[6] Tacconelli E, Sifakis F, Harbarth S, et al.Surveillance for control of antimicrobial resistance[J].The Lancet Infectious Diseases, 2018, 18(3): e99-e106
[7] Wernli D, Jørgensen P S, Morel C M, et al.Mapping global policy discourse on antimicrobial resistance[J].BMJ Global Health, 2017, 2(2): e000378
[8] Pruden A, Pei R T, Storteboom H, et al.Antibiotic resistance genes as emerging contaminants: Studies in northern Colorado[J].Environmental Science &Technology, 2006, 40(23): 7445-7450
[9] Zhao W X, Wang B, Yu G.Antibiotic resistance genes in China: Occurrence, risk, and correlation among different parameters[J].Environmental Science and Pollution Research, 2018, 25(22): 21467-21482
[10] Qiao M, Ying G G, Singer A C, et al.Review of antibiotic resistance in China and its environment[J].Environment International, 2018, 110: 160-172
[11] Zhu Y G, Zhao Y, Zhu D, et al.Soil biota, antimicrobial resistance and planetary health[J].Environment International, 2019, 131: 105059
[12] Ju F, Li B, Ma L P, et al.Antibiotic resistance genes and human bacterial pathogens: Co-occurrence, removal, and enrichment in municipal sewage sludge digesters[J].Water Research, 2016, 91: 1-10
[13] Zeng S Z, Hou D W, Liu J, et al.Antibiotic supplement in feed can perturb the intestinal microbial composition and function in Pacific white shrimp[J].Applied Microbiology and Biotechnology, 2019, 103(7): 3111-3122
[14] Ding C S, Jin M, Ma J, et al.Nano-Al2O3can mediate transduction-like transformation of antibiotic resistance genes in water[J].Journal of Hazardous Materials, 2021, 405: 124224
[15] Gao H, Zhang L X, Lu Z H, et al.Complex migration of antibiotic resistance in natural aquatic environments[J].Environmental Pollution, 2018, 232: 1-9
[16] Miranda C D, Godoy F A, Lee M R.Current status of the use of antibiotics and the antimicrobial resistance in the Chilean salmon farms[J].Frontiers in Microbiology, 2018, 9: 1284
[17] Gao P P, Mao D Q, Luo Y, et al.Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment[J].Water Research, 2012, 46(7): 2355-2364
[18] Yuan Q B, Zhai Y F, Mao B Y, et al.Fates of antibiotic resistance genes in a distributed swine wastewater treatment plant[J].Water Environment Research, 2019, 91(12): 1565-1575
[19] Guo X P, Yang Y, Lu D P, et al.Biofilms as a sink for antibiotic resistance genes(ARGs)in the Yangtze Estuary[J].Water Research, 2018, 129: 277-286
[20] Chen B W, Liang X M, Nie X P, et al.The role of class I integrons in the dissemination of sulfonamide resistance genes in the Pearl River and Pearl River Estuary, South China[J].Journal of Hazardous Materials, 2015, 282: 61-67
[21] Niu Z G, Zhang K, Zhang Y.Occurrence and distribution of antibiotic resistance genes in the coastal area of the Bohai Bay, China[J].Marine Pollution Bulletin, 2016, 107(1): 245-250
[22] Suzuki S, Ogo M, Miller T W, et al.Who possesses drug resistance genes in the aquatic environment?: Sulfamethoxazole(SMX)resistance genes among the bacterial community in water environment of Metro-Manila, Philippines[J].Frontiers in Microbiology, 2013, 4: 102
[23] Lu J, Zhang Y X, Wu J, et al.Effects of microplastics on distribution of antibiotic resistance genes in recirculating aquaculture system[J].Ecotoxicology and Environmental Safety, 2019, 184: 109631
[24] Germond A, Kim S J.Genetic diversity of oxytetracycline-resistant bacteria and tet(M)genes in two major coastal areas of South Korea[J].Journal of Global Antimicrobial Resistance, 2015, 3(3): 166-173
[25] Lu J, Zhang Y X, Wu J, et al.Occurrence and spatial distribution of antibiotic resistance genes in the Bohai Sea and Yellow Sea areas, China[J].Environmental Pollution, 2019, 252: 450-460
[26] Chen J Y, Su Z G, Dai T J, et al.Occurrence and distribution of antibiotic resistance genes in the sediments of the East China Sea Bays[J].Journal of Environmental Sciences(China), 2019, 81: 156-167
[27] Li Q W, Na G S, Zhang L X, et al.Effects of corresponding and non-corresponding contaminants on the fate of sulfonamide and quinolone resistance genes in the Laizhou Bay, China[J].Marine Pollution Bulletin, 2018, 128: 475-482
[28] Ullah R, Yasir M, Bibi F, et al.Taxonomic diversity of antimicrobial-resistant bacteria and genes in the Red Sea coast[J].Science of the Total Environment, 2019, 677: 474-483
[29] Ng C, Chen H J, Goh S G, et al.Microbial water quality and the detection of multidrug resistant E. coliand antibiotic resistance genes in aquaculture sites of Singapore[J].Marine Pollution Bulletin, 2018, 135: 475-480
[30] Guo X P, Sun X L, Chen Y R, et al.Antibiotic resistance genes in biofilms on plastic wastes in an estuarine environment[J].The Science of the Total Environment, 2020, 745: 140916
[31] Dong H, Chen Y L, Wang J, et al.Interactions of microplastics and antibiotic resistance genes and their effects on the aquaculture environments[J].Journal of Hazardous Materials, 2021, 403: 123961
[32] Hong B, Ba Y B, Niu L, et al.A comprehensive research on antibiotic resistance genes in microbiota of aquatic animals[J].Frontiers in Microbiology, 2018, 9: 1617
[33] Muziasari W I, Pitkänen L K, Sørum H, et al.The resistome of farmed fish feces contributes to the enrichment of antibiotic resistance genes in sediments below Baltic Sea fish farms[J].Frontiers in Microbiology, 2016, 7: 2137
[34] Giatsis C, Sipkema D, Smidt H, et al.The impact of rearing environment on the development of gut microbiota in tilapia larvae[J].Scientific Reports, 2015, 5: 18206
[35] Wang Z Y, Chen Q W, Zhang J Y, et al.Critical roles of cyanobacteria as reservoir and source for antibiotic resistance genes[J].Environment International, 2020, 144: 106034
[36] Na G S, Wang C X, Gao H, et al.The occurrence of sulfonamide and quinolone resistance genes at the Fildes Peninsula in Antarctica[J].Marine Pollution Bulletin, 2019, 149: 110503
[37] Tan L, Li L Y, Ashbolt N, et al.Arctic antibiotic resistance gene contamination, a result of anthropogenic activities and natural origin[J].The Science of the Total Environment, 2018, 621: 1176-1184
[38] Barkovskii A L, Green C, Hurley D.The occurrence, spatial and temporal distribution, and environmental routes of tetracycline resistance and integrase genes in Crassostrea virginicabeds[J].Marine Pollution Bulletin, 2010, 60(12): 2215-2224
[39] Li A L, Chen L J, Zhang Y, et al.Occurrence and distribution of antibiotic resistance genes in the sediments of drinking water sources, urban rivers, and coastal areas in Zhuhai, China[J].Environmental Science and Pollution Research International, 2018, 25(26): 26209-26217
[40] Suzuki S, Ogo M, Takada H, et al.Contamination of antibiotics and suland tet(M)genes in veterinary wastewater, river, and coastal sea in Thailand[J].The Science of the Total Environment, 2021, 791: 148423
[41] 胡付品, 郭燕, 朱德妹, 等.2020年CHINET中国细菌耐药监测[J].中国感染与化疗杂志, 2021, 21(4): 377-387
Hu F P, Guo Y, Zhu D M, et al.CHINET surveillance of bacterial resistance: Results of 2020[J].Chinese Journal of Infection and Chemotherapy, 2021, 21(4): 377-387(in Chinese)
[42] Li J N, Cheng W X, Xu L K, et al.Antibiotic-resistant genes and antibiotic-resistant bacteria in the effluent of urban residential areas, hospitals, and a municipal wastewater treatment plant system[J].Environmental Science and Pollution Research International, 2015, 22(6): 4587-4596
[43] Wang Q, Wang P L, Yang Q X.Occurrence and diversity of antibiotic resistance in untreated hospital wastewater[J].The Science of the Total Environment, 2018, 621: 990-999
[44] Lucas D, Badia-Fabregat M, Vicent T, et al.Fungal treatment for the removal of antibiotics and antibiotic resistance genes in veterinary hospital wastewater[J].Chemosphere, 2016, 152: 301-308
[45] Guo X Y, Yan Z, Zhang Y, et al.Behavior of antibiotic resistance genes under extremely high-level antibiotic selection pressures in pharmaceutical wastewater treatment plants[J].The Science of the Total Environment, 2018, 612: 119-128
[46] Liu X H, Zhang G D, Liu Y, et al.Occurrence and fate of antibiotics and antibiotic resistance genes in typical urban water of Beijing, China[J].Environmental Pollution, 2019, 246: 163-173
[47] Zhang S C, Huang J Q, Zhao Z C, et al.Hospital wastewater as a reservoir for antibiotic resistance genes: A meta-analysis[J].Frontiers in Public Health, 2020, 8: 574968
[48] Zhang Q Q, Ying G G, Pan C G, et al.Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resistance[J].Environmental Science &Technology, 2015, 49(11): 6772-6782
[49] Lu Z H, Na G S, Gao H, et al.Fate of sulfonamide resistance genes in estuary environment and effect of anthropogenic activities[J].The Science of the Total Environment, 2015, 527-528: 429-438
[50] Sala-Comorera L, Nolan T M, Reynolds L J, et al.Bacterial and bacteriophage antibiotic resistance in marine bathing waters in relation to rivers and urban streams[J].Frontiers in Microbiology, 2021, 12: 718234
[51] Mao D Q, Yu S, Rysz M, et al.Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J].Water Research, 2015, 85: 458-466
[52] Lee J, Jeon J H, Shin J, et al.Quantitative and qualitative changes in antibiotic resistance genes after passing through treatment processes in municipal wastewater treatment plants[J].The Science of the Total Environment, 2017, 605-606: 906-914
[53] Kotlarska E,  uczkiewicz A, Pisowacka M, et al.Antibiotic resistance and prevalence of class 1 and 2 integrons in Escherichia coliisolated from two wastewater treatment plants, and their receiving waters(Gulf of Gdansk, Baltic Sea, Poland)[J].Environmental Science and Pollution Research International, 2015, 22(3): 2018-2030
uczkiewicz A, Pisowacka M, et al.Antibiotic resistance and prevalence of class 1 and 2 integrons in Escherichia coliisolated from two wastewater treatment plants, and their receiving waters(Gulf of Gdansk, Baltic Sea, Poland)[J].Environmental Science and Pollution Research International, 2015, 22(3): 2018-2030
[54] Amos G C, Gozzard E, Carter C E, et al.Validated predictive modelling of the environmental resistome[J].The ISME Journal, 2015, 9(6): 1467-1476
[55] Chen J Y, Zhang Z Y, Lei Z, et al.Occurrence and distribution of antibiotic resistance genes in the coastal sediments of effluent-receiving areas of WWTPs, China[J].Bioresource Technology Reports, 2020, 11: 100511
[56] Xu J, Xu Y, Wang H M, et al.Occurrence of antibiotics and antibiotic resistance genes in a sewage treatment plant and its effluent-receiving river[J].Chemosphere, 2015, 119: 1379-1385
[57] Romero V L, Pons P, Bocco J L, et al.Eudragit E100®potentiates the bactericidal action of ofloxacin against fluoroquinolone-resistant Pseudomonas aeruginosa[J].FEMS Microbiology Letters, 2012, 334(2): 102-110
[58] Chen H, Liu S, Xu X R, et al.Tissue distribution, bioaccumulation characteristics and health risk of antibiotics in cultured fish from a typical aquaculture area[J].Journal of Hazardous Materials, 2018, 343: 140-148
[59] Chen B, Hao L J, Guo X Y, et al.Prevalence of antibiotic resistance genes of wastewater and surface water in livestock farms of Jiangsu Province, China[J].Environmental Science and Pollution Research, 2015, 22(18): 13950-13959
[60] 刘锐.规模化猪场废水典型抗生素抗性基因的调查研究[J].家畜生态学报, 2017, 38(11): 68-71
Liu R.A study on typical antibiotic resistance genes in largescale pig farm wastewater[J].Journal of Domestic Animal Ecology, 2017, 38(11): 68-71(in Chinese)
[61] Jang H M, Kim Y B, Choi S, et al.Prevalence of antibiotic resistance genes from effluent of coastal aquaculture, South Korea[J].Environmental Pollution, 2018, 233: 1049-1057
[62] Gao Q X, Li Y L, Qi Z H, et al.Diverse and abundant antibiotic resistance genes from mariculture sites of China’s coastline[J].The Science of the Total Environment, 2018, 630: 117-125
[63] Jo H, Raza S, Farooq A, et al.Fish farm effluents as a source of antibiotic resistance gene dissemination on Jeju Island, South Korea[J].Environmental Pollution, 2021, 276: 116764
[64] Zheng D S, Yin G Y, Liu M, et al.A systematic review of antibiotics and antibiotic resistance genes in estuarine and coastal environments[J].The Science of the Total Environment, 2021, 777: 146009
[65] Li L G, Huang Q, Yin X L, et al.Source tracking of antibiotic resistance genes in the environment—Challenges, progress, and prospects[J].Water Research, 2020, 185: 116127
[66] Li L G, Yin X L, Zhang T.Tracking antibiotic resistance gene pollution from different sources using machine-learning classification[J].Microbiome, 2018, 6(1): 93
[67] Chen H Y, Li Y Z, Sun W C, et al.Characterization and source identification of antibiotic resistance genes in the sediments of an interconnected river-lake system[J].Environment International, 2020, 137: 105538
[68] Aminov R I, Mackie R I.Evolution and ecology of antibiotic resistance genes[J].FEMS Microbiology Letters, 2007, 271(2): 147-161
[69] Partridge S R, Kwong S M, Firth N, et al.Mobile genetic elements associated with antimicrobial resistance[J].Clinical Microbiology Reviews, 2018, 31(4): e00088-e00017
[70] Lauren Brito I.Examining horizontal gene transfer in microbial communities[J].Nature Reviews Microbiology, 2021, 19(7): 442-453
[71] Ilangovan A, Connery S, Waksman G.Structural biology of the Gram-negative bacterial conjugation systems[J].Trends in Microbiology, 2015, 23(5): 301-310
[72] Zhu Y G, Zhao Y, Li B, et al.Continental-scale pollution of estuaries with antibiotic resistance genes[J].Nature Microbiology, 2017, 2: 16270
[73] Zhang Y J, Hu H W, Yan H, et al.Salinity as a predominant factor modulating the distribution patterns of antibiotic resistance genes in ocean and river beach soils[J].The Science of the Total Environment, 2019, 668: 193-203
[74] Jun L J, Jeong J B, Huh M D, et al.Detection of tetracycline-resistance determinants by multiplex polymerase chain reaction in Edwardsiella tardaisolated from fish farms in Korea[J].Aquaculture, 2004, 240(1-4): 89-100
[75] Wachino J I, Jin W C, Kimura K, et al.Intercellular transfer of chromosomal antimicrobial resistance genes between Acinetobacter baumanniistrains mediated by prophages[J].Antimicrobial Agents and Chemotherapy, 2019, 63(8): e00334-e00319
[76] Calero-Cáceres W, Balcázar J L.Antibiotic resistance genes in bacteriophages from diverse marine habitats[J].Science of the Total Environment, 2019, 654: 452-455
[77] Thomas C M, Nielsen K M.Mechanisms of, and barriers to, horizontal gene transfer between bacteria[J].Nature Reviews Microbiology, 2005, 3(9): 711-721
[78] Nonaka L, Yamamoto T, Maruyama F, et al.Interplay of a non-conjugative integrative element and a conjugative plasmid in the spread of antibiotic resistance via suicidal plasmid transfer from an aquaculture Vibrioisolate[J].PLoS One, 2018, 13(6): e0198613
[79] Walsh T R, Weeks J, Livermore D M, et al.Dissemination of NDM-1 positive bacteria in the New Delhi environment and its implications for human health: An environmental point prevalence study[J].The Lancet Infectious Diseases, 2011, 11(5): 355-362
[80] Rodríguez-Verdugo A, Lozano-Huntelman N, Cruz-Loya M, et al.Compounding effects of climate warming and antibiotic resistance[J].iScience, 2020, 23(4): 101024
[81] Joint I, Smale D A.Marine heatwaves and optimal temperatures for microbial assemblage activity[J].FEMS Microbiology Ecology, 2017, 93(2): fiw243
[82] Gabashvili E, Kobakhidze S, Chkhikvishvili T, et al.Metagenomic and recombination analyses of antimicrobial resistance genes from recreational waters of black sea coastal areas and other marine environments unveil extensive evidence for their both intrageneric and intergeneric transmission across genetically very diverse microbial communities[J].Marine Genomics, 2022, 61: 100916
[83] Zhang Y P, Niu Z G, Zhang Y, et al.Occurrence of intracellular and extracellular antibiotic resistance genes in coastal areas of Bohai Bay(China)and the factors affecting them[J].Environmental Pollution, 2018, 236: 126-136
[84] Andersson D I, Hughes D.Microbiological effects of sublethal levels of antibiotics[J].Nature Reviews Microbiology, 2014, 12(7): 465-478
[85] Shun-Mei E, Zeng J M, Yuan H, et al.Sub-inhibitory concentrations of fluoroquinolones increase conjugation frequency[J].Microbial Pathogenesis, 2018, 114: 57-62
[86] Na G S, Zhang W R, Zhou S Y, et al.Sulfonamide antibiotics in the Northern Yellow Sea are related to resistant bacteria: Implications for antibiotic resistance genes[J].Marine Pollution Bulletin, 2014, 84(1-2): 70-75
[87] Wang J H, Lu J, Wu J, et al.Proliferation of antibiotic resistance genes in coastal recirculating mariculture system[J].Environmental Pollution, 2019, 248: 462-470
[88] Zhang S, Lu J, Wang Y, et al.Insights of metallic nanoparticles and ions in accelerating the bacterial uptake of antibiotic resistance genes[J].Journal of Hazardous Materials, 2022, 421: 126728
[89] Chen H Y, Bai X M, Li Y Z, et al.Characterization and source-tracking of antibiotic resistomes in the sediments of a peri-urban river[J].The Science of the Total Environment, 2019, 679: 88-96
[90] Ding J, An X L, Lassen S B, et al.Heavy metal-induced co-selection of antibiotic resistance genes in the gut microbiota of collembolans[J].The Science of the Total Environment, 2019, 683: 210-215
[91] Chen Y R, Guo X P, Niu Z S, et al.Antibiotic resistance genes(ARGs)and their associated environmental factors in the Yangtze Estuary, China: From inlet to outlet[J].Marine Pollution Bulletin, 2020, 158: 111360
[92] 屈沙沙, 朱会卷, 刘锋平, 等.微塑料吸附行为及对生物影响的研究进展[J].环境卫生学杂志, 2017, 7(1): 75-78
Qu S S, Zhu H J, Liu F P, et al.Adsorption behavior and effect on biont of microplastic[J].Journal of Environmental Hygiene, 2017, 7(1): 75-78(in Chinese)
[93] Wang S S, Xue N N, Li W F, et al.Selectively enrichment of antibiotics and ARGs by microplastics in river, estuary and marine waters[J].The Science of the Total Environment, 2020, 708: 134594
[94] Yang Y Y, Liu G H, Song W J, et al.Plastics in the marine environment are reservoirs for antibiotic and metal resistance genes[J].Environment International, 2019, 123: 79-86
[95] Liao H P, Li X, Yang Q E, et al.Herbicide selection promotes antibiotic resistance in soil microbiomes[J].Molecular Biology and Evolution, 2021, 38(6): 2337-2350
[96] 文汉卿, 史俊, 寻昊, 等.抗生素抗性基因在水环境中的分布、传播扩散与去除研究进展[J].应用生态学报, 2015, 26(2): 625-635
Wen H Q, Shi J, Xun H, et al.Distribution, dissemination and removal of antibiotic resistant genes(ARGs)in the aquatic environment[J].Chinese Journal of Applied Ecology, 2015, 26(2): 625-635(in Chinese)
[97] Sui Q W, Zhang J Y, Chen M X, et al.Distribution of antibiotic resistance genes(ARGs)in anaerobic digestion and land application of swine wastewater[J].Environmental Pollution, 2016, 213: 751-759
[98] Zhao H X, Yan B, Mo X Y, et al.Prevalence and proliferation of antibiotic resistance genes in the subtropical mangrove wetland ecosystem of South China Sea[J].MicrobiologyOpen, 2019, 8(11): e871
[99] Tang X J, Lou C L, Wang S X, et al.Effects of long-term manure applications on the occurrence of antibiotics and antibiotic resistance genes(ARGs)in paddy soils: Evidence from four field experiments in south of China[J].Soil Biology and Biochemistry, 2015, 90: 179-187
[100] Cruz-Loya M, Kang T M, Lozano N A, et al.Stressor interaction networks suggest antibiotic resistance co-opted from stress responses to temperature[J].The ISME Journal, 2019, 13(1): 12-23
[101] Guo J H, Li J, Chen H, et al.Metagenomic analysis reveals wastewater treatment plants as hotspots of antibiotic resistance genes and mobile genetic elements[J].Water Research, 2017, 123: 468-478
[102] Jiang C X, Diao X P, Wang H H, et al.Diverse and abundant antibiotic resistance genes in mangrove area and their relationship with bacterial communities—A study in Hainan Island, China[J].Environmental Pollution, 2021, 276: 116704
[103] 李十盛, 高会, 赵富强, 等.水产养殖环境中抗生素抗性基因的研究进展[J].中国环境科学, 2021, 41(11): 5314-5325
Li S S, Gao H, Zhao F Q, et al.Research progress on the occurrence and influencing factors of antibiotic resistance genes in aquaculture environment[J].China Environmental Science, 2021,41(11): 5314-5325(in Chinese)
[104] Hatha M, Vivekanandhan A A, Joice G J, et al.Antibiotic resistance pattern of motile aeromonads from farm raised fresh water fish[J].International Journal of Food Microbiology, 2005, 98(2): 131-134
[105] Kumaran S, Deivasigamani B, Alagappan K, et al.Antibiotic resistant Esherichia colistrains from seafood and its susceptibility to seaweed extracts[J].Asian Pacific Journal of Tropical Medicine, 2010, 3(12): 977-981
[106] 蔡天贵, 张龙, 张晋东.抗生素抗性基因的生态风险研究进展[J].应用生态学报, 2022, 33(5): 1435-1440
Cai T G, Zhang L, Zhang J D.Research advances in ecological risk of antibiotic resistance genes[J].Chinese Journal of Applied Ecology, 2022, 33(5): 1435-1440(in Chinese)
[107] Tomova A, Ivanova L, Buschmann A H, et al.Antimicrobial resistance genes in marine bacteria and human uropathogenic Escherichia colifrom a region of intensive aquaculture[J].Environmental Microbiology Reports, 2015, 7(5): 803-809
[108] 龙文芳, 李天娇, 黄飞羽, 等.海水浴场革兰阴性菌的检测及耐药性研究[J].环境与健康杂志, 2016, 33(3): 226-228
Long W F, Li T J, Huang F Y, et al.Screening and antibiotic resistance of Gram negative bacteria in bathing beach[J].Journal of Environment and Health, 2016, 33(3): 226-228(in Chinese)
[109] Leonard A F, Morris D, Schmitt H, et al.Natural recreational waters and the risk that exposure to antibiotic resistant bacteria poses to human health[J].Current Opinion in Microbiology, 2022, 65: 40-46
[110] Ma L P, Xia Y, Li B, et al.Metagenomic assembly reveals hosts of antibiotic resistance genes and the shared resistome in pig, chicken, and human feces[J].Environmental Science &Technology, 2016, 50(1): 420-427
[111] Hendriksen R S, Munk P, Njage P, et al.Global monitoring of antimicrobial resistance based on metagenomics analyses of urban sewage[J].Nature Communications, 2019, 10(1): 1124
[112] Zhang Z Y, Zhang Q, Wang T Z, et al.Assessment of global health risk of antibiotic resistance genes[J].Nature Communications, 2022, 13(1): 1553