N-亚硝胺类化合物(N-nitrosamines, NAs)是指结构通式为R1(R2)N—N![]() O的化合物,其中R1、R2是不同的取代基团。NAs存在于食品、烟草、饮用水、药物和化妆品中[1-2],同时在地下水、土壤等环境介质中也有检出[3-4]。自1956年N-二甲基亚硝胺(NDMA)被证明容易诱导大鼠肝脏恶性肿瘤的发生以来[5],迄今发现了200多种NAs具有致癌活性[6],并且NAs会在包括高等灵长动物在内的40多种动物中诱发癌症[7]。人体受NAs影响会导致食道癌、胃癌、鼻咽癌、乳腺癌等癌症的发生[7-8]。细胞色素P450酶(P450酶)在环境污染物的生物转化过程中起着重要的作用,大约2/3人类酶活化致癌物的反应都与P450酶有关[9-10]。NAs进入生物体后,可被P450酶代谢生成比母体化合物毒性更强的代谢产物[11]。其中,经过P450酶代谢后形成的羟基化产物会自发分解,形成烷基重氮离子,与DNA发生加合反应,从而激活NAs的致癌的潜力[12-13]。因此,明确P450酶代谢NAs的反应机理对于NAs致癌性的研究有着极其重要的作用。
O的化合物,其中R1、R2是不同的取代基团。NAs存在于食品、烟草、饮用水、药物和化妆品中[1-2],同时在地下水、土壤等环境介质中也有检出[3-4]。自1956年N-二甲基亚硝胺(NDMA)被证明容易诱导大鼠肝脏恶性肿瘤的发生以来[5],迄今发现了200多种NAs具有致癌活性[6],并且NAs会在包括高等灵长动物在内的40多种动物中诱发癌症[7]。人体受NAs影响会导致食道癌、胃癌、鼻咽癌、乳腺癌等癌症的发生[7-8]。细胞色素P450酶(P450酶)在环境污染物的生物转化过程中起着重要的作用,大约2/3人类酶活化致癌物的反应都与P450酶有关[9-10]。NAs进入生物体后,可被P450酶代谢生成比母体化合物毒性更强的代谢产物[11]。其中,经过P450酶代谢后形成的羟基化产物会自发分解,形成烷基重氮离子,与DNA发生加合反应,从而激活NAs的致癌的潜力[12-13]。因此,明确P450酶代谢NAs的反应机理对于NAs致癌性的研究有着极其重要的作用。
P450酶代谢的α-羟基化反应是NAs代谢活化的关键步骤[14]。测定动力学同位素效应的实验结果表明,由P450酶活性中心进行的氢提取过程是α-羟基化反应的速率决定步骤[15]。铁(Ⅳ)-氧血红素阳离子自由基化合物Ⅰ(compound Ⅰ, Cpd Ⅰ)是被普遍认可的代谢活性物种,由P450酶在其催化循环过程中形成[16]。Cpd Ⅰ的[Fe![]() O]部分可以对NDMA甲基上的氢原子进行提取,羟基反弹到α-亚硝胺自由基,从而完成羟基化过程[17]。由于Cpd Ⅰ反应活性高、存在寿命短,通常很难在实验中对其进行捕捉和表征,理论计算可以作为研究P450酶代谢机理的有效手段[18]。已有研究者对P450酶代谢活化少量典型的NAs (例如:N-甲基-N-亚硝基甲胺、4-甲基亚硝氨基-1-(3-吡啶基)-1-丁酮)的反应进行了密度泛函理论(DFT)计算,分析了P450酶代谢这些NAs的反应路径,包括羟基化反应、脱亚硝基反应、N-氧化反应[17-20]。Li和Hecht[21]总结了人类频繁接触的10种NAs,分为环状和非环状2种类型讨论了其致癌活化的反应过程。然而,已有的研究是仅针对某种具体化合物的反应,很难判别种类众多的NAs的反应活性和致癌潜力。因此,本研究借助理论计算方法,基于活化反应机理,探究不同NAs的代谢规律。
O]部分可以对NDMA甲基上的氢原子进行提取,羟基反弹到α-亚硝胺自由基,从而完成羟基化过程[17]。由于Cpd Ⅰ反应活性高、存在寿命短,通常很难在实验中对其进行捕捉和表征,理论计算可以作为研究P450酶代谢机理的有效手段[18]。已有研究者对P450酶代谢活化少量典型的NAs (例如:N-甲基-N-亚硝基甲胺、4-甲基亚硝氨基-1-(3-吡啶基)-1-丁酮)的反应进行了密度泛函理论(DFT)计算,分析了P450酶代谢这些NAs的反应路径,包括羟基化反应、脱亚硝基反应、N-氧化反应[17-20]。Li和Hecht[21]总结了人类频繁接触的10种NAs,分为环状和非环状2种类型讨论了其致癌活化的反应过程。然而,已有的研究是仅针对某种具体化合物的反应,很难判别种类众多的NAs的反应活性和致癌潜力。因此,本研究借助理论计算方法,基于活化反应机理,探究不同NAs的代谢规律。
反应物的键能是预测其反应性的重要指标[22]。特别地,氢原子提取的反应活化能与C—H键的解离能(BDEC-H)呈显著相关[23-24]。理论计算存在着耗时、对硬件设备要求高等问题,体系复杂的反应计算难度也会较大[23]。但是,BDEC-H的计算相对来说体系简单,计算方便快捷。因此,本研究将通过BDEC-H来预测NAs氢提取反应的活化能,有助于快速地判断脱烷基的反应活性。
本研究通过DFT计算,得到BDEC-H,并且计算了链状和环状NAs的反应活化能。通过数学模型构建NAs的α-羟基化反应活化能与BDEC-H的相关关系,揭示NAs代谢规律,以此对不同种类的NAs的反应性和致癌潜力进行快速有效的评估;同时,对含有C![]() C键的NAs羟基化和环氧化的区域选择性进行分析,以加深对P450酶催化不同类型NAs反应的理解。
C键的NAs羟基化和环氧化的区域选择性进行分析,以加深对P450酶催化不同类型NAs反应的理解。
1 计算方法(Computational methods)
1.1 DFT计算
本研究选取六配体三自由基复合物[Fe4+O2-(C20N4H12)-1(SH)-1](Cpd Ⅰ)作为活性位点模型,NAs(结构式见表1)作为P450酶催化反应的底物,对Cpd Ⅰ在高自旋(HS)四重态或低自旋(LS)二重态下进行考察[25]。几何结构采用非限制性的DFT优化,B3LYP杂化密度泛函结合LANL2DZ基组用于Fe,而6-31G**基组用于C、H、N、O原子(记作UB3LYP/BSⅠ,简称BSⅠ)。采用B3LYP的原因是它可以准确地重现P450酶催化反应的动力学同位素效应的测量值[26]和P450酶亚铁血红素的电子顺磁共振参数值[27],形成与结晶结构一致的几何形状[28],并且得到相对于基准CASSCF计算更加精准的能量值[29]。
表1 亚硝胺类化合物结构、C—H键解离能(BDEC-H)和四重态Cpd Ⅰ介导的氢提取反应活化能(ΔE*)以及分子描述符
Table 1 Structures, bond dissociation energies (BDEC-H) and activation energies (ΔE*) of hydrogen-abstraction by Cpd Ⅰ in HS state and molecular descriptors of NAs
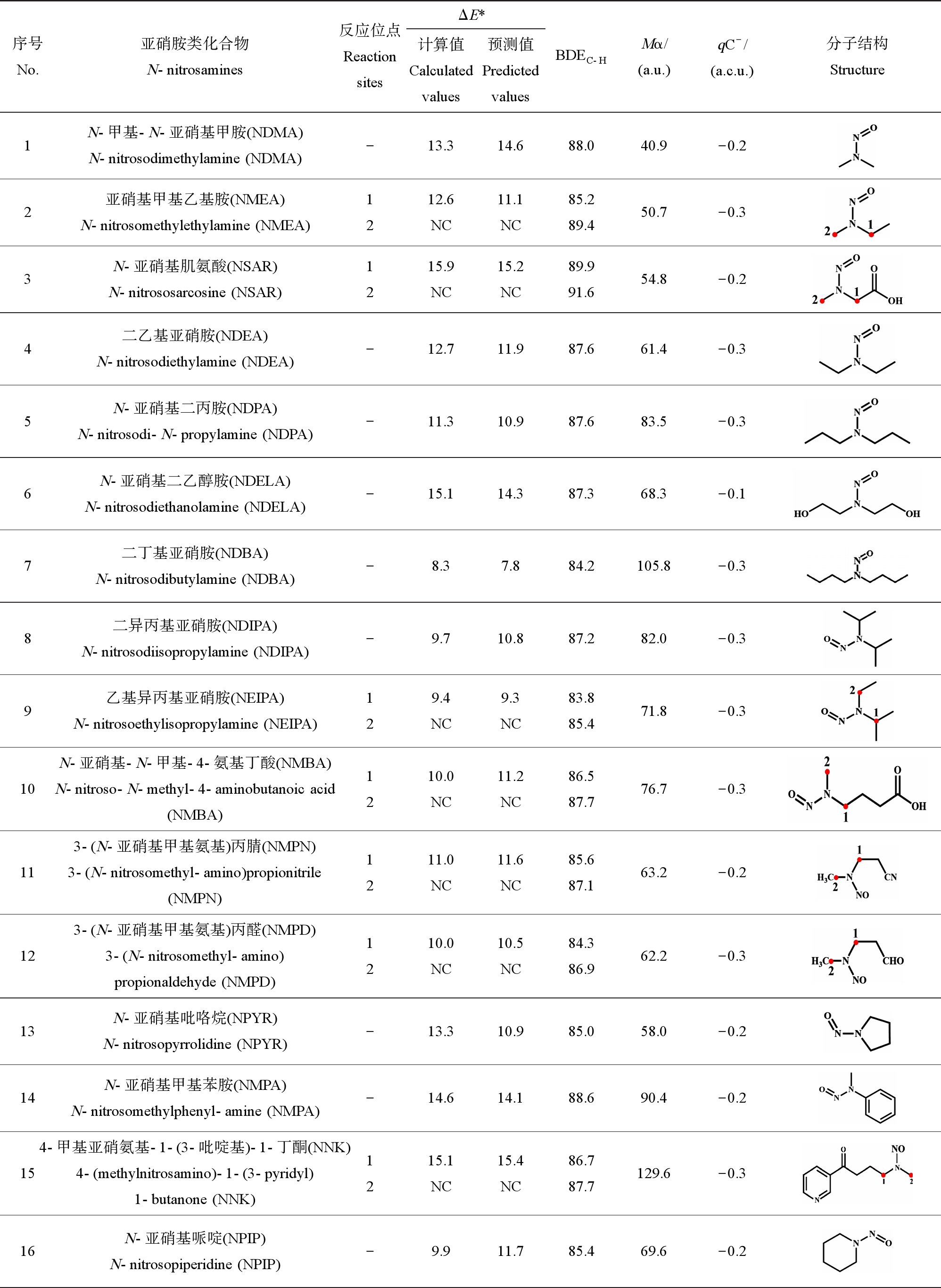
序号No.亚硝胺类化合物N-nitrosamines反应位点Reaction sitesΔE*计算值Calculated values预测值PredictedvaluesBDEC-HMα/(a.u.)qC-/(a.c.u.)分子结构Structure1N-甲基-N-亚硝基甲胺(NDMA)N-nitrosodimethylamine (NDMA)-13.314.688.040.9-0.22亚硝基甲基乙基胺(NMEA)N-nitrosomethylethylamine (NMEA)1212.6NC11.1NC85.289.450.7-0.33N-亚硝基肌氨酸(NSAR)N-nitrososarcosine (NSAR)1215.9NC15.2NC89.991.654.8-0.24二乙基亚硝胺(NDEA)N-nitrosodiethylamine (NDEA)-12.711.987.661.4-0.35N-亚硝基二丙胺(NDPA)N-nitrosodi-N-propylamine (NDPA)-11.310.987.683.5-0.36N-亚硝基二乙醇胺(NDELA)N-nitrosodiethanolamine (NDELA)-15.114.387.368.3-0.17二丁基亚硝胺(NDBA)N-nitrosodibutylamine (NDBA)-8.37.884.2105.8-0.38二异丙基亚硝胺(NDIPA)N-nitrosodiisopropylamine (NDIPA)-9.710.887.282.0-0.39乙基异丙基亚硝胺(NEIPA)N-nitrosoethylisopropylamine (NEIPA)129.4NC9.3NC83.885.471.8-0.310N-亚硝基-N-甲基-4-氨基丁酸(NMBA)N-nitroso-N-methyl-4-aminobutanoic acid(NMBA)1210.0NC11.2NC86.587.776.7-0.3113-(N-亚硝基甲基氨基)丙腈(NMPN)3-(N-nitrosomethyl-amino)propionitrile(NMPN)1211.0NC11.6NC85.687.163.2-0.2123-(N-亚硝基甲基氨基)丙醛(NMPD)3-(N-nitrosomethyl-amino)propionaldehyde (NMPD)1210.0NC10.5NC84.386.962.2-0.313N-亚硝基吡咯烷(NPYR)N-nitrosopyrrolidine (NPYR)-13.310.985.058.0-0.214N-亚硝基甲基苯胺(NMPA)N-nitrosomethylphenyl-amine (NMPA)-14.614.188.690.4-0.2154-甲基亚硝氨基-1-(3-吡啶基)-1-丁酮(NNK)4-(methylnitrosamino)-1-(3-pyridyl) 1-butanone (NNK)1215.1NC15.4NC86.787.7129.6-0.316N-亚硝基哌啶(NPIP)N-nitrosopiperidine (NPIP)-9.911.785.469.6-0.2
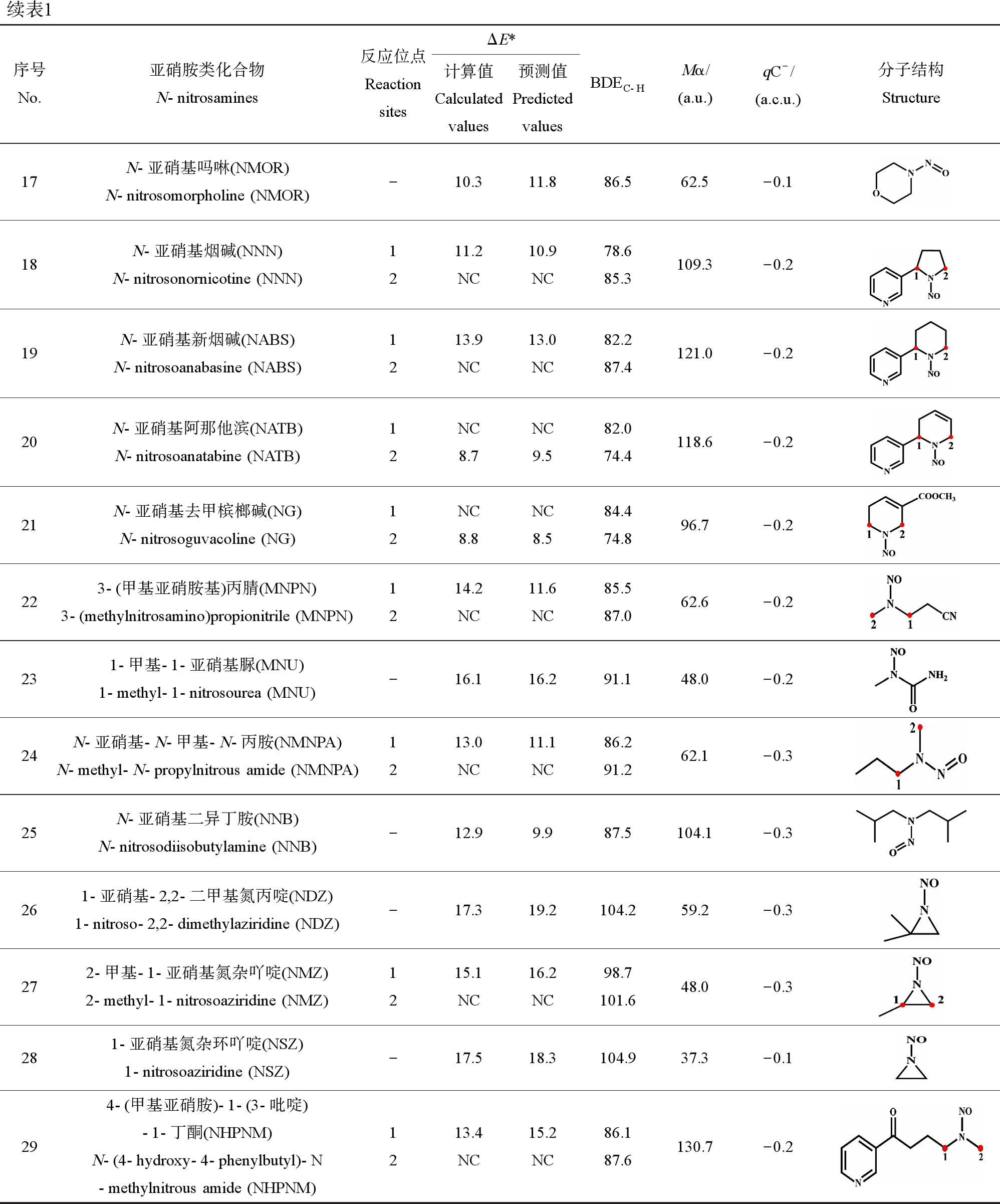
注:BDEC-H为解离键能,kcal·mol-1 (1 kcal = 4.1859 kJ);Mα为平均分子极化率;qC-为分子中碳原子的最负静电荷;ΔE*为氢提取活化能垒,kcal·mol-1 (1 kcal = 4.1859 kJ);-表示该化合物无反应位点区分;NC表示未计算该反应位点的ΔE*。
Note: BDEC-H is bond dissociation energies of the C—H bond, kcal·mol-1 (1 kcal = 4.1859 kJ); Mα is the average molecular polarizability; qC- is the most negative electrostatic charge of the carbon atom in the molecule; ΔE*is the activation energies of hydrogen-abstraction, kcal·mol-1 (1 kcal = 4.1859 kJ); - indicates that the reaction sites of the compound are not different; NC indicates that ΔE*was not calculated.
续表1序号No.亚硝胺类化合物N-nitrosamines反应位点Reaction sitesΔE*计算值Calculated values预测值PredictedvaluesBDEC-HMα/(a.u.)qC-/(a.c.u.)分子结构Structure17N-亚硝基吗啉(NMOR)N-nitrosomorpholine (NMOR)-10.311.886.562.5-0.118N-亚硝基烟碱(NNN)N-nitrosonornicotine (NNN)1211.2NC10.9NC78.685.3109.3-0.219N-亚硝基新烟碱(NABS)N-nitrosoanabasine (NABS)1213.9NC13.0NC82.287.4121.0-0.220N-亚硝基阿那他滨(NATB)N-nitrosoanatabine (NATB)12NC8.7NC9.582.074.4118.6-0.221N-亚硝基去甲槟榔碱(NG)N-nitrosoguvacoline (NG)12NC8.8NC8.584.474.896.7-0.2223-(甲基亚硝胺基)丙腈(MNPN)3-(methylnitrosamino)propionitrile (MNPN)1214.2NC11.6NC85.587.062.6-0.2231-甲基-1-亚硝基脲(MNU)1-methyl-1-nitrosourea (MNU)-16.116.291.148.0-0.224N-亚硝基-N-甲基-N-丙胺(NMNPA)N-methyl-N-propylnitrous amide (NMNPA)1213.0NC11.1NC86.291.262.1-0.325N-亚硝基二异丁胺(NNB)N-nitrosodiisobutylamine (NNB)-12.99.987.5104.1-0.3261-亚硝基-2,2-二甲基氮丙啶(NDZ)1-nitroso-2,2-dimethylaziridine (NDZ)-17.319.2104.259.2-0.3272-甲基-1-亚硝基氮杂吖啶(NMZ)2-methyl-1-nitrosoaziridine (NMZ)1215.1NC16.2NC98.7101.648.0-0.3281-亚硝基氮杂环吖啶(NSZ)1-nitrosoaziridine (NSZ)-17.518.3104.937.3-0.1294-(甲基亚硝胺)-1-(3-吡啶)-1-丁酮(NHPNM)N-(4-hydroxy-4-phenylbutyl)-N-methylnitrous amide (NHPNM)1213.4NC15.2NC86.187.6130.7-0.2
反应分子的振动频率进一步用于零点能(ZPE)校正,并且在298.15 K和101.325 kPa的条件下进行吉布斯自由能校正。通过振动模式分析,所有的基态的频率都为实频,过渡态只有一个虚频,振动方向沿着反应坐标方向。采用PCM连续化溶剂模型(介电常数ε=5.7,氯苯)模拟酶环境,评估周围蛋白质的极化效应[30]。通过UB3LYP-D3/BSⅠ基组进行单点能计算得到D3色散校正项,以此弥补B3LYP方法所缺乏的对色散相互作用的考虑[31]。采用计算铁原子的SDD基组,计算其他原子的6-311++G**基组(记作UB3LYP/BSⅡ/BSⅠ,简称BSⅡ),在更高水平下进行单点(SP)计算以获得更加精确的能量[32]。本研究中反应的相对自由能是在BSⅠ水平下进行结构优化,与ZPE校正、B3LYP/BSⅡ单点能校正、PCM溶剂化校正和色散D3校正结合来估计的。
BDEC-H与羟基化的活化能紧密相关[24],选取BDEC-H作为评估反应底物的NAs的反应性的重要参数。BDEC-H是指氢原子和解离掉氢原子的自由基的能量之和减去底物原始的能量,表示底物解离一个氢原子的难易程度[33]。在BSⅠ水平上进行焓能校正,获得NAs的BDEC-H能量。所有计算均由Gaussian16软件包完成[34]。
1.2 模型构建
为了考察BDEC-H与羟基化的活化能紧密相关性,使用模型预测相应的活化能。借助羟基化的活化能作为终点值,BDEC-H以及NAs的电荷等量子化学描述符作为变量,使用Weka 3.0进行多元线性回归(MLR)模拟[35]获得模型的拟合可决系数(R2)和平均绝对误差(MAE),并且对8种相似结构的NAs[36-40](化合物22~化合物25为链状:MNPN、MNU、NMNPA、NNB;化合物26~化合物29为环状:NDZ、NMZ、NSZ、NHPNM)进行外部验证。对模型进行内部去一法交叉验证以考察模型的稳健性,得到交叉验证的预测相关系数![]() 和均方根误差(RMSECV)。其中,拟合优度评价系数(R2和MAE)的计算公式如下,其余公式参考文献[41]:
和均方根误差(RMSECV)。其中,拟合优度评价系数(R2和MAE)的计算公式如下,其余公式参考文献[41]:
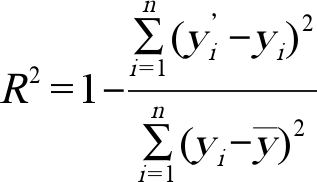
(1)
(2)
式中:![]() 为模型回归拟合值;yi为实验值;
为模型回归拟合值;yi为实验值;![]() 为实验值的平均值;
为实验值的平均值;![]() 为预测值;n为数据个数。
为预测值;n为数据个数。
2 结果(Results)
2.1 DFT计算结果
根据有无环状结构,将21种NAs分为链状(化合物1~化合物12)和环状(化合物13~化合物21)2种类别。NMEA (化合物2)、NSAR (化合物3)、NDEA (化合物4)、NEIPA (化合物9)、NMBA (化合物10)、NMPN (化合物11)、NMPD (化合物12)、NMPA (化合物14)、NNK (化合物15)、NNN (化合物18)、NABS (化合物19)、NATB (化合物20)、NG (化合物21)的两侧取代基不同,有2个α-C,存在2个不同的反应位点。DFT计算21种NAs的所有α-C的BDEC-H,BDEC-H在74.4~91.6 kcal·mol-1(表1)范围内,链状NAs的BDEC-H为83.8~91.6 kcal·mol-1,环状NAs的BDEC-H为74.4~88.6 kcal·mol-1 (1 kcal = 4.1859 kJ)。其中,BDEC-H最大的是NSAR (化合物3),其反应位点2(91.6 kcal·mol-1,1 kcal = 4.1859 kJ),NSAR为链状化合物,存在与碳相连的羧基;最小的是NATB (化合物20),其反应位点2(74.4 kcal·mol-1,1 kcal = 4.1859 kJ),NATB为环状化合物,存在环内双键。
根据BDEC-H选取C—H键断裂的α-C所在的反应位置,BDEC-H越小代表C—H键的断裂所需要的能量越低,断键越容易[42]。进一步对BDEC-H较小的反应位点上Cpd Ⅰ在四重态下氢提取步骤的活化能进行计算。在进行零点能校正、B3LYP/BSⅡ单点能校正、PCM溶剂化(ε = 5.7,氯苯)校正和色散D3校正后,得到Cpd Ⅰ在四重态下进行氢提取步骤的活化能结果(表1)。链状化合物活化能ΔE*的范围为8.3~15.9 kcal·mol-1,环状化合物活化能ΔE*的范围为8.7~15.1 kcal·mol-1 (1 kcal = 4.1859 kJ)。其中,ΔE*最大的是链状化合物NSAR(化合物3),其活化能为15.9 kcal·mol-1;最小的是链状化合物NDBA(化合物7),活化能为8.3 kcal·mol-1 (1 kcal = 4.1859 kJ),其取代基为2个正丁基。
2.2 MLR模型的建立
为了有效预测环境中不同类型NAs的α-羟基化反应性,本研究建立了21种NAs在Cpd Ⅰ氢提取过程的活化能与BDEC-H相关的模型并验证模型的稳健性。21种NAs的氢提取活化能垒ΔE*作为终点,借助Weka软件筛选了化合物的BDEC-H、2种电荷描述符(分子中碳原子最正静电荷qC+和碳原子最负静电荷qC-)和部分量子化学描述符(例如:分子平均极化率Mα)。多元线性回归构建的模型2.1如下:
ΔE*= 0.3×BDEC-H + 9.7×qC--11.4
(3)
模型2.1的R2为0.6,MAE为1.5 kcal·mol-1 (1 kcal = 4.1859 kJ)。由于R2较小,根据21种NAs的结构,将其分为12种链状化合物以及9种环状化合物,分别进行模型构建,得到12种链状NAs如下结果:
ΔE*=0.6×BDEC-H -4.9×10-2×Mα+11.0×qC--32.2
(4)
9种环状NAs如下结果:
ΔE*=0.4×BDEC-H + 5.3×10-2× Mα-28.6
(5)
模型2.2的R2为0.9,MAE为0.8 kcal·mol-1,4种链状NAs的外部验证结果R2为0.9,MAE为2.6 kcal·mol-1;模型2.3的R2为0.9,MAE为1.0 kcal·mol-1,4种环状NAs的外部验证结果R2为0.9,MAE为1.7 kcal·mol-1 (1 kcal = 4.1859 kJ)。模型2.2和2.3的ΔE*的实验值与预测值拟合图如图1(a)和图1(b),可知模型具有较好的拟合能力和预测能力。对模型2.2和2.3进行去一法交叉验证,得到的交叉验证系数,![]() 为0.8,RMSECV为1.5和
为0.8,RMSECV为1.5和![]() 为0.6,RMSECV为2.2,表明模型具有较好的稳健性。采用这一模型可以快速有效地评估NAs在Cpd Ⅰ氢提取过程中的活化能,进而对NAs的反应活性进行评估,外部验证结果也证明了这一点。
为0.6,RMSECV为2.2,表明模型具有较好的稳健性。采用这一模型可以快速有效地评估NAs在Cpd Ⅰ氢提取过程中的活化能,进而对NAs的反应活性进行评估,外部验证结果也证明了这一点。
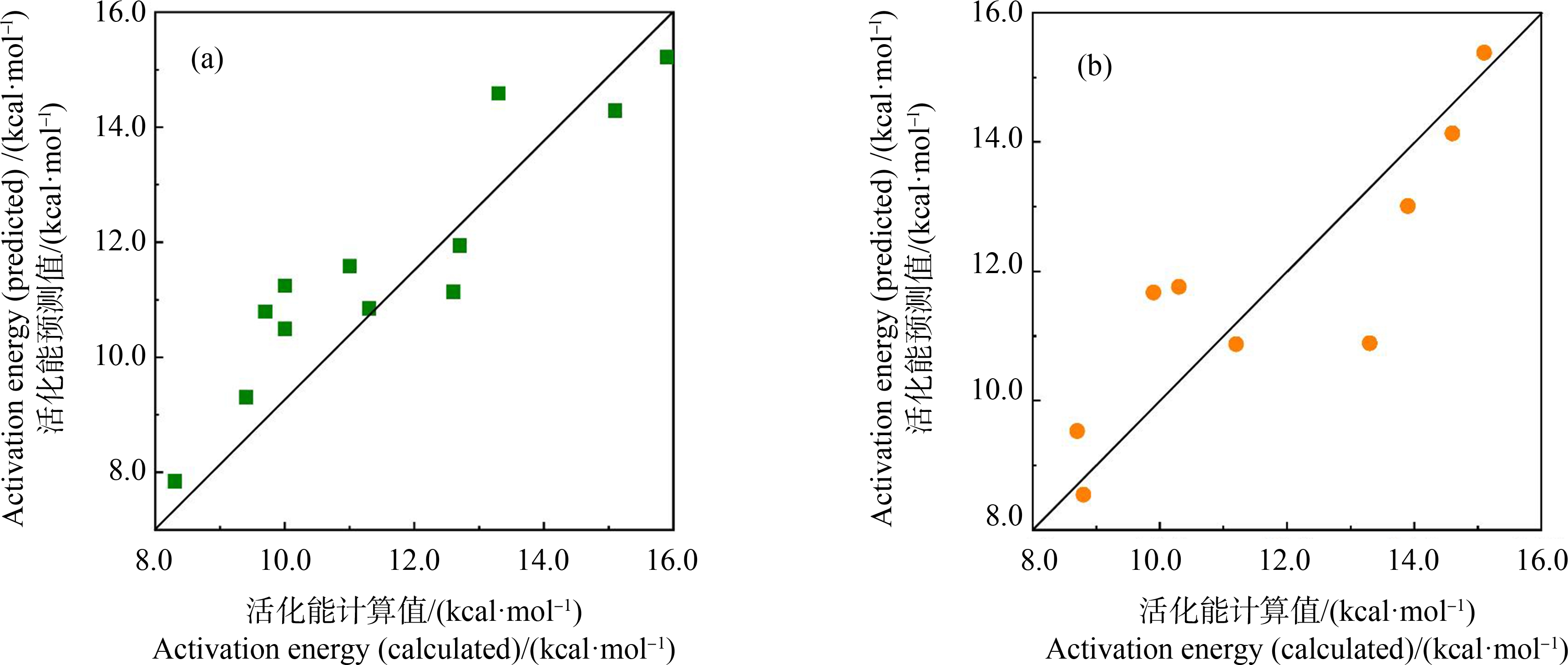
图1 活化能的计算值和预测值模型(1 kcal = 4.1859 kJ):(a)为模型2.2,(b)为模型2.3
Fig. 1 The activation energy calculated and predicted values of models (1 kcal = 4.1859 kJ): (a) for model 2.2 and (b) for model 2.3
模型筛选出的描述符表明,无论链状还是环状NAs,其BDEC-H均与活化能呈正相关关系,表明解离能越大的化合物,P450酶进行催化氧化时所需要的能量越大。分子中的负电荷数和平均分子极化率也可能对活化能产生影响。具有较小qC-值的化合物分子中碳原子上集中了较多的负原子电荷,且qC-值均在亚硝基相邻的碳上。因此,推测亚硝基相邻的碳上具有较多的负电荷,可能对活化能垒产生影响。本文中选择的亚硝胺类化合物是极性化合物,具有极性基团N—N![]() O,其结构的参与会影响R1和R2的电子排布。分子极化是电荷再分配过程,其相关分子性质是分子平均极化率[43]。Mα表征平均分子色散力,表征电子云的可变形性,模型表明了电子的分布与活化能相关[44-45]。本研究建立的模型预测C—H键活化能ΔE*,表明其与BDEC-H具有较强的相关关系,以此对不同种类的NAs的反应活性进行快速有效地评估,为NAs的环境健康和生态风险评估提供理论依据。
O,其结构的参与会影响R1和R2的电子排布。分子极化是电荷再分配过程,其相关分子性质是分子平均极化率[43]。Mα表征平均分子色散力,表征电子云的可变形性,模型表明了电子的分布与活化能相关[44-45]。本研究建立的模型预测C—H键活化能ΔE*,表明其与BDEC-H具有较强的相关关系,以此对不同种类的NAs的反应活性进行快速有效地评估,为NAs的环境健康和生态风险评估提供理论依据。
2.3 羟基化和环氧化反应的区域选择性
当P450酶催化反应的底物为NAs,并且取代基又含有C![]() C键时,就存在α-C的羟基化和C
C键时,就存在α-C的羟基化和C![]() C键环氧化反应的竞争关系[46]。羟基化和环氧化是2种典型的P450酶代谢反应,可能经常存在区域选择性的现象。甲基乙烯基亚硝胺(N-nitroso-N-methylvinylamine)是一种典型的NAs,经过大鼠肝微粒体的代谢之后,2种反应途径生成的产物均被检出[47]。因此,以甲基乙烯基亚硝胺为例,考察了P450酶的活性中心Cpd Ⅰ在高自旋四重态和低自旋二重态下介导的α-羟基化以及C
C键环氧化反应的竞争关系[46]。羟基化和环氧化是2种典型的P450酶代谢反应,可能经常存在区域选择性的现象。甲基乙烯基亚硝胺(N-nitroso-N-methylvinylamine)是一种典型的NAs,经过大鼠肝微粒体的代谢之后,2种反应途径生成的产物均被检出[47]。因此,以甲基乙烯基亚硝胺为例,考察了P450酶的活性中心Cpd Ⅰ在高自旋四重态和低自旋二重态下介导的α-羟基化以及C![]() C键环氧化反应的具体路径,揭示了某些含有C
C键环氧化反应的具体路径,揭示了某些含有C![]() C键的NAs在P450酶催化作用下的反应机理。
C键的NAs在P450酶催化作用下的反应机理。
羟基化反应的相对能量分布如图2所示,4,2RC是反应的起点,高自旋四重态和低自旋二重态之间能量简并。反应的第一步是Cpd Ⅰ提取α-C上的氢原子,经过一个过渡态(4TSH = 15.8 kcal·mol-1,2TSH= 14.0 kcal·mol-1,1 kcal = 4.1859 kJ)使C—H键断裂,这一步也是反应的速率决定步骤。此时,氢原子在碳原子和Cpd Ⅰ中的氧原子之间振动,通过接近线性的O···H···C和高虚频(四重态νim = i1792.52 cm-1,二重态νim=i1725.42 cm-1),形成FeⅣ—OH和烷基自由基构成的中间体4,2IM(4IM = 0.5 kcal·mol-1,2IM=0.3 kcal·mol-1,1 kcal=4.1859 kJ)。随后,FeⅣ—OH上的羟基自由基经过无能垒的反弹(四重态4TSreb=-1.5 kcal·mol-1,1 kcal=4.1859 kJ,νim=i129.74 cm-1;二重态无能垒)到烷基自由基上,从而形成强放热的产物(4P=-52.7 kcal·mol-1, 2P=-52.2 kcal·mol-1,1 kcal=4.1859 kJ)。
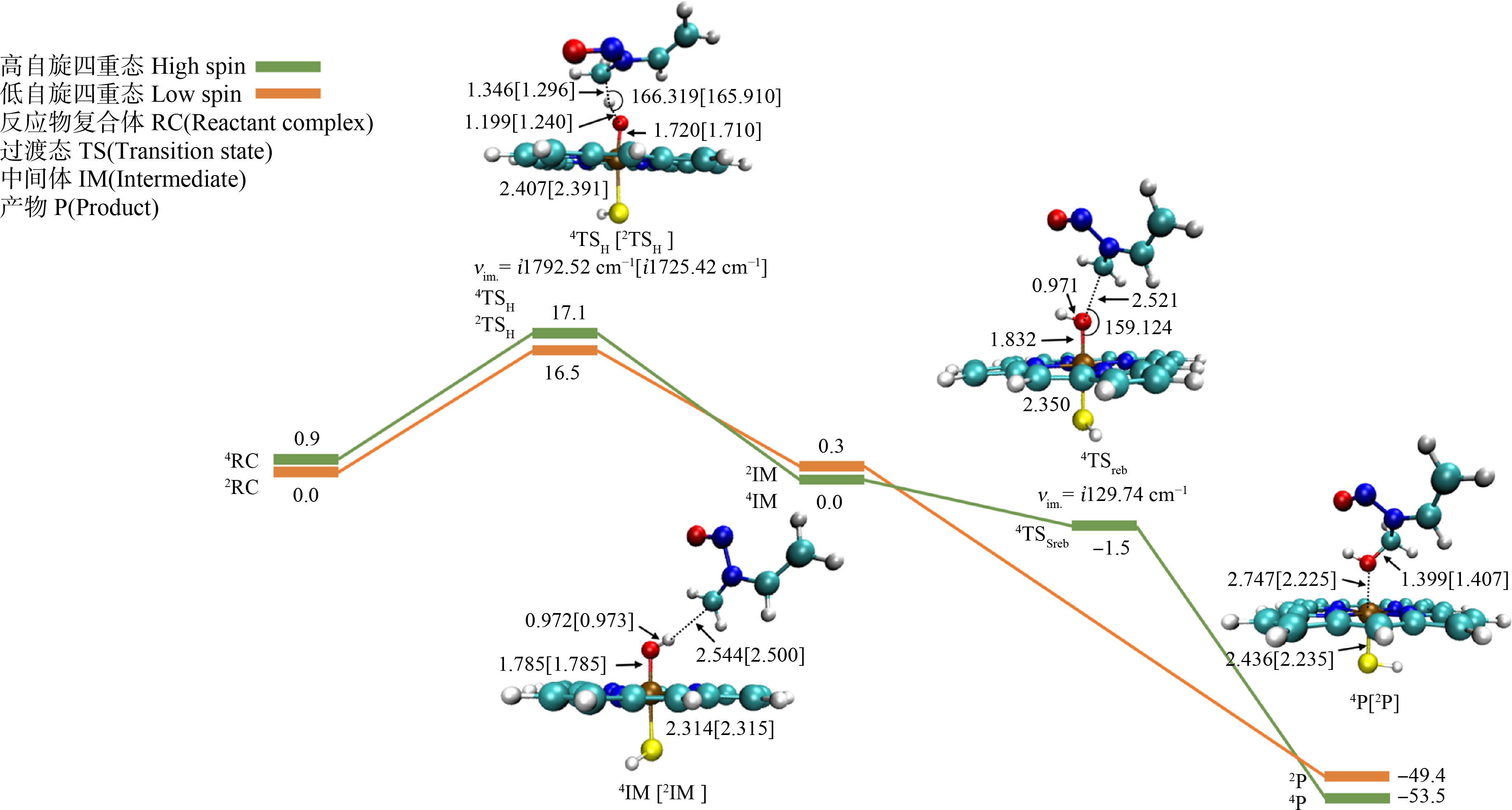
图2 甲基乙烯基亚硝胺经P450酶活性氧化单体Cpd Ⅰ羟基化的能量势能图、四重态HS和二重态LS下关键位点的优化结构
注:键长单位为0.1 nm,顺序为HS [LS];键角单位为(°);活化能量单位为kcal·mol-1 (1 kcal = 4.1859 kJ)。
Fig. 2 Relative energy of hydroxylation of N-nitroso-N-methylvinylamine by Cpd Ⅰ, and the optimized geometries of the important reaction sites in the HS and LS
Note: Geometric parameters (the unit of bond length is 0.1 nm) are given as HS [LS]; the unit of bond angle is (°) and the unit of activation energy is kcal·mol-1 (1 kcal = 4.1859 kJ).
图3为Cpd Ⅰ在简并双态下介导的甲基乙烯基亚硝胺的C![]() C键环氧化的相对能量分布图。4,2RC经过一个较低的过渡态(4TSH=7.8 kcal·mol-1,2TSH=6.4 kcal·mol-1,1 kcal=4.1859 kJ),Cpd Ⅰ中FeⅣ
C键环氧化的相对能量分布图。4,2RC经过一个较低的过渡态(4TSH=7.8 kcal·mol-1,2TSH=6.4 kcal·mol-1,1 kcal=4.1859 kJ),Cpd Ⅰ中FeⅣ ![]() O的氧原子向C
O的氧原子向C![]() C双键上的C原子靠近,形成C—O键从而生成中间产物IM。仅获得四重态时自由基中间体4IM(4IM=-16.9 kcal·mol-1,1 kcal=4.1859 kJ),并且在环氧化的环闭合过程中,四重态下的环闭合过渡态4TSrc能量为-18.6 kcal·mol-1,1 kcal=4.1859 kJ,νim=i221.05 cm-1。二重态为无能垒反应,二重态的自由基中间体的寿命很短,从而快速、自发地形成了最终的环氧化产物[25]。环氧化产物能量4P = -44.1 kcal·mol-1, 2P = -43.6 kcal·mol-1,1 kcal = 4.1859 kJ。四重态下环闭合的过渡态能垒远低于C—O键形成时的过渡态(ΔErc = -1.7 kcal·mol-1,ΔEH = 7.7 kcal·mol-1,1 kcal = 4.1859 kJ),因此C—O键形成时的过渡态是反应的速率决定步骤。针对C—O键的形成,在底物中有2个不同的反应位点,经过初步计算,Cpd Ⅰ上的氧原子与—CH2上的碳原子反应所需要的能量要低于—CH上的碳原子(平均值:Δ
C双键上的C原子靠近,形成C—O键从而生成中间产物IM。仅获得四重态时自由基中间体4IM(4IM=-16.9 kcal·mol-1,1 kcal=4.1859 kJ),并且在环氧化的环闭合过程中,四重态下的环闭合过渡态4TSrc能量为-18.6 kcal·mol-1,1 kcal=4.1859 kJ,νim=i221.05 cm-1。二重态为无能垒反应,二重态的自由基中间体的寿命很短,从而快速、自发地形成了最终的环氧化产物[25]。环氧化产物能量4P = -44.1 kcal·mol-1, 2P = -43.6 kcal·mol-1,1 kcal = 4.1859 kJ。四重态下环闭合的过渡态能垒远低于C—O键形成时的过渡态(ΔErc = -1.7 kcal·mol-1,ΔEH = 7.7 kcal·mol-1,1 kcal = 4.1859 kJ),因此C—O键形成时的过渡态是反应的速率决定步骤。针对C—O键的形成,在底物中有2个不同的反应位点,经过初步计算,Cpd Ⅰ上的氧原子与—CH2上的碳原子反应所需要的能量要低于—CH上的碳原子(平均值:Δ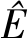 -CH2 = 7.1 kcal·mol-1 vs. Δ
-CH2 = 7.1 kcal·mol-1 vs. Δ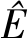 -CH = 8.9 kcal·mol-1,1 kcal = 4.1859 kJ),因此考察反应更容易进行的—CH2上的碳原子,这也与实验所得结果一致[47]。
-CH = 8.9 kcal·mol-1,1 kcal = 4.1859 kJ),因此考察反应更容易进行的—CH2上的碳原子,这也与实验所得结果一致[47]。
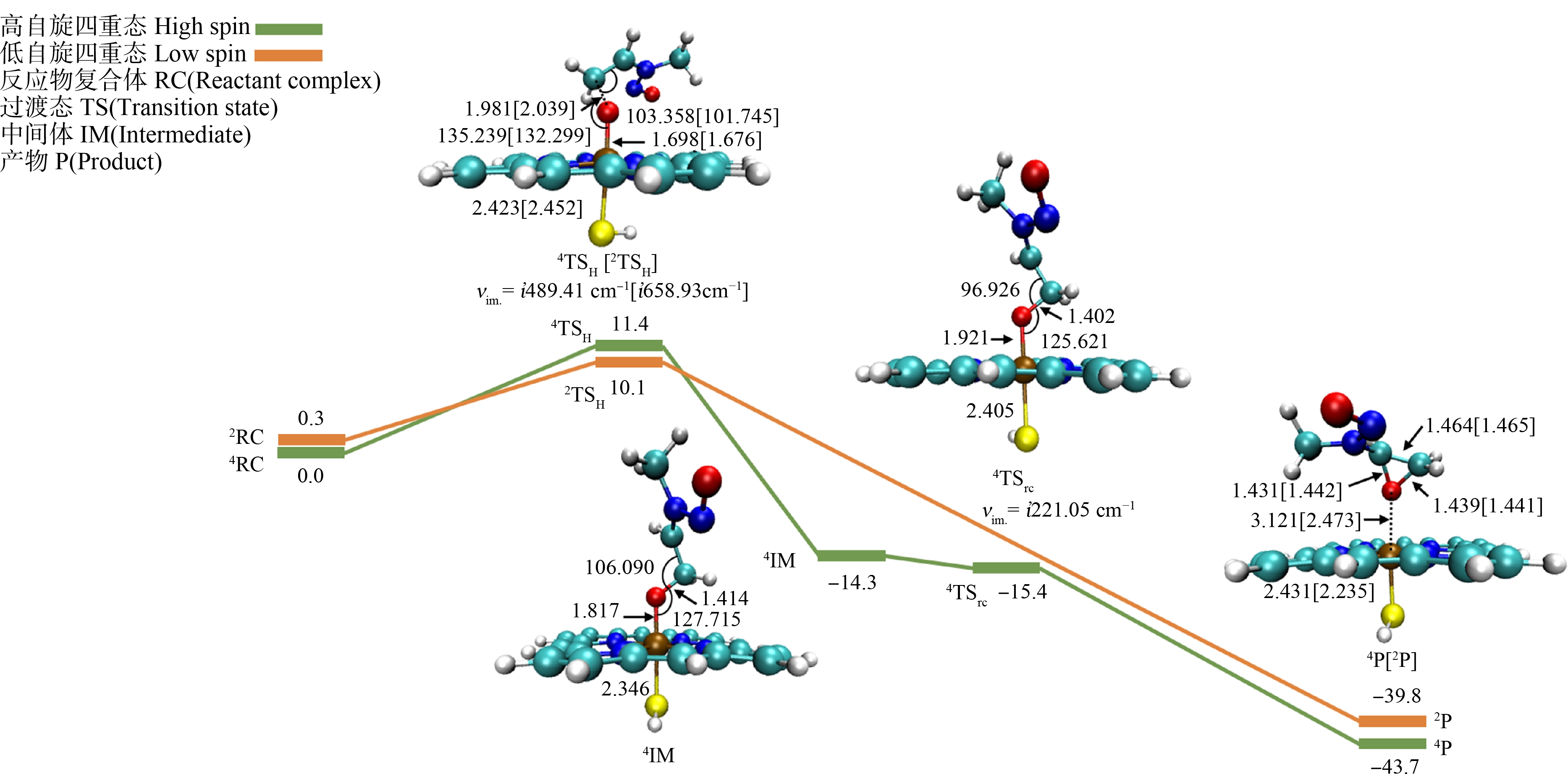
图3 甲基乙烯基亚硝胺经P450酶活性氧化单体Cpd Ⅰ环氧化的相对能量势能图、四重态HS和二重态LS下关键位点的优化结构
注:键长单位为0.1 nm,顺序为HS [LS];键角单位为(°);活化能量单位为kcal·mol-1 (1 kcal = 4.1859 kJ)。
Fig. 3 Relative energy of epoxidation of N-nitroso-N-methylvinylamine by Cpd Ⅰ, and the optimized geometries of the important reaction sites in the HS and LS
Note: Geometric parameters (the unit of bond length is 0.1 nm) are given as HS [LS]; the unit of bond angle is (°) and the unit of activation energy is kcal·mol-1 (1 kcal = 4.1859 kJ).
3 讨论(Discussions)
BDEC-H的计算结果表明,环状NAs的C—H的解离能要略低于链状NAs。对于非对称的链状化合物,较长的支链上的BDEC-H要更小,化合物NMEA (化合物2)、NSAR (化合物3)、NEIPA (化合物9)、NMBA (化合物10)、NMPN (化合物11)、NMPD (化合物12)完全符合这一规律。对于非对称的环状化合物,NNK (化合物15)的亚甲基上的C—H的解离能更小(BDE1 = 86.7 kcal·mol-1;BDE2 = 87.7 kcal·mol-1;1 kcal = 4.1859 kJ),NG (化合物21)的反应位点2的解离能更小(BDE1 = 84.4 kcal·mol-1;BDE2=74.8 kcal·mol-1,1 kcal = 4.1859 kJ)。对于有2个环的NNN (化合物18)和NABS (化合物19),2个环的桥接处(反应位点1)的解离能更小(NNN:BDE1=78.6 kcal·mol-1;BDE2=85.3 kcal·mol-1;NABS:BDE1=82.2 kcal·mol-1;BDE2=87.4 kcal·mol-1;1 kcal=4.1859 kJ)。但NATB (化合物20)的桥接处(反应位点1)解离能却更大(BDE1=82.0 kcal·mol-1;BDE2=74.4 kcal·mol-1;1 kcal = 4.1859 kJ),可能是由于NATB中C![]() C键吸电子的诱导效应导致的。
C键吸电子的诱导效应导致的。
活化能ΔE*的计算结果表明,对于NDMA (化合物1)、NDEA (化合物4)、NDPA (化合物5)、NDBA (化合物7),分子内2个取代基都是相同的烷基,随着烷基链的增长,活化能有逐渐下降的趋势(13.3 kcal·mol-1 > 12.7 kcal·mol-1 > 11.3 kcal·mol-1 >8.3 kcal·mol-1;1 kcal=4.1859 kJ);对于NSAR (化合物3)和NMBA (化合物10),可以看出随着另一个取代基的增长,发生氢提取的亚甲基上的活化能降低了很多(15.9 kcal·mol-1 vs. 10.0 kcal·mol-1;1 kcal=4.1859 kJ)。对于环状NAs,NPYR (化合物13)和NPIP (化合物16),随着环上C原子的增加,α-羟基化的活化能减小(13.3 kcal·mol-1 vs. 9.9 kcal·mol-1;1 kcal = 4.1859 kJ),表明α-羟基化反应更易发生。
量子化学计算结果表明,动力学结果反映了环氧化速决步骤的反应能垒要略低于羟基化反应速决步骤的反应能垒(平均值:Δ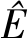 环氧化=7.1 kcal·mol-1 vs. Δ
环氧化=7.1 kcal·mol-1 vs. Δ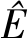 羟基化 = 14.5 kcal·mol-1;1 kcal = 4.1859 kJ)。因此,环氧化反应要比羟基化反应更快发生。然而,热力学结果与动力学不同,羟基化的反应产物要比环氧化产物放热更多,形成的能量更低,更加稳定。羟基化和环氧化相比,羟基化是否发生主要取决于底物的BDEC-H,而环氧化的发生主要取决于底物的电离能[48]。在热力学和动力学2种角度上反映优势产物不同,2种产物理论上均可生成,这与实验中2类产物的检出相一致[47]。
羟基化 = 14.5 kcal·mol-1;1 kcal = 4.1859 kJ)。因此,环氧化反应要比羟基化反应更快发生。然而,热力学结果与动力学不同,羟基化的反应产物要比环氧化产物放热更多,形成的能量更低,更加稳定。羟基化和环氧化相比,羟基化是否发生主要取决于底物的BDEC-H,而环氧化的发生主要取决于底物的电离能[48]。在热力学和动力学2种角度上反映优势产物不同,2种产物理论上均可生成,这与实验中2类产物的检出相一致[47]。
综上所述,本研究使用一种简易的理论计算方法,对细胞色素P450酶催化NAs反应机理进行探究,线性回归模型预测了环状和链状NAs的反应活化能,并且通过理论预测了P450酶代谢的环氧化反应和羟基化反应均可发生,可为NAs的环境健康和生态风险评估提供理论依据。
通信作者简介:季力(1981—),男,博士,教授,主要研究方向为污染物的代谢机制。
[1] Brunnemann K D, Hoffmann D. Analytical studies on tobacco-specific N-nitrosamines in tobacco and tobacco smoke [J]. Critical Reviews in Toxicology, 1991, 21(4): 235-240
[2] Lu S M, Wu D, Li G L, et al. Facile and sensitive determination of N-nitrosamines in food samples by high-performance liquid chromatography via combining fluorescent labeling with dispersive liquid-liquid microextraction [J]. Food Chemistry, 2017, 234: 408-415
[3] Jurado-Sánchez B, Ballesteros E, Gallego M. Comparison of microwave assisted, ultrasonic assisted and Soxhlet extractions of N-nitrosamines and aromatic amines in sewage sludge, soils and sediments [J]. Science of the Total Environment, 2013, 463-464: 293-301
[4] 赵仑山, 岑况, 刘秀丽, 等. 珠江三角洲含N-亚硝胺地下水与地区性癌症[J]. 地学前缘, 2019, 26(2): 335-349
Zhao L S, Cen K, Liu X L, et al. N-nitrosamine containing underground waters and regional cancer incidence in the Pearl River Delta region [J]. Earth Science Frontiers, 2019, 26(2): 335-349 (in Chinese)
[5] Magee P N, Barnes J M. The production of malignant primary hepatic tumours in the rat by feeding dimethylnitrosamine [J]. British Journal of Cancer, 1956, 10(1): 114-122
[6] Hecht S S. Biochemistry, biology, and carcinogenicity of tobacco-specific N-nitrosamines [J]. Chemical Research in Toxicology, 1998, 11(6): 559-603
[7] Tricker A R, Preussmann R. Carcinogenic N-nitrosamines in the diet: Occurrence, formation, mechanisms and carcinogenic potential [J]. Mutation Research/Genetic Toxicology, 1991, 259(3-4): 277-289
[8] Terry P D, Rohan T E. Cigarette smoking and the risk of breast cancer in women: A review of the literature [J]. Cancer Epidemiology, Biomarkers &Prevention: A Publication of the American Association for Cancer Research, Cosponsored by the American Society of Preventive Oncology, 2002, 11(10 Pt 1): 953-971
[9] Chai L H, Zhang H N, Song R Q, et al. Precision biotransformation of emerging pollutants by human cytochrome P450 using computational-experimental synergy: A case study of tris(1,3-dichloro-2-propyl) phosphate [J]. Environmental Science &Technology, 2021, 55(20): 14037-14050
[10] Zhang H N, Song R Q, Guo F J, et al. Using physical organic chemistry knowledge to predict unusual metabolites of synthetic phenolic antioxidants by cytochrome P450 [J]. Chemical Research in Toxicology, 2022, 35(5): 840-848
[11] Sheweita S A, El Banna Y Y, Balbaa M, et al. N-nitrosamines induced infertility and hepatotoxicity in male rabbits [J]. Environmental Toxicology, 2017, 32(9): 2212-2220
[12] Hecht S S. Approaches to cancer prevention based on an understanding of N-nitrosamine carcinogenesis [J]. Proceedings of the Society for Experimental Biology and Medicine Society for Experimental Biology and Medicine, 1997, 216(2): 181-191
[13] Hecht S S. DNA adduct formation from tobacco-specific N-nitrosamines [J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 1999, 424(1-2): 127-142
[14] Lu X F, Heilman J M, Blans P, et al. The structure of DNA dictates purine atom site selectivity in alkylation by primary diazonium ions [J]. Chemical Research in Toxicology, 2005, 18(9): 1462-1470
[15] Chowdhury G, Calcutt M W, Guengerich F P. Oxidation of N-nitrosoalkylamines by human cytochrome P450 2A6: Sequential oxidation to aldehydes and carboxylic acids and analysis of reaction steps [J]. The Journal of Biological Chemistry, 2010, 285(11): 8031-8044
[16] de Visser S P, Ogliaro F, Sharma P K, et al. What factors affect the regioselectivity of oxidation by cytochrome P450? A DFT study of allylic hydroxylation and double bond epoxidation in a model reaction [J]. Journal of the American Chemical Society, 2002, 124(39): 11809-11826
[17] Ji L, Schüürmann G. Computational evidence for α-nitrosamino radical as initial metabolite for both the P450 dealkylation and denitrosation of carcinogenic nitrosamines [J]. The Journal of Physical Chemistry B, 2012, 116(2): 903-912
[18] Ji L. Synergy between experiments and computations: A green channel for revealing metabolic mechanism of xenobiotics in chemical toxicology [J]. Chemical Research in Toxicology, 2020, 33(7): 1539-1550
[19] Ma G C, Yu H Y, Xu T, et al. Computational insight into the activation mechanism of carcinogenic N’-nitrosonornicotine (NNN) catalyzed by cytochrome P450 [J]. Environmental Science &Technology, 2018, 52(20): 11838-11847
[20] Ma G C, Yu H Y, Xu X Q, et al. Molecular basis for metabolic regioselectivity and mechanism of cytochrome P450s toward carcinogenic 4-(methylnitrosamino)-(3-pyridyl)-1-butanone [J]. Chemical Research in Toxicology, 2020, 33(2): 436-447
[21] Li Y P, Hecht S S. Metabolic activation and DNA interactions of carcinogenic N-nitrosamines to which humans are commonly exposed [J]. International Journal of Molecular Sciences, 2022, 23(9): 4559
[22] de Visser S P, Kumar D, Cohen S, et al. A predictive pattern of computed barriers for C-H hydroxylation by compound Ⅰ of cytochrome P450 [J]. Journal of the American Chemical Society, 2004, 126(27): 8362-8363
[23] Olsen L, Rydberg P, Rod T H, et al. Prediction of activation energies for hydrogen abstraction by cytochrome P450 [J]. Journal of Medicinal Chemistry, 2006, 49(22): 6489-6499
[24] Drew K L M, Reynisson J. The impact of carbon-hydrogen bond dissociation energies on the prediction of the cytochrome P450 mediated major metabolic site of drug-like compounds [J]. European Journal of Medicinal Chemistry, 2012, 56: 48-55
[25] Shaik S, Cohen S, Wang Y, et al. P450 enzymes: Their structure, reactivity, and selectivity-modeled by QM/MM calculations [J]. Chemical Reviews, 2010, 110(2): 949-1017
[26] Kumar D, de Visser S P, Shaik S. How does product isotope effect prove the operation of a two-state rebound mechanism in C-H hydroxylation by cytochrome P450? [J]. Journal of the American Chemical Society, 2003, 125(43): 13024-13025
[27] Porro C S, Kumar D, de Visser S P. Electronic properties of pentacoordinated heme complexes in cytochrome P450 enzymes: Search for an Fe(Ⅰ) oxidation state [J]. Physical Chemistry Chemical Physics, 2009, 11(43): 10219-10226
[28] Strickland N, Harvey J N. Spin-forbidden ligand binding to the ferrous-heme group: Ab initio and DFT studies [J]. The Journal of Physical Chemistry B, 2007, 111(4): 841-852
[29] Altun A, Breidung J, Neese F, et al. Correlated ab initio and density functional studies on H2 activation by FeO(·) [J]. Journal of Chemical Theory and Computation, 2014, 10(9): 3807-3820
[30] Miertuš S, Scrocco E, Tomasi J. Electrostatic interaction of a solute with a continuum. A direct utilizaion of ab initio molecular potentials for the prevision of solvent effects [J]. Chemical Physics, 1981, 55(1): 117-129
[31] Grimme S. Semiempirical GGA-type density functional constructed with a long-range dispersion correction [J]. Journal of Computational Chemistry, 2006, 27(15): 1787-1799
[32] Cao X Y, Dolg M, Stoll H.. Valence basis sets for relativistic energy-consistent small-core actinide pseudopotentials [J]. Journal of Chemical Physics, 2003, 118(2): 487-496
[33] Zhang H N, Wang C C, Guo F J, et al. In silico simulation of cytochrome P450-mediated metabolism of aromatic amines: A case study of N-hydroxylation [J]. Ecotoxicology and Environmental Safety, 2022, 237: 113544
[34] Frisch M J, Trucks G W, Schlegel H B, et al. Gaussian, Inc [CP]. Wallingford Ct, 2016
[35] Hall M, Frank E, Holmes G, et al. The WEKA data mining software: An update [J]. ACM SIGKDD Explorations Newsletter, 2009, 11: 10-18
[36] Guan H Y, Feng Y F, Sun B H, et al. NMR assignments of six asymmetrical N-nitrosamine isomers determined in an active pharmaceutical ingredient by DFT calculations [J]. Molecules, 2022, 27(15): 4749
[37] Anna V, Rimma S, Lev O, et al. GC determination of N-nitrosamines by supersonic molecular beam MS equipped with triple quadrupole analyzer, GC/SMB/QQQ/MS [J]. Analytica Chimica Acta, 2011, 685(2): 162-169
[38] 李丕, 白桦, 李海玉, 等. 固相萃取-气相色谱-串联质谱法测定乳胶儿童用品中15种N-亚硝胺及其前体物的迁移量[J]. 色谱, 2014, 32(1): 81-88
Li P, Bai H, Li H Y, et al. Determination of migration of 15 N-nitrosamines and N-nitrosatable substances from children’s latex articles by gas chromatography-tandem mass spectrometry using solid phase extraction [J]. Chinese Journal of Chromatography, 2014, 32(1): 81-88 (in Chinese)
[39] Novak I, ![]() B. Nitrosamines: A challenge for theory and experiment [J]. Chemical Physics Letters, 2007, 445(4-6): 129-132
B. Nitrosamines: A challenge for theory and experiment [J]. Chemical Physics Letters, 2007, 445(4-6): 129-132
[40] Hecht S S. DNA adduct formation from tobacco-specific N-nitrosamines [J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 1999, 424(1-2): 127-142
[41] Organisation for Economic Co-operation and Development (OECD). Guidance document on the validation of (quantitative) structure-activity relationships [(Q)SAR] models. OECD Series on Testing and Assessment No 69 ENV/JM/MONO 2 [R]. Paris: OECD, 2007: 1-154
[42] Zhao S W, Liu L, Fu Y, et al. Assessment of the metabolic stability of the methyl groups in heterocyclic compounds using C-H bond dissociation energies: Effects of diverse aromatic groups on the stability of methyl radicals [J]. Journal of Physical Organic Chemistry, 2005, 18: 353-367
[43] Topham B J, Soos Z. Ionization in organic thin films: Electrostatic potential, electronic polarization, and dopants in pentacene films [J]. Physical Review B, 2011, 84(16): 165405
[44] Yu X L, Wang X Y, Li X B, et al. Quantitative structure-property relationship studies of polymeric cohesive energy by density functional theory [J]. Journal of Polymer Science Part B-Polymer Physics, 2006, 44(2): 409-415
[45] Wang Y, Yang X H, Zhang S Y, et al. Polarizability and aromaticity index govern AhR-mediated potencies of PAHs: A QSAR with consideration of freely dissolved concentrations [J]. Chemosphere, 2021, 268: 129343
[46] Kumar D, Tahsini L, de Visser S P, et al. Effect of porphyrin ligands on the regioselective dehydrogenation versus epoxidation of olefins by oxoiron(Ⅳ) mimics of cytochrome P450 [J]. The Journal of Physical Chemistry A, 2009, 113(43): 11713-11722
[47] Okazaki O, Persmark M, Guengerich F P. N-nitroso-N-methylvinylamine: Reaction of the epoxide with guanyl and adenyl moieties to yield adducts derived from both parts of the molecule [J]. Chemical Research in Toxicology, 1993, 6(2): 168-173
[48] Kumar D, Karamzadeh B, Sastry G N, et al. What factors influence the rate constant of substrate epoxidation by compound Ⅰ of cytochrome P450 and analogous iron(Ⅳ)-oxo oxidants? [J]. Journal of the American Chemical Society, 2010, 132(22): 7656-7667