自主神经系统(autonomic nervous system)是神经系统的重要组成部分,由交感神经和副交感神经两部分自主神经组成,参与心跳、呼吸、体温、泌尿、消化等重要生理功能的调节[1]。化学品通过引起神经元损伤、轴突损伤、髓鞘损伤和影响神经传递[2-3],促进或抑制自主神经作用,进而诱发自主神经功能紊乱(autonomic dysfunction, AD),导致AD症状和疾病发作,如直立性低血压、尿失禁、胆碱能综合征、肾上腺素能综合征、体位性心动过速综合征等[4-7]。例如,氯氮平(clozapine)通过引起肾上腺素能受体阻断,促进交感神经作用,从而引发心律失常[4,8-9]。化学品通过诱发AD,可导致长期不可逆的病变[3],对人体健康造成的危害不容忽视。
化学品诱发AD潜力,可通过动物实验和临床测试评价。在整体动物水平,通过功能观察试验组合(functional observation battery)以及神经学检查进行评价[10]。在临床水平,通过自主神经反射筛查标准化方式,测试心血管、促汗、肾上腺素指标进行评价[11-12]。这些评价方法,虽然可以提供化学品AD的毒性信息,但过程复杂、耗时长且成本高[13]。全球生产和使用的化学品及其混合物超过35万种[14],传统试验方法无法满足种类众多的化学品自主神经毒性测试需求,相关毒性数据大量缺失[15],亟待发展AD化学品的高通量筛查模型。
目前,化学品诱发AD的机制仍不够清楚,尚缺乏AD化学品的机器学习筛查模型。虽然一些研究和数据库提供了AD化学品的阳性数据[4,9,16-17],但阴性数据十分缺乏,这在一定程度上限制了筛查模型的发展。而且,在传统的筛查模型构建范式下,数据集一般需要去除无机物、混合物、含盐物质,仅保留有机化合物[18-20],使得可用于建模的数据量减少,导致模型应用域小。此外,已有研究大多是对某类化学品引起AD的实验性描述[9],但化学品结构与AD的内在关联仍不明确。机器学习算法可用于高阶、高维和非线性关系的数据分析,为构效关系和毒性机制理解提供有效手段[21-24]。
本研究基于文献和数据库挖掘,构建了涵盖4种临床不良症状(直立性低血压、失禁、尿失禁、肛门失禁)的数据集,包括466种阳性数据,427种阴性数据。基于该数据集,计算了ToxPrint毒性指纹,采用5种机器学习算法建立了AD化学品的筛查模型。ToxPrint毒性指纹可提供无机卤化键的信息,本研究将包含HCl、HBr盐类碎片的化学品也纳入建模数据集,扩大了模型应用域,所建立的模型不仅可用于有机化合物,还可用于含无机卤化键的化学品自主神经毒性数据填补。此外,本研究耦合SHAP (SHapley Additive exPlanations)方法和子结构片段频率分析方法,揭示了诱发AD的16种警示子结构,拓展了对AD机制的理解,以期为神经毒性化学品的筛查和评价提供参考。
1 材料与方法(Materials and methods)
1.1 数据集
从ADReCS数据库(http://bioinf.xmu.edu.cn/ADReCS/)不良反应层次结构树中的神经系统功能紊乱层次下,收集诱发AD(阳性)的化学品,涵盖直立性低血压、失禁、尿失禁和肛门失禁4种AD常见并发症[4,25]。本研究将Zhang等[26]研究中的非神经毒物作为不引发AD(阴性)的化学品,并通过与ADReCS数据库的匹配确认了该文献中报道的非神经毒物不是诱发AD的化学品。去除无机化合物、有机金属化合物、大分子盐类混合物,所收集的化学品共有893个。通过PubChem数据库和OpenBabel软件(3.1.1版本)获取化学品的2D结构,以sdf格式保存以备计算分子特征使用。
1.2 分子特征计算和选择
使用ChemoTyper软件(1.0版本, https://chemotyper.org/),通过“ToxPrint_v2.0_r212.xml”文件,计算得到729种ToxPrint毒性二进制指纹(https://ToxPrint.org/),包括原子(atom)、键(bond)、链(chain)、环(ring)和基团(group)五大类[27]。考虑到ToxPrint毒性指纹是专门为促进结构毒性关系建模而设计,便于毒性机理解释,因此基于该指纹建立模型。ToxPrint毒性指纹可提供无机卤化键信息,本研究将包含HCl、HBr盐类碎片的化学品纳入数据集,考虑了其对AD的影响。建模前,首先去除方差为0的毒性指纹,减少冗余的特征;其次过滤掉毒性指纹之间相关系数(r)> 0.9的特征,处理多重共线性特征;最后基于随机森林的递归特征消除法,选择与AD高度相关的毒性指纹,作为分子特征用于模型构建。
1.3 建模机器学习算法
将建模数据进行随机划分,其中80%为训练集,20%为验证集。训练集用于训练模型,测试集用于评估模型的性能。采用5种机器学习算法开发模型,包括决策树(decision tree, DT)分类[28]、支持向量机(support vector classification, SVC)分类[29]、k近邻(k-nearest neighbor, kNN)分类[30]、随机森林(random forest, RF)分类[31]、梯度提升决策树(gradient boosting decision tree, GBDT)分类[32]。DT、SVC和kNN是单一机器学习算法,RF和GBDT是基于决策树的集成学习算法。通过网格搜索确定机器学习算法的最佳超参数。模型构建基于Python语言实现,使用NumPy、Pandas和Scikit-learn库进行数据处理和机器学习,采用Matplotlib和Seaborn库进行数据可视化。
1.4 模型评价
采用十折交叉验证的方法进行内部验证,评价模型稳健性。使用验证集数据进行外部验证,评价模型的预测能力。采用6个指标评价模型的预测性能,包括准确率(accuracy, ACC)、敏感性(sensitivity , SE)、特异性(specificity, SP)、F1分数(F1-score, F1)、受试者工作特征曲线下面积(area under curve of receiver operating characteristic, AUC)和马修斯相关系数(Matthews correlation coefficient, MCC),各指标计算公式如下:
式中:TP是正确分类为阳性的类别;TN是正确分类为阴性的类别;FP是错误分类为阳性的类别;FN是错误分类为阴性的类别[33];精确率(precision, P)是预测正确的阳性样本占预测为阳性样本的比例。
ACC表示模型预测正确的结果占所有分类结果的比例,其取值范围为0~1,该值越大越好;SE是被模型预测正确的阳性样本占实际阳性样本的比例,也被称为召回率;SP是模型预测正确的阴性样本占实际阴性样本的比例;F1是P与召回率的调和平均,AUC常被用于评价模型是否具有区分能力,其取值范围为0.5~1,1表示模型具有完美区分能力,可完全区分2类样本,0.5表示模型没有区分能力;MCC是评估分类模型质量的综合性指标,考虑了真阳性、真阴性、假阳性、假阴性,其取值范围为-1~1,该值越大越好[34-35]。
此外,还进行了Y随机性检验,即随机调整化学品的毒性标签Y,分子指纹矩阵不变,建立模型,重复100次,得到基于随机数据建模的ACC。如果ACC越低,则证明原模型的非偶然性,反之,模型偶然相关[36]。
1.5 模型应用域表征
由于MACCS指纹能识别167种一般结构模式,比ToxPrint毒性指纹具有更好的泛化能力,因此通过计算化学品的MACCS指纹相似度来表征模型的应用域。基于化学品的MACCS指纹,计算验证集化学品A与训练集化学品B之间的谷本相似性(Tanimoto coefficient)[37],计算公式如下:
式中:SAB为化学品A和B之间的相似性;xjA为化学品A的第j个特征;xjB为化学品B的第j个特征;n为指纹的特征位数。本研究通过确定相似性阈值和最少相似分子数量来定义分类模型的应用域,最少相似分子数量设为1,如果化学品的相似性不低于预定义的相似阈值,则认为其在应用域内。
1.6 警示子结构识别
警示子结构是诱发毒性效应的毒效基团或有毒碎片,包括子结构(例如,羟基、氨基、苯基等)或者这些子结构的组合[38]。本研究耦合SHAP方法和子结构片段频率分析,揭示诱发AD的警示子结构。
SHAP(https://shap.readthedocs.io/en/latest/index.html)是一种博弈论方法,它可以为每个特征分配一个特定预测的重要性值,即SHAP值,并将其作为特征重要性的度量[39]。一个毒性指纹的SHAP值(正或负)越大,意味着该毒性指纹对模型输出的影响越大。不同的颜色表示毒性指纹的大小,红色点表示存在该子结构(1),蓝色点表示不存在该子结构(0)。如果红色点在右侧(具有正的SHAP值),则该子结构的存在对AD有正向的贡献,反之亦然。
子结构片段频率分析法已被广泛用于获取警示子结构[19,40]。如果某子结构在阳性化学品中出现的频率比阴性化学品中出现的频率更高,该子结构就可以被认为是易于诱发AD的特征子结构,需要警惕。AD化学品中,片段出现的频率定义为如下公式计算:
频率![]()
式中:N子结构类别表示在阳性化学品中包含此片段的总数目;N总数表示数据集中所有化学品数目;N子结构总数表示包含该子结构片段的所有化学品数目;N类别表示数据集中阳性化学品的数目。
2 结果(Results)
2.1 数据集建立与分子特征选择
本研究建立了化学品AD的数据集,该数据集包含466种阳性数据,427种阴性数据。数据集涵盖AD的常见并发症,包括直立性低血压、失禁、尿失禁和肛门失禁。采用ChemoTyper软件计算得到了729种ToxPrint毒性指纹,包括7种原子、414种键、95种链、144种环、69种基团。经过方差过滤,保留了385个指纹特征。经过皮尔逊相关系数过滤,保留了326个指纹特征。基于随机森林的递归特征消除法,筛选出了120种毒性指纹作为模型输入,涵盖61种键、25种链、29种环和5种基团结构。如图1所示,当特征数目>120个,模型随着毒性指纹数量的增加,十折交叉验证性能并无显著提升。表1列出了基于特征重要性排序的前20种毒性指纹。
表1 筛选出的前20种毒性指纹
Table 1 Top 20 ToxPrints obtained by feature screening

排序Ranking毒性指纹ToxPrints毒性指纹释义Definition of ToxPrints1bond: CN_amine_sec-NH_generic键:CN_胺_仲-NH_通用2bond: CN_amine_aliphatic_generic键:CN_胺_脂肪族_通用3bond: X[any_!C]_halide_inorganic键:X[any_!C]_卤化物_无机4bond: C(=O)N_carboxamide_generic键:C(=O)N_酰胺_通用5bond: X[any]_halide键:X[any]_卤化物6chain: alkaneBranch_neopentyl_C5链:烷烃支链_新戊基_C57bond: CN_amine_ter-N_aliphatic键:CN_胺_三氮_脂肪族8bond: CN_amine_alicyclic_generic键:CN_胺_脂环族_通用9bond: CN_amine_sec-NH_alkyl键:CN_胺_仲-NH_烷基10chain: alkaneLinear_propyl_C3链:线性烷烃_丙基_C311bond: C(=O)N_carboxamide_(NR2)键:C(=O)N_酰胺_(NR2)12group: aminoAcid_aminoAcid_generic基团:氨基酸_氨基酸_通用13ring: hetero_[5]_Z_1-Z环:杂_[5]_Z_1-Z14ring: hetero_[5_6]_Z_generic环:杂_[5_6]_Z_通用15bond: S(=O)N_sulfonamide键:S(=O)N_磺胺16chain: aromaticAlkane_Ph-C1_cyclic链:芳香胺_Ph-C1_环17chain: alkaneCyclic_hexyl_C6链:环烷烃_己基_C618ring: hetero_[5]_N_pyrrole_generic环:杂_[5]_N_吡咯_通用19chain: alkaneLinear_ethyl_C2(H_gt_1)链:线性烷烃_乙基_C2(H_gt_1)20bond: C=O_carbonyl_generic键:C=O_羰基_通用
注:各毒性指纹的释义在使用手册和推荐文献中可查询(https://ToxPrint.org/)。
Note: Definition of ToxPrints can be found in the user manual and recommended literature (https://ToxPrint.org/).
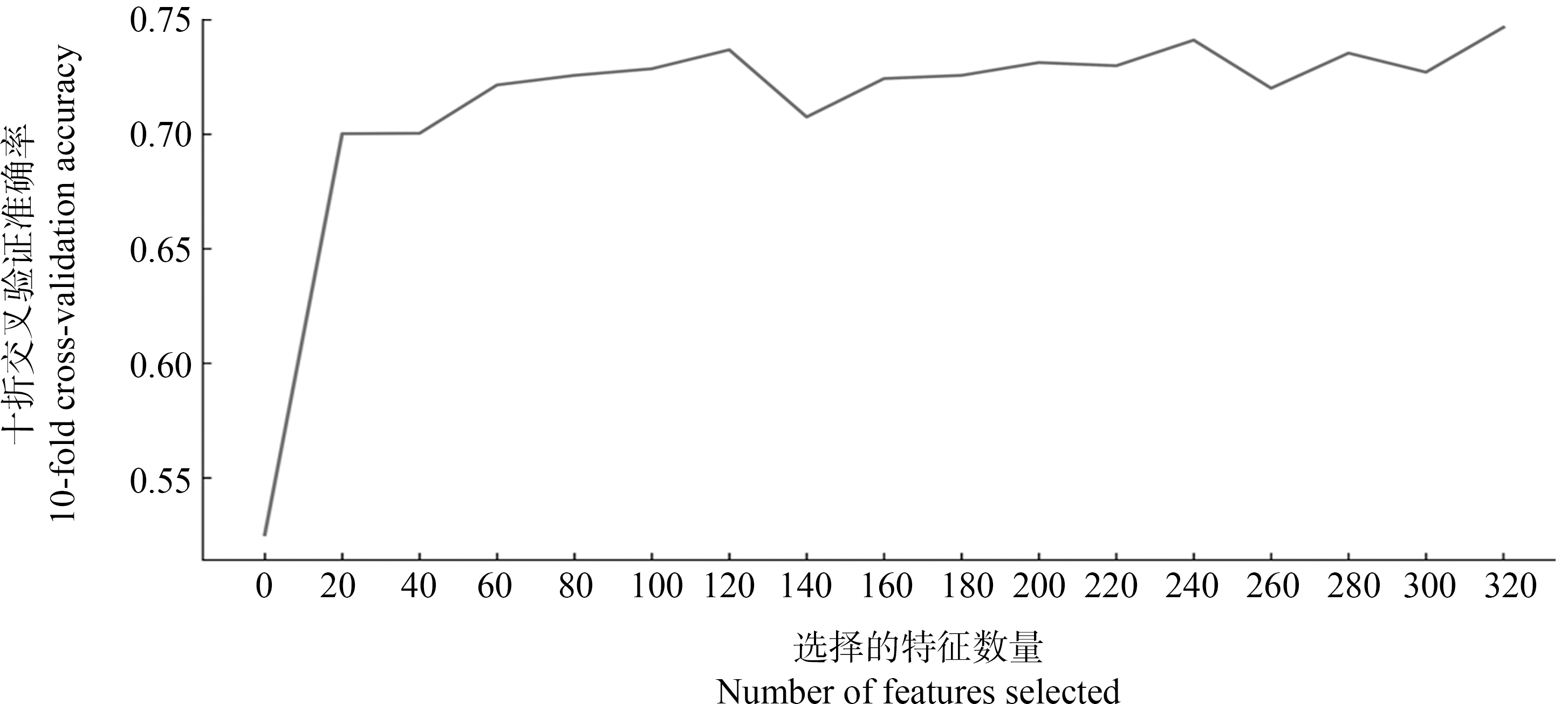
图1 基于随机森林的递归特征消除特征选择
Fig. 1 Feature selection plot of the recursive feature elimination incorporated with random forest
2.2 模型构建
本研究采用了5种机器学习算法开发数据驱动的AD化学品筛查模型,各模型的超参数如表2所示。模型的性能如表3所示,整体而言,2种基于树的集成模型(RF、GBDT)比3种经典单一模型(DT、kNN、SVC)具有更好的稳健性和预测能力。综合考虑训练集和验证集的6种评价指标,随机森林模型的分类性能最佳,训练集准确率达0.738,验证集准确率达0.737。对随机森林模型进行Y随机性检验(100次),训练集和验证集准确率约为0.5,表明随机森林模型非偶然相关。
表2 5种机器学习模型的超参数
Table 2 Hyperparameters for five machine learning models

模型 Model超参数 HyperparametersDT决策树最大深度(max_depth) = 5, 叶子节点最小样本数(min_samples_leaf) = 15, 分割所需最小样本数(min_samples_split) = 5kNN邻近点数(n_neighbors) = 1SVC正则化参数(C) = 100, 核系数(gamma) = 0.01RF决策树的数目(n_estimators) = 260, 随机种子(random_state) = 86, 决策树最大深度(max_depth) = 19GBDT随机种子(random_state) = 290, 决策树的数目(n_estimators) = 68
注:DT为决策树分类、kNN为k近邻分类、SVC为支持向量机分类、RF为随机森林分类、GBDT为梯度提升决策树分类。
Note: DT represents decision tree, SVC represents support vector classification, kNN represents k-nearest neighbor, RF represents random forest, and GBDT represents gradient boosting decision tree.
表3 分类模型的预测性能
Table 3 Prediction performance of classification models
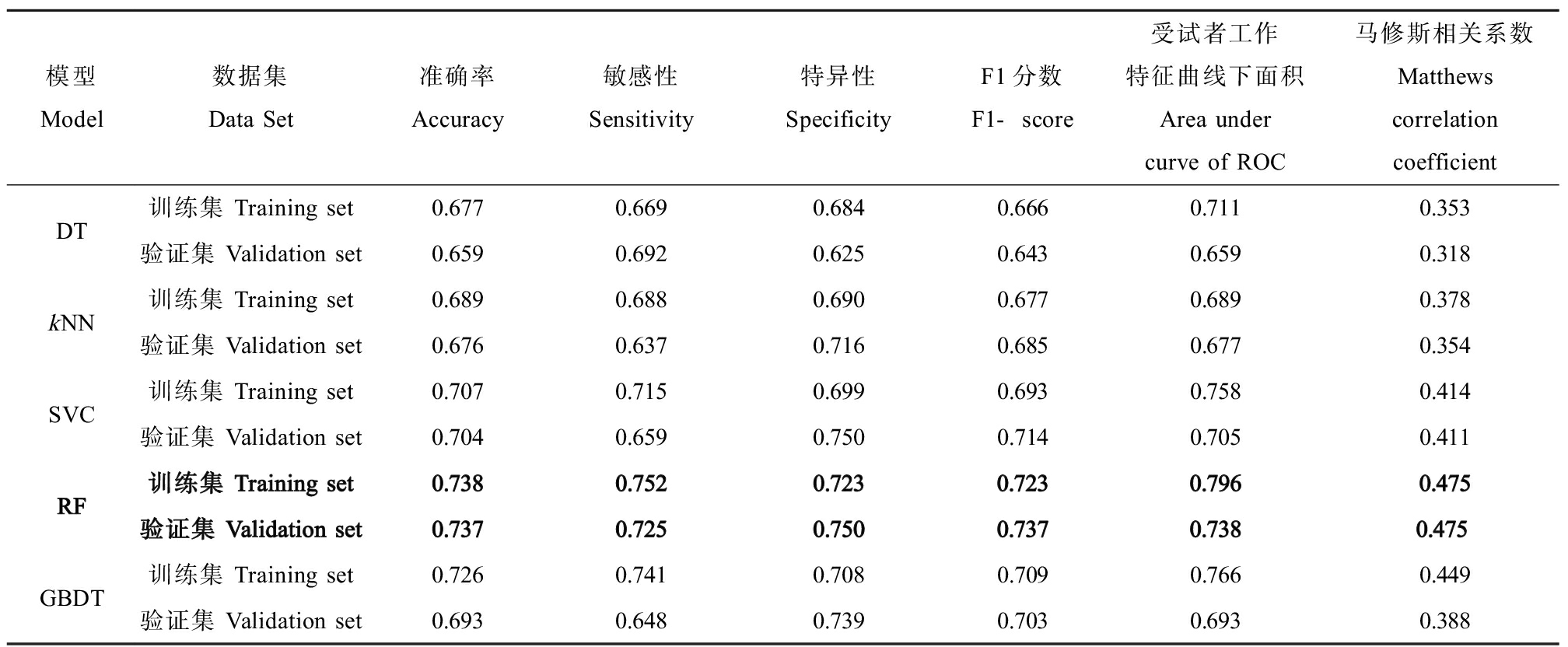
模型Model数据集Data Set准确率Accuracy敏感性Sensitivity特异性SpecificityF1分数F1-score受试者工作特征曲线下面积Area under curve of ROC马修斯相关系数Matthews correlation coefficientDT训练集 Training set0.6770.6690.6840.6660.7110.353验证集 Validation set0.6590.6920.6250.6430.6590.318kNN训练集 Training set0.6890.6880.6900.6770.6890.378验证集 Validation set0.6760.6370.7160.6850.6770.354SVC训练集 Training set0.7070.7150.6990.6930.7580.414验证集 Validation set0.7040.6590.7500.7140.7050.411RF训练集 Training set0.7380.7520.7230.7230.7960.475验证集 Validation set0.7370.7250.7500.7370.7380.475GBDT训练集 Training set0.7260.7410.7080.7090.7660.449验证集 Validation set0.6930.6480.7390.7030.6930.388
注:DT为决策树分类、kNN为k近邻分类、SVC为支持向量机分类、RF为随机森林分类、GBDT为梯度提升决策树分类、ROC为受试者工作特征曲线。
Note: DT represents decision tree, SVC represents support vector classification, kNN represents k-nearest neighbor, RF represents random forest, GBDT represents gradient boosting decision tree, and ROC is receiver operating characteristic.
2.3 应用域表征
本研究基于MACCS指纹的相似度,表征随机森林分类模型的应用域。整体来说,如表4所示,随着相似性阈值的增加(0.55~0.95),模型分类准确率增加(0.740~0.941),所包含的验证集化学品数量减少(173~17)。当相似性阈值设置为0.65时,验证集中81.56%的化学品位于应用域内,模型预测准确率达0.747。相似性阈值为0.75时,验证集中60.89%的化学品位于应用域内,此时模型预测准确率达0.752。
表4 随机森林模型的应用域
Table 4 Application domain of the random forest model
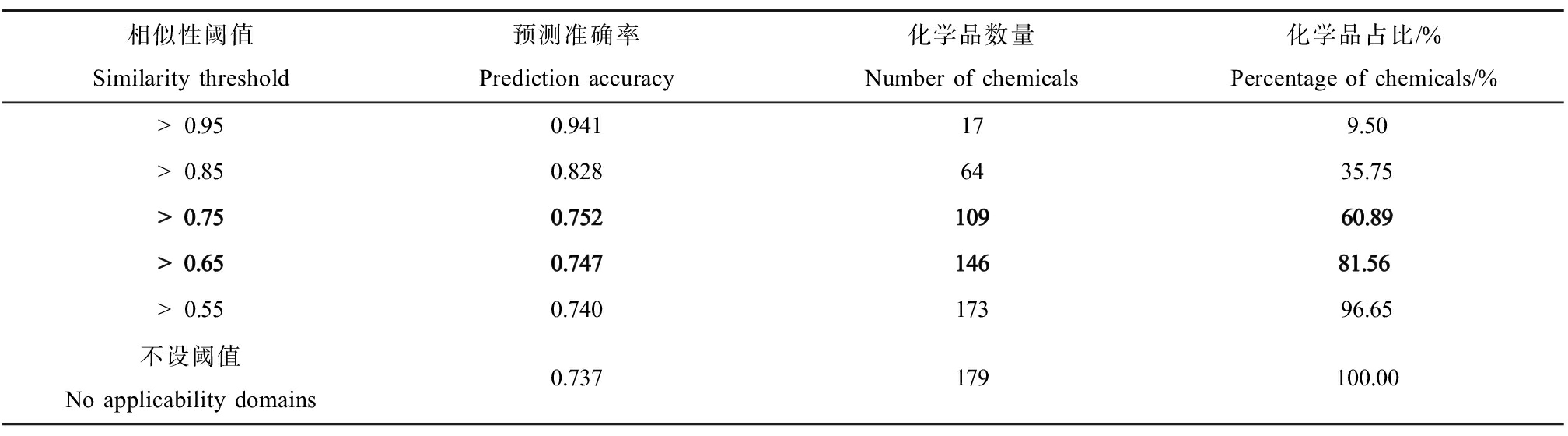
相似性阈值Similarity threshold预测准确率Prediction accuracy化学品数量Number of chemicals化学品占比/%Percentage of chemicals/%>0.950.941179.50>0.850.8286435.75>0.750.75210960.89>0.650.74714681.56>0.550.74017396.65不设阈值 No applicability domains0.737179100.00
2.4 警示子结构识别
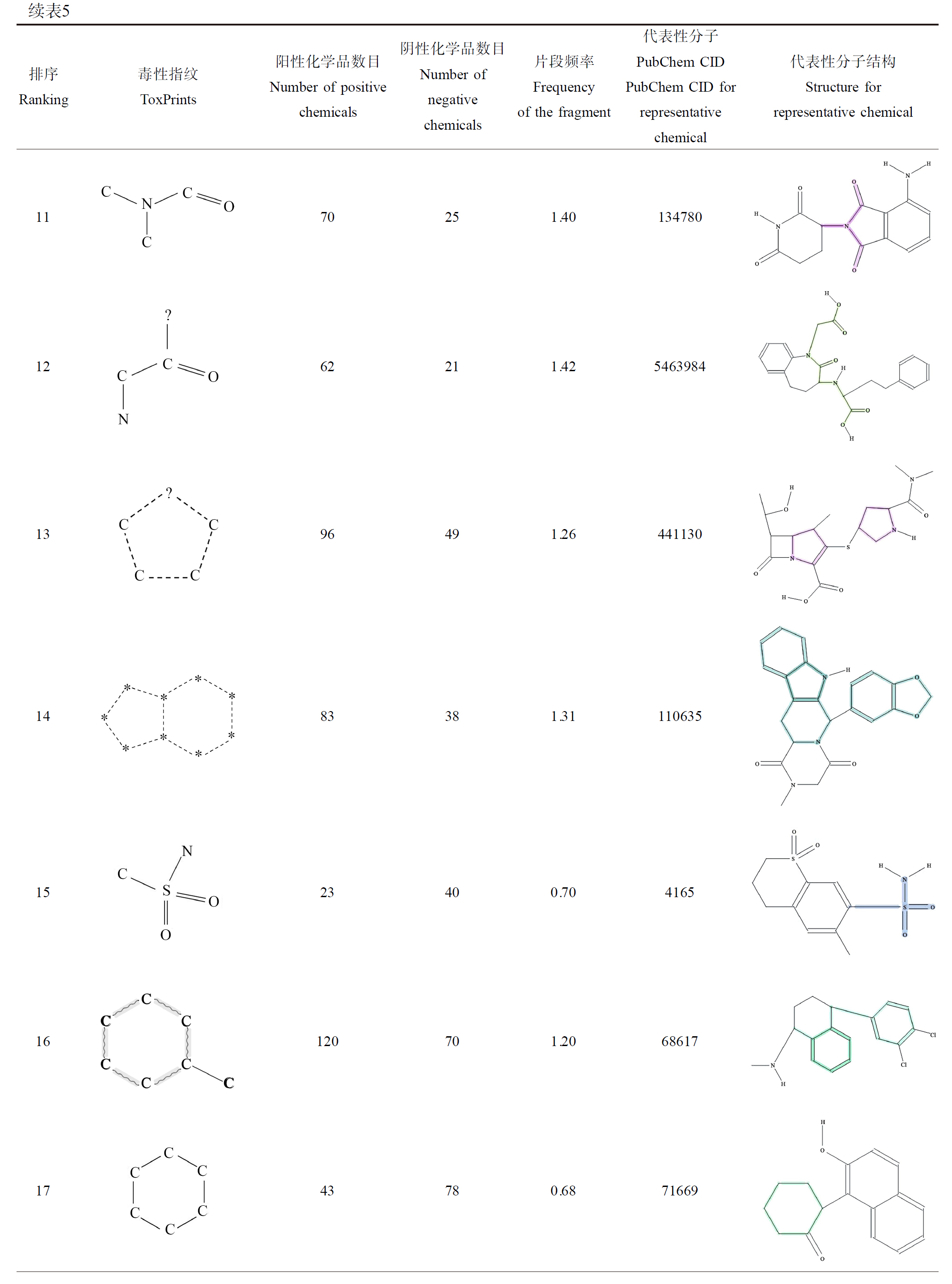
本研究计算了随机森林模型分子特征的SHAP值,对前20个影响自主神经功能的结构片段进行重要性排序,结果见图2(a)。前20个毒性指纹的SHAP值高低(红色或蓝色)与自主神经毒性的关系,结果见图2(b)。在20个子结构中,16个子结构的红色点绝大部分处于SHAP值>0的一侧,蓝色点处于SHAP值<0的一侧。表明以下毒性指纹在分子结构中存在,会导致AD,包括9种键结构:CN_胺_仲-NH_通用、CN_胺_脂肪族_通用、X[any_!C]_卤化物_无机、C(=O)N_酰胺_通用、X[any]_卤化物、CN_胺_三氮_脂肪族、CN_胺_脂环族_通用、CN_胺_仲-NH_烷基、C(=O)N_酰胺_(NR2),3种链结构:线性烷烃_丙基_C3、芳香胺_Ph-C1_环、线性烷烃_乙基_C2(H_gt_1),3种环结构:杂_[5]_Z_1-Z、杂_[5_6]_Z_通用、杂_[5]_N_吡咯_通用,以及1种基团结构:氨基酸_氨基酸_通用。本研究进一步采用子结构片段频率分析法对上述结构片段进行频率计算,发现其频率均>1(表5)。
表5 毒性指纹重要性排序及其频率
Table 5 ToxPrints importance ranking and its frequency
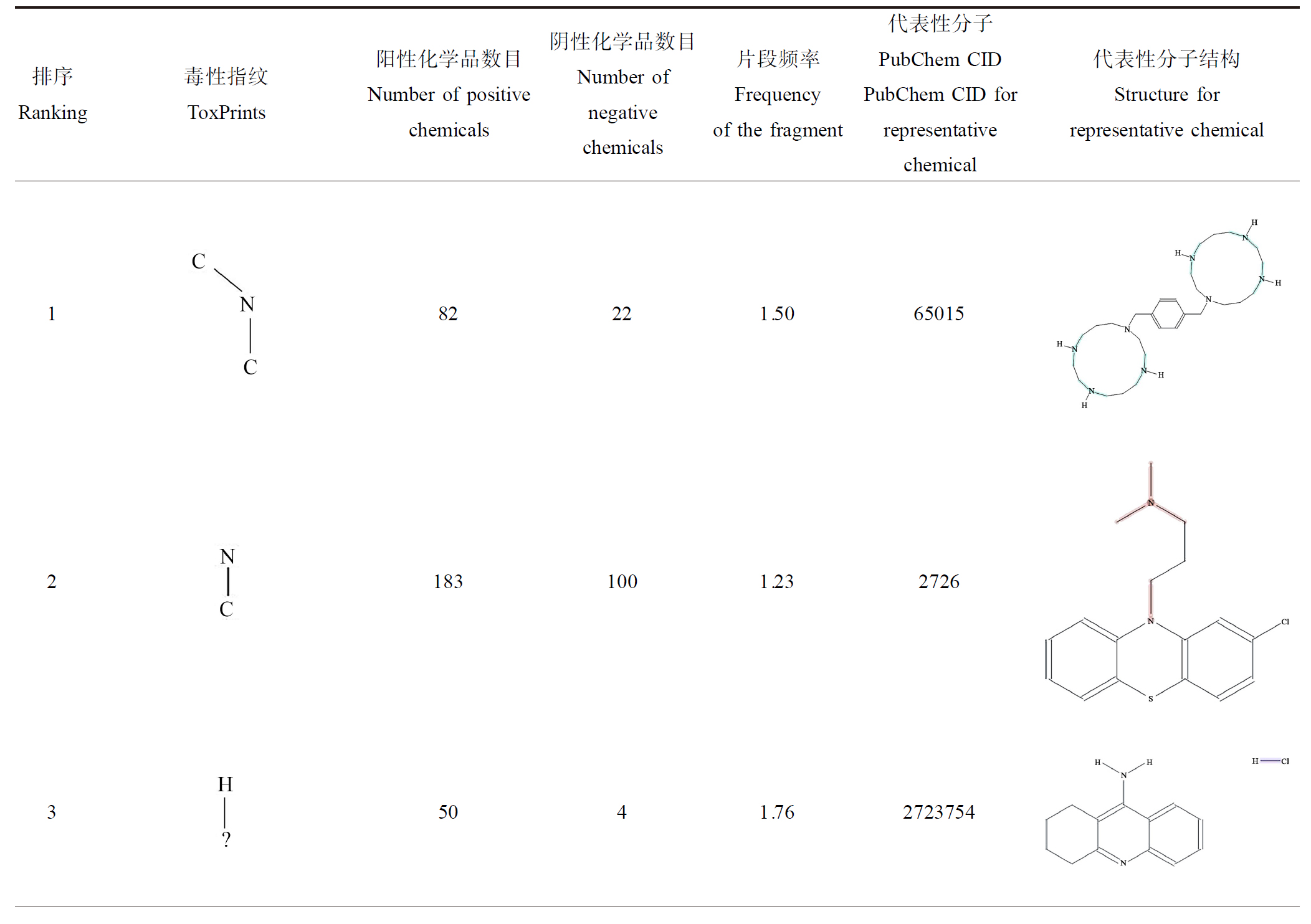
排序Ranking毒性指纹ToxPrints阳性化学品数目Number of positive chemicals阴性化学品数目Number of negative chemicals片段频率Frequency of the fragment代表性分子PubChem CID PubChem CID for representative chemical代表性分子结构Structure for representative chemical182221.50 6501521831001.23 272635041.76 27237544167991.20 52845835151901.19 216239640840.61 4912

续表5排序Ranking毒性指纹ToxPrints阳性化学品数目Number of positive chemicals阴性化学品数目Number of negative chemicals片段频率Frequency of the fragment代表性分子PubChem CID PubChem CID for representative chemical代表性分子结构Structure for representative chemical7132711.24 119788138122571.30 2678968181.51 236610121711.20 98527461170251.40 1347801262211.42 54639841396491.26 441130
续表5排序Ranking毒性指纹ToxPrints阳性化学品数目Number of positive chemicals阴性化学品数目Number of negative chemicals片段频率Frequency of the fragment代表性分子PubChem CID PubChem CID for representative chemical代表性分子结构Structure for representative chemical1483381.31 110635 1523400.70 416516120701.20 686171743780.68 716691858211.40 5329102191731261.10 49831411
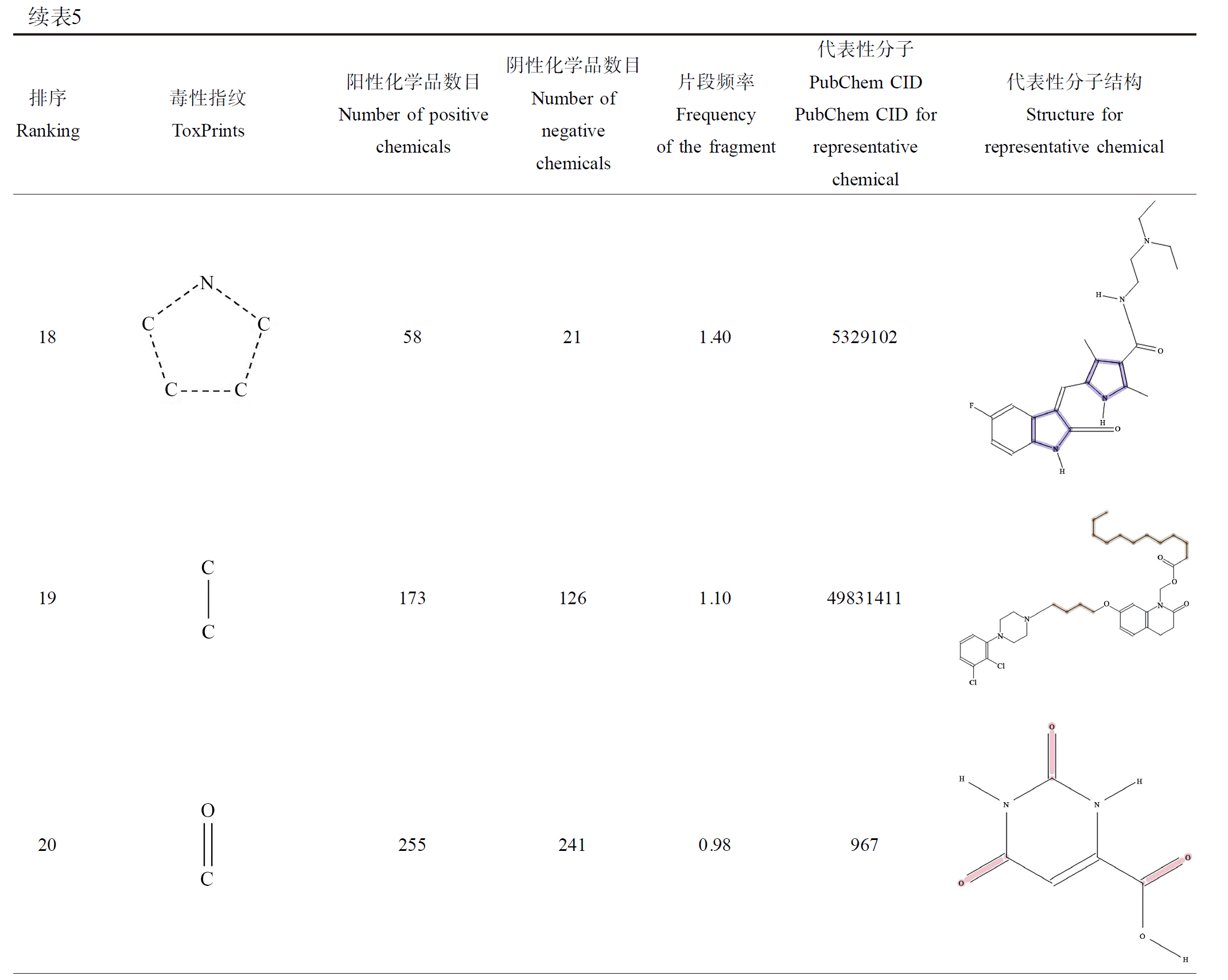
续表5排序Ranking毒性指纹ToxPrints阳性化学品数目Number of positive chemicals阴性化学品数目Number of negative chemicals片段频率Frequency of the fragment代表性分子PubChem CID PubChem CID for representative chemical代表性分子结构Structure for representative chemical202552410.98 967
注:毒性指纹中为“芳香族”的原子和化学键用灰色标记;“?”为通配符用于任何字符,“*”用于未定义的字符序列;片段出现的频率>1时列出代表性阳性分子的PubChem CID及其结构;片段出现的频率<1时列出代表性阴性分子的PubChem CID及其结构;代表性分子结构中的颜色标注表示对应的毒性指纹。
Note: Atoms and bonds in ToxPrints that are flagged as “aromatic” are marked by the grey; the wild card characters “?” for any character and “*” for an undefined sequence of characters; when the frequency of a fragment is greater than 1, PubChem CIDs and structures for those representative chemicals that indicate toxicity are listed; when the frequency of a fragment is less than 1, PubChem CIDs and structures for those representative chemicals that indicate non-toxicity are listed; color annotations in structures for representative chemicals represent corresponding toxicity fingerprints.

图2 训练集前20个特征的平均绝对SHAP值(a)和单个SHAP值(b)
Fig. 2 Mean absolute SHAP values (a) and individual SHAP values (b) for TOP 20 features of the training set
3 讨论(Discussion)
3.1 模型性能和应用域分析
本研究比较了5种机器学习算法,相比于DT模型,RF模型没有出现过拟合,训练集和验证集性能不存在很大的差距。RF算法通过从数据集中随机选择单个DT来控制模型过拟合,减少方差,因此通常RF模型比DT模型性能高。GBDT算法基于回归树,在相对少的调参情况下,获得了较好的预测效果。SVM算法比较适用于小样本情况下的预测,而且对参数和核函数的选择比较敏感[41],SVM模型性能略逊色于随机森林模型。kNN算法在样本不平衡的情况下,训练容易偏向训练样本中数量占优的类别,导致错误预测[42]。本研究由于样本相对平衡,kNN算法的表现也较好。一般来说,DT模型可解释性较好,但是准确率通常不如其他模型,其在所构建的5个模型中准确率最低,但模型性能仍是可接受的(>0.65)[43]。
在传统的筛查模型构建时,数据集一般需要去除无机物、混合物、含盐物质,仅保留有机化合物[18-20],考虑到ToxPrint毒性指纹可提供无机卤化键的信息,本研究将包含HCl、HBr盐类碎片的化学品纳入数据集,扩大了模型应用域。数据集中的893种化学品包括有机酸、醚、酯、酮、醇、酰胺、苯胺、多环芳烃及其取代物、卤代烷烃、卤代烯烃、杂环化合物及其衍生物等。将该数据集的阳性化学品与美国环境保护局的室内环境物质清单(https://comptox.epa.gov/dashboard/chemical-lists/INDOORCT16)比对发现,其中27种阳性化学品是室内环境污染相关化学品(西酞普兰、二甲双胍、喹硫平等),它们多为抗精神病、抗抑郁、抗癌、抗炎、抗菌、治疗糖尿病、治疗心血管疾病和止痛类的药物和动物饲料添加剂。Writer等[44]报道了西酞普兰在环境相关的暴露浓度下引发神经毒性。
本研究使用人类临床不良反应AD数据,而非动物数据开发模型,可以作为化学品诱发人体自主神经毒性筛查的有效工具。当然,本研究构建的模型也存在一些局限性,ToxPrint是专家依据经验设计的毒性指纹,其只能定性分子结构中是否存在某种子结构,却不能进行定量,即知晓分子结构中存在某种子结构的个数。后续通过更优的分子特征表示或者结合体外细胞数据和高通量数据探索化学品诱发的AD,模型性能有希望进一步提高。
3.2 模型机理解释和警示子结构
随机森林模型所识别的前20个关键的结构中,35%包含CN键,即CN_胺_仲-NH_通用、CN_胺_脂肪族_通用、C(=O)N_酰胺_通用、CN_胺_三氮_脂肪族、CN_胺_脂环族_通用、CN_胺_仲-NH_烷基、C(=O)N_酰胺_(NR2)。这可能是由于氮是高电负性原子,其存在通常会增强化学品的毒性作用,特别是大分子碳骨架中CN片段的存在可能有助于保持化学品的亲脂特性[45],脂溶性高的化学品更容易穿过血-神经屏障而作用于自主神经系统。Liu等[46]使用体外生物活性数据和化学结构预测器官毒性,也发现键:C(=O)N_酰胺_通用、键:C(=O)N_酰胺_(NR2)、键:CN_胺_脂肪族_通用和键:CN_胺_三氮_脂肪族与大脑毒性具有很强的相关性,与本研究的结果一致。
本研究所构建的模型识别出的16种警示子结构中,涵盖3种杂环结构[杂_[5]_Z_1-Z、杂_[5_6]_Z_通用、杂_[5]_N_吡咯_通用]诱发AD。Zhao等[18]的研究也发现了杂环([5_6]_Z)是药物诱导神经毒性的警示结构片段。Xu等[47]的结果表明杂_[5]_N_吡咯_通用是对大脑产生毒性的重要化学结构特征。本研究除了发现这2种杂环外,还发现了杂_[5]_Z_1-Z结构,是影响自主神经毒性的重要结构片段。此外,本研究发现卤化物键[X[any_!C]_卤化物_无机、X[any]_卤化物]的存在也能诱发AD,这可能是由于卤(例如氯)原子的存在可增强化学品的亲脂性,从而增加毒性[45]。
子结构片段频率分析与SHAP分析结果具有良好的一致性,上述结构碎片的频率分析结果均>1,即在阳性化学品中出现的频率比阴性化学品中出现的频率更高,进一步证实了这些结构易于诱发AD。含有不同子结构的化学品在能否诱发AD上有显著的区别,本研究通过警示子结构进行了初步探索,可为化学品AD的作用机制提供参考,但该结果还需实验进一步验证。
综上,本研究基于ToxPrint毒性指纹,采用5种机器学习算法开发了AD化学品的筛查模型,使用十折交叉验证和Y随机性检验的方法验证了模型的稳健性和可靠性。随机森林模型具有最优的分类性能,其验证集准确率达0.737。当模型应用域的相似性阈值为0.75时,验证集准确率提高至0.752。本研究还耦合SHAP方法和子结构片段频率分析方法,揭示了易于诱发AD的16种警示子结构,包括碳氮键[CN_胺_仲-NH_通用、CN_胺_脂肪族_通用、C(=O)N_酰胺_通用、CN_胺_三氮_脂肪族、CN_胺_脂环族_通用、CN_胺_仲-NH_烷基、C(=O)N_酰胺_(NR2)]、卤化物键[X[any_!C]_卤化物_无机、X[any]_卤化物]、杂环[杂_[5]_Z_1-Z、杂_[5_6]_Z_通用、杂_[5]_N_吡咯_通用]等结构,拓展了对AD机制的理解,对神经毒性化学品的人体健康风险评价具有指导意义。
[1] Cardinali D P. Clinical Implications of the Enlarged Autonomic Nervous System[M]//Autonomic Nervous System. Cham: Springer International Publishing, 2017: 287-312
[2] 庄志雄. 靶器官毒理学[M]. 北京: 化学工业出版社, 2006: 163-171
[3] 赵超英, 姜允申. 神经系统毒理学[M]. 北京: 北京大学医学出版社, 2009: 91-136
[4] Jain K K. Drug-induced Disorders of the Autonomic Nervous System[M]// Drug-induced Neurological Disorders. Cham: Springer, 2021: 469-479
[5] Herring N, Kalla M, Paterson D J. The autonomic nervous system and cardiac arrhythmias: Current concepts and emerging therapies [J]. Nature Reviews Cardiology, 2019, 16(12): 707-726
[6] Ehmke H. The mechanotransduction of blood pressure [J]. Science, 2018, 362(6413): 398-399
[7] Shewale S V, Anstadt M P, Horenziak M, et al. Sarin causes autonomic imbalance and cardiomyopathy: An important issue for military and civilian health [J]. Journal of Cardiovascular Pharmacology, 2012, 60(1): 76-87
[8] Nguyen L S, Cooper L T, Kerneis M, et al. Systematic analysis of drug-associated myocarditis reported in the World Health Organization pharmacovigilance database [J]. Nature Communications, 2022, 13(1): 25
[9] Leung J Y T, Barr A M, Procyshyn R M, et al. Cardiovascular side-effects of antipsychotic drugs: The role of the autonomic nervous system [J]. Pharmacology &Therapeutics, 2012, 135(2): 113-122
[10] 周宗灿. 毒理学教程[M]. 3版. 北京: 北京大学医学出版社, 2006: 486-502
[11] Cheshire W P, Freeman R, Gibbons C H, et al. Electrodiagnostic assessment of the autonomic nervous system: A consensus statement endorsed by the American Autonomic Society, American Academy of Neurology, and the International Federation of Clinical Neurophysiology [J]. Clinical Neurophysiology, 2021, 132(2): 666-682
[12] Freeman R, Wieling W, Axelrod F B, et al. Consensus statement on the definition of orthostatic hypotension, neurally mediated syncope and the postural tachycardia syndrome [J]. Clinical Autonomic Research, 2011, 21(2): 69-72
[13] 邓东阳, 于红霞, 张效伟, 等. 基于毒性效应的非目标化学品鉴别技术进展[J]. 生态毒理学报, 2015, 10(2): 13-25
Deng D Y, Yu H X, Zhang X W, et al. Development and application of nontargeted analysis in effect directed analysis [J]. Asian Journal of Ecotoxicology, 2015, 10(2): 13-25(in Chinese)
[14] Wang Z Y, Walker G W, Muir D C G, et al. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories [J]. Environmental Science &Technology, 2020, 54(5): 2575-2584
[15] Johnson A C, Jin X W, Nakada N, et al. Learning from the past and considering the future of chemicals in the environment [J]. Science, 2020, 367(6476): 384-387
[16] Kasahara Y, Yoshida C, Nakanishi K, et al. Alterations in the autonomic nerve activities of prenatal autism model mice treated with valproic acid at different developmental stages [J]. Scientific Reports, 2020, 10: 17722
[17] Pognan F, Beilmann M, Boonen H C M, et al. The evolving role of investigative toxicology in the pharmaceutical industry [J]. Nature Reviews Drug Discovery, 2023, 22(4): 317-335
[18] Zhao X, Sun Y H, Zhang R Q, et al. Machine learning modeling and insights into the structural characteristics of drug-induced neurotoxicity [J]. Journal of Chemical Information and Modeling, 2022, 62(23): 6035-6045
[19] Wang Z Y, Zhao P P, Zhang X X, et al. In silico prediction of chemical respiratory toxicity via machine learning [J]. Computational Toxicology, 2021, 18: 100155
[20] Tang W H, Chen J W, Hong H X. Development of classification models for predicting inhibition of mitochondrial fusion and fission using machine learning methods [J]. Chemosphere, 2021, 273: 128567
[21] Crofton K M, Bassan A, Behl M, et al. Current status and future directions for a neurotoxicity hazard assessment framework that integrates in silico approaches [J]. Computational Toxicology, 2022, 22: 100223
[22] Jeong J, Choi J. Artificial intelligence-based toxicity prediction of environmental chemicals: Future directions for chemical management applications [J]. Environmental Science &Technology, 2022, 56(12): 7532-7543
[23] 滕跃发, 王晓晴, 李斐, 等. 大数据挖掘和机器学习在毒理学中的应用[J]. 生态毒理学报, 2022, 17(1): 93-101
Teng Y F, Wang X Q, Li F, et al. Application of data mining and machine learning in toxicology [J]. Asian Journal of Ecotoxicology, 2022, 17(1): 93-101 (in Chinese)
[24] 张家晨, 张良, 庄树林. 分子起始事件在计算毒理学中的研究展望[J]. 环境化学, 2021, 40(9): 2629-2632
Zhang J C, Zhang L, Zhuang S L. Perspective of molecular initiating events in computational toxicology [J]. Environmental Chemistry, 2021, 40(9): 2629-2632 (in Chinese)
[25] Garland E M, Robertson D. Autonomic Failure [M]//Encyclopedia of Neuroscience. Amsterdam: Elsevier, 2009: 825-832
[26] Zhang H, Mao J, Qi H Z, et al. Developing novel computational prediction models for assessing chemical-induced neurotoxicity using naïve Bayes classifier technique [J]. Food and Chemical Toxicology, 2020, 143: 111513
[27] Yang C, Tarkhov A, Marusczyk J, et al. New publicly available chemical query language, CSRML, to support chemotype representations for application to data mining and modeling [J]. Journal of Chemical Information and Modeling, 2015, 55(3): 510-528
[28] Nguyen T N, Nakanowatari S, Nhat Tran T P, et al. Learning catalyst design based on bias-free data set for oxidative coupling of methane [J]. ACS Catalysis, 2021, 11(3): 1797-1809
[29] Specht T, Münnemann K, Hasse H, et al. Automated methods for identification and quantification of structural groups from nuclear magnetic resonance spectra using support vector classification [J]. Journal of Chemical Information and Modeling, 2021, 61(1): 143-155
[30] Sarkar N, Gupta R, Keserwani P K, et al. Air Quality Index prediction using an effective hybrid deep learning model [J]. Environmental Pollution, 2022, 315: 120404
[31] Cheng W X, Ng C A. Using machine learning to classify bioactivity for 3486 per- and polyfluoroalkyl substances (PFASs) from the OECD list [J]. Environmental Science &Technology, 2019, 53(23): 13970-13980
[32] Zulfiqar H, Yuan S S, Huang Q L, et al. Identification of cyclin protein using gradient boost decision tree algorithm [J]. Computational and Structural Biotechnology Journal, 2021, 19: 4123-4131
[33] 王园宁, 刘会会, 杨先海. 构建有机化合物斑马鱼雌激素干扰效应的二元分类模型[J]. 生态毒理学报, 2019, 14(4): 163-169
Wang Y N, Liu H H, Yang X H. Development of binary classification models for predicting estrogenic activity of organic compounds on zebrafish [J]. Asian Journal of Ecotoxicology, 2019, 14(4): 163-169 (in Chinese)
[34] Huang Y, Li X H, Xu S J, et al. Quantitative structure-activity relationship models for predicting inflammatory potential of metal oxide nanoparticles [J]. Environmental Health Perspectives, 2020, 128(6): 67010
[35] Huang Y, Li X H, Cao J Y, et al. Use of dissociation degree in lysosomes to predict metal oxide nanoparticle toxicity in immune cells: Machine learning boosts nano-safety assessment [J]. Environment International, 2022, 164: 107258
[36] 陈景文, 全燮. 环境化学[M]. 大连: 大连理工大学出版社, 2009: 260-289
[37] Wang Z Y, Chen J W, Hong H X. Applicability domains enhance application of PPARγ agonist classifiers trained by drug-like compounds to environmental chemicals [J]. Chemical Research in Toxicology, 2020, 33(6): 1382-1388
[38] Yang H B, Lou C F, Li W H, et al. Computational approaches to identify structural alerts and their applications in environmental toxicology and drug discovery [J]. Chemical Research in Toxicology, 2020, 33(6): 1312-1322
[39] Lundberg S M, Lee S I. A unified approach to interpreting model predictions [J]. Advances in Neural Information Processing Systems, 2017, 30: 4765-4774
[40] 孙露, 陈英杰, 吴曾睿, 等. 有机化合物生物富集因子的计算机预测研究[J]. 生态毒理学报, 2015, 10(2): 173-182
Sun L, Chen Y J, Wu Z R, et al. In silico prediction of chemical bioconcentration factor [J]. Asian Journal of Ecotoxicology, 2015, 10(2): 173-182 (in Chinese)
[41] Hochrein J, Klein M S, Zacharias H U, et al. Performance evaluation of algorithms for the classification of metabolic 1H NMR fingerprints [J]. Journal of Proteome Research, 2012, 11(12): 6242-6251
[42] Liu W P, Zhang L R, Bao L J, et al. Accurate classification and prediction of acute myocardial infarction through an ARMD procedure [J]. Journal of Proteome Research, 2023, 22(3): 758-767
[43] Jain S, Norinder U, Escher S E, et al. Combining in vivo data with in silico predictions for modeling hepatic steatosis by using stratified bagging and conformal prediction [J]. Chemical Research in Toxicology, 2021, 34(2): 656-668
[44] Writer J H, Antweiler R C, Ferrer I, et al. In-stream attenuation of neuro-active pharmaceuticals and their metabolites [J]. Environmental Science &Technology, 2013, 47(17): 9781-9790
[45] Mukherjee R K, Kumar V, Roy K. Ecotoxicological QSTR and QSTTR modeling for the prediction of acute oral toxicity of pesticides against multiple avian species [J]. Environmental Science &Technology, 2022, 56(1): 335-348
[46] Liu J, Patlewicz G, Williams A J, et al. Predicting organ toxicity using in vitro bioactivity data and chemical structure [J]. Chemical Research in Toxicology, 2017, 30(11): 2046-2059
[47] Xu T, Ngan D K, Ye L, et al. Predictive models for human organ toxicity based on in vitro bioactivity data and chemical structure [J]. Chemical Research in Toxicology, 2020, 33(3): 731-741