鱼类多样性是反映水生态健康的重要指标,鱼类生物多样性监测通常采用网具拦截法、电鱼法、网具拖拽捕获和固定式诱捕等方法采集鱼类个体。肉眼观察计数,同时采用传统形态学鉴定方法识别鱼类物种。但是该方法效率低、耗费人力物力,且具有极大的偶然性、对生态系统具有一定的破坏。近年来,环境DNA(environmental DNA, eDNA)宏条形码监测技术的发展,为鱼类多样性资源调查与监测提供了新方向。
eDNA技术是通过从环境介质中提取DNA,对基因组的特定DNA片段进行PCR扩增和高通量测序,从而实现对生物群落的监测[1-3]。例如,舒璐等[4]利用eDNA技术对鱼类多样性进行了研究,结果显示eDNA宏条形码技术可以快速检测鱼类多样性及其空间分布,克服了传统方法的局限性。Evans等[5]利用206 L的中型生态系统的研究证明了eDNA方法研究鱼类和两栖类的多样性及生物量的可行性。我国国内越来越多的研究学者也陆续开展基于eDNA技术的生物多样性研究工作,包括淮河流域、长江流域等全国各河流湖泊[6-9]。
太湖是我国第三大淡水湖泊,渔业资源十分丰富,太湖鱼类志共记载太湖鱼类107种。近年来,对太湖鱼类的调查较多[10-11],均是基于传统的形态学调查方法,基于太湖的eDNA鱼类多样性的研究鲜有报道。在长江“十年禁渔”的背景下,2020年10月太湖实施了全面退捕,为了更好地对太湖渔业资源进行监测,全面进行禁捕退捕效果评价,进而为保护太湖渔业生态环境提供较好支撑,本研究基于eDNA宏条形码技术开展太湖鱼类多样性资源调查,评估eDNA技术在太湖鱼类多样性监测与调查中的实用性。
1 材料与方法(Materials and methods)
1.1 采样点位设置
根据鱼类资源调查采样需求,依据网格法布点原则,在太湖设置了16个有代表性的采样点位,采样点分布见图1(采样位点简称S1~S16)。
1.2 采样及样品前处理
样品采集于2020年12月份。在每个采样点,用无菌聚丙烯瓶(Thermo, USA)采集2 L表层水(在水面以下约20 cm处采集),现场用0.45 μm混合纤维素酯(MCE)滤膜(易基诺,南京,中国)过滤,每张滤膜过滤300 mL水样,每个点位过滤滤膜6张作为平行样品,过滤后的滤膜采用干冰存储。16个采样位点、每个位点6个重复,后续分析按照每个位点的平均序列数计算。
1.3 DNA提取
对土壤基因组DNA提取试剂盒(天根,上海,中国)说明书上的方法略作修改,提取滤膜DNA。具体操作如下:将滤膜置于带有研磨珠的5 mL离心管内,加入800 μL缓冲液SA,使用eDNA均质仪(E01HBB012,易基诺,南京,中国)12 m·s-1的速度震荡30 s,间隔30 s,循环3次。加入150 μL缓冲液SC和15 μL蛋白酶K,使用eDNA均质仪(E01HBB012,易基诺,南京,中国)以12 m·s-1的速度震荡30 s,间隔30 s,循环3次。70 ℃加热裂解15 min,离心2 min,转移全部上清至新的2 mL离心管。加入10 μL RNase A,涡旋混匀,室温放置5 min。加入200 μL缓冲液SH,涡旋混匀后于4 ℃静置5 min,离心3 min;取上清400 μL至96深孔板第1/7列,后续使用全自动核酸提取纯化仪(E02EP32,易基诺,南京,中国)自动化程序提取。提取结束后将深孔板第5/11列DNA吸出,用于下游实验。
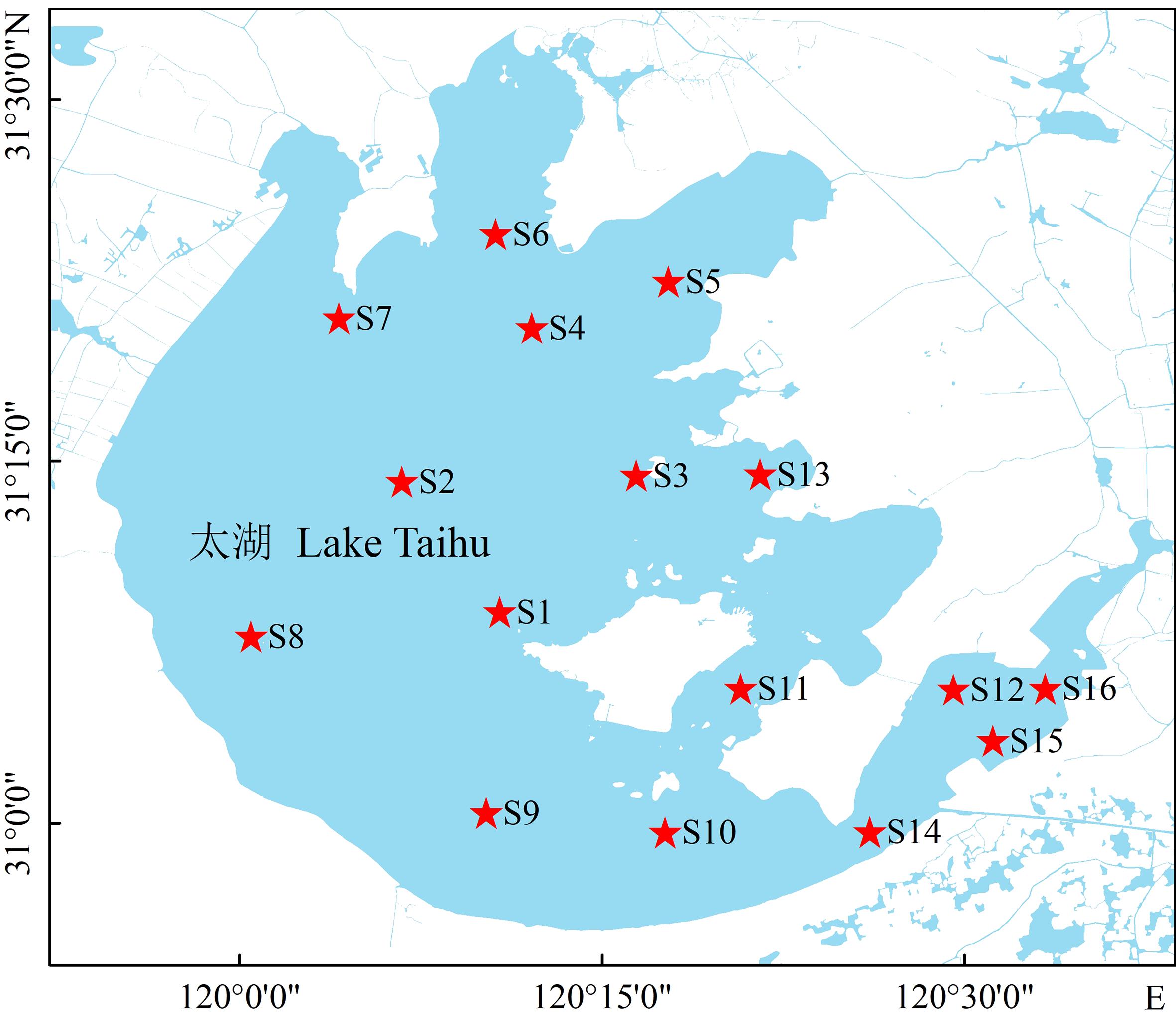
图1 监测点位图(S1~S16)
Fig. 1 Map of sampling stations (S1~S16)
1.4 PCR扩增
本研究利用线粒体DNA 12S rRNA区域引物进行鱼类宏条形码扩增[12],线粒体12S rRNA区域是鱼类检测中常用的引物区域,具有高可变性和高度保守性,该引物提高了鱼类eDNA扩增能力[12-13]。12S引物PCR扩增体系:总体系30 μL,包含Taq DNA polymerase 0.3 μL(Takara, Japan),10×PCR Buffer 3 μL,dNTP mixture 2.4 μL,上下游引物各1.5 μL,DNA模板2 μL,ddH2O 19.3 μL。扩增条件:95 ℃预变性3 min,95 ℃变性15 s,57 ℃退火20 s,72 ℃延伸20 s,35个循环。设置ddH2O为PCR阴性对照。PCR产物使用2%琼脂糖凝胶电泳进行目的条带检测,PCR阴性对照扩增结果无目的条带。使用AMPure XP核酸纯化试剂盒(Beckman Coulter, USA)磁珠纯化PCR产物。
1.5 高通量测序及数据分析
使用DNA Library Prep Kit试剂盒(诺唯赞,南京,中国)构建高通量测序文库,利用Ion Torrent平台测序仪(Ion Proton,赛默飞,美国)进行样品的高通量测序。测序数据获得后,全部基于EcoView软件(ubuntu 14.04版本)完成数据分析。首先过滤低质量序列、长度<80 bp和>250 bp的序列,再利用EcoView软件中USEARCH工具进行物种OTUs(Operational Taxonomic Units)聚类,基于NCBI数据库完成物种注释。获得的结果与刘燕山等[14]于2019—2020年对太湖的5次传统调查数据进行对比分析。
2 结果(Results)
2.1 高通量测序物种组成
高通量测序共获得2 522 310条序列,经注释后获得749 449条序列(图2(a))。其中,注释到辐鳍鱼纲的序列数308 906条,占总序列数的41.22%;注释到哺乳纲的序列数410 606条,占总序列数的54.79%;注释到鸟纲的序列数25 588条,占总序列数的3.41%;注释到两栖纲的序列数仅有135条,未注释出的序列数4 214条,占总序列数的0.56%。按序列相似性97%进行物种聚类,共获得432个OTUs(图2(b))。其中,252个辐鳍鱼纲OTUs,占比58.33%;147个哺乳纲OTUs,占比34.03%;24个鸟类OTUs,占比5.56%;1个两栖纲OTUs,8个未知OTUs,占比1.85%。
2.2 eDNA鱼类物种检测分析
经进一步质控,注释到鱼类物种的序列有253 129条,共注释到77个辐鳍鱼纲OTUs,隶属于8目20科47属54种(表1)。2个OTUs未注释到“种”水平,而只注释到“属”水平,为鳗虾虎鱼属某种(Taenioides sp.)、吻虾虎鱼属某种(Rhinogobius sp.)。注释到鲤形目的序列数最多,占比68.94%,物种数28种,主要是鲫、蒙古鲌、鳙等;注释到鲱形目序列数占比14.15%,物种数1种,主要为刀鲚;注释到鲈形目序列数占比13.10%,物种数8种,主要是子陵吻虾虎鱼(Rhinogobius similis)、河川沙塘鳢(Odontobutis potamophila)和小黄黝鱼(Micropercops swinhonis)等(图3(a))。
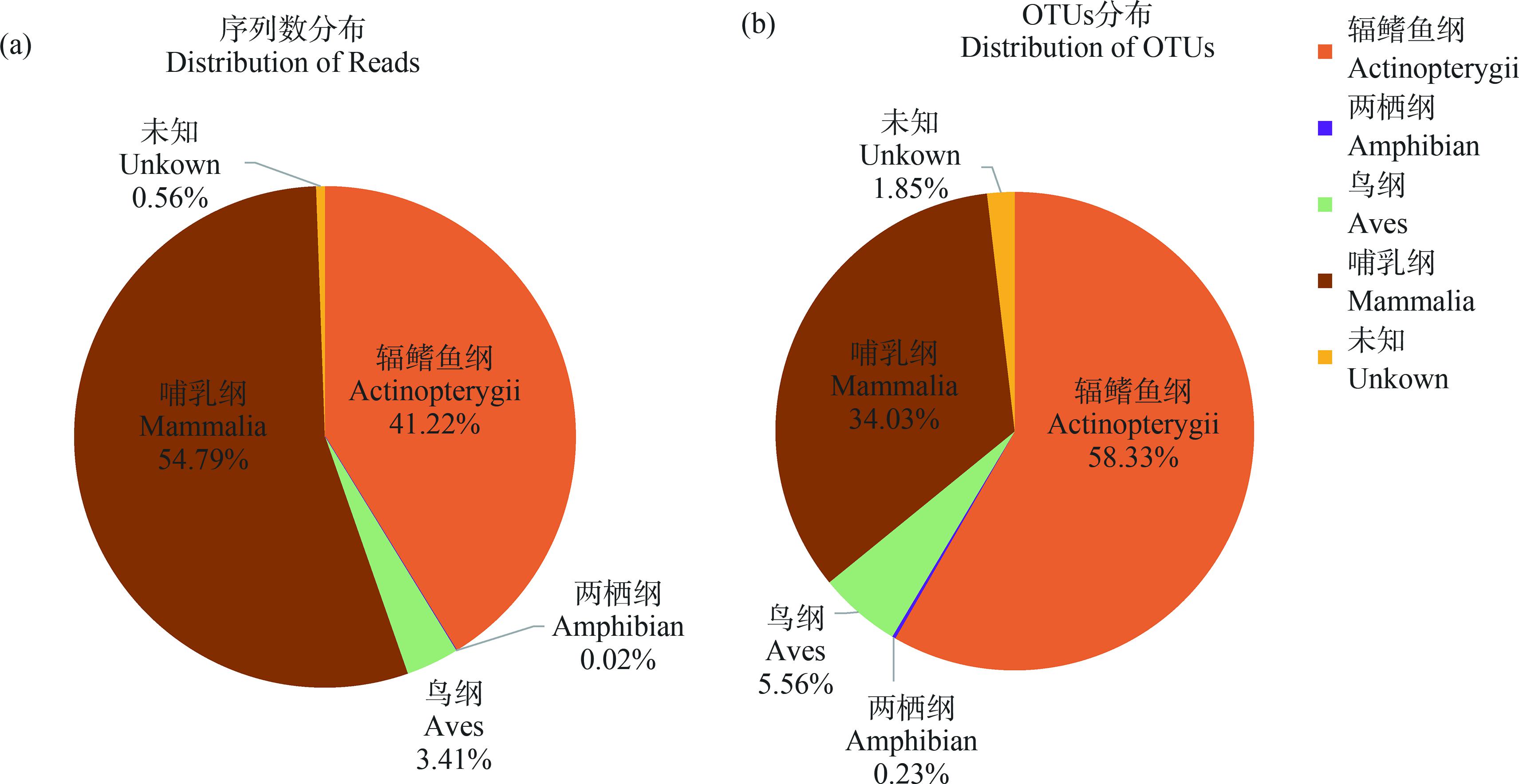
图2 太湖鱼类多样性测序结果
Fig. 2 The sequencing results of fish diversity in Lake Taihu
从16个采样点位分析(图3(b)),各位点检出的物种数不一,平均每个位点检出鱼类24种,其中,S3位点检出物种数最多,达33种,其次为S10,检出物种数31种;S8位点鱼类检出数最少,仅15种。7个

图3 基于eDNA技术的太湖鱼类群落组成及其与传统调查方法结果的比较
注:(a) 鱼类群落组成的序列占比与质量占比;(b) eDNA技术与传统调查方法在各位点发现物种数的比较, 数据点表示鱼类物种数,须线表示物种数偏差,方箱表示物种数平均值。
Fig. 3 The fish community based on eDNA metabarcoding and comparison of fish species investigation results between eDNA metabarcoding and the fishing survey
Note: (a) Sequence abundances and biomass percentage of fish community; (b) Comparison of fish species abundance at each sampling stations between the results based on eDNA metabarcoding and the fishing survey; spot represents number of species; whisker represents deviation; box represents average number of species.
表1 基于eDNA宏条形码的太湖鱼类物种
Table 1 The list of fish species detected by eDNA metabarcoding in Lake Taihu

目Order科Family属Genus种Species拉丁名Latin name检出频数 *Frequency鲤形目Cypriniformes鲤科Cyprinidae棒花鱼属棒花鱼Abbottina rivularis10鲌属蒙古鲌Culter mongolicus16翘嘴鲌Culter alburnus3赤眼鳟属赤眼鳟Squaliobarbus curriculus3鲂属鲂Megalobrama skolkovii2鱤属鳡Elopichthys bambusa1鲴属细鳞鲴Xenocypris microlepis15圆吻鲴属圆吻鲴Distoechodon tumirostris4光唇鱼属温州光唇鱼Acrossocheilus wenchowensis3p属唇pHemibarbus labeo9花pHemibarbus maculatus1鲫属鲫Carassius auratus16鲤属鲤Cyprinus carpio16鲢属鲢Hypophthalmichthys molitrix16马口鱼属马口鱼Opsariichthys bidens3麦穗鱼属麦穗鱼Pseudorasbora parva16鳑鲏属高体鳑鲏Rhodeus ocellatus6中华鳑鲏Rhodeus sinensis1飘鱼属寡鳞飘鱼Pseudolaubuca engraulis8鳈属黑鳍鳈Sarcocheilichthys nigripinnis2似鳊属似鳊Pseudobrama simoni6似刺鳊鮈属似刺鳊鮈Paracanthobrama guichenoti1似鱎属似鱎Toxabramis swinhonis12小鳔鮈属福建小鳔鮈Microphysogobio fukiensis7鳙属鳙Hypophthalmichthys nobilis16鱊属大鳍鱊Acheilognathus macropterus9兴凯鱊Acheilognathus chankaensis11鳅科Cobitidae泥鳅属泥鳅Misgurnus anguillicaudatus4鲱形目Clupeiformes鳀科Engraulidae鲚属刀鲚Coilia nasus16鲑形目Salmoniformes银鱼科Salangidae新银鱼属陈氏新银鱼Neosalanx tangkahkeii6颌针鱼目Beloniformes鱵科Hemiramphidae下鱵属间下鱵Hyporhamphus intermedius15鳉形目Cyprinodontiformes胎鳉科Poeciliidae食蚊鱼属食蚊鱼Gambusia affinis5鲈形目Perciformes沙塘鳢科Odontobutidae小黄黝鱼属小黄黝鱼Micropercops swinhonis12沙塘鳢属河川沙塘鳢Odontobutis potamophila12塘鳢科 Eleotridae塘鳢属尖头塘鳢Eleotris oxycephala4虾虎鱼科Gobiidae缟虾虎鱼属吻虾虎鱼属鲻虾虎鱼属鳗虾虎鱼属纹缟虾虎鱼Tridentiger trigonocephalus5子陵吻虾虎鱼Rhinogobius giurinus15波氏吻虾虎鱼Rhinogobius cliffordpopei4吻虾虎鱼某种Rhinogobius sp.2黏皮鲻虾虎鱼Mugilogobius myxodermus2鳗虾虎鱼某种Taenioides sp.8
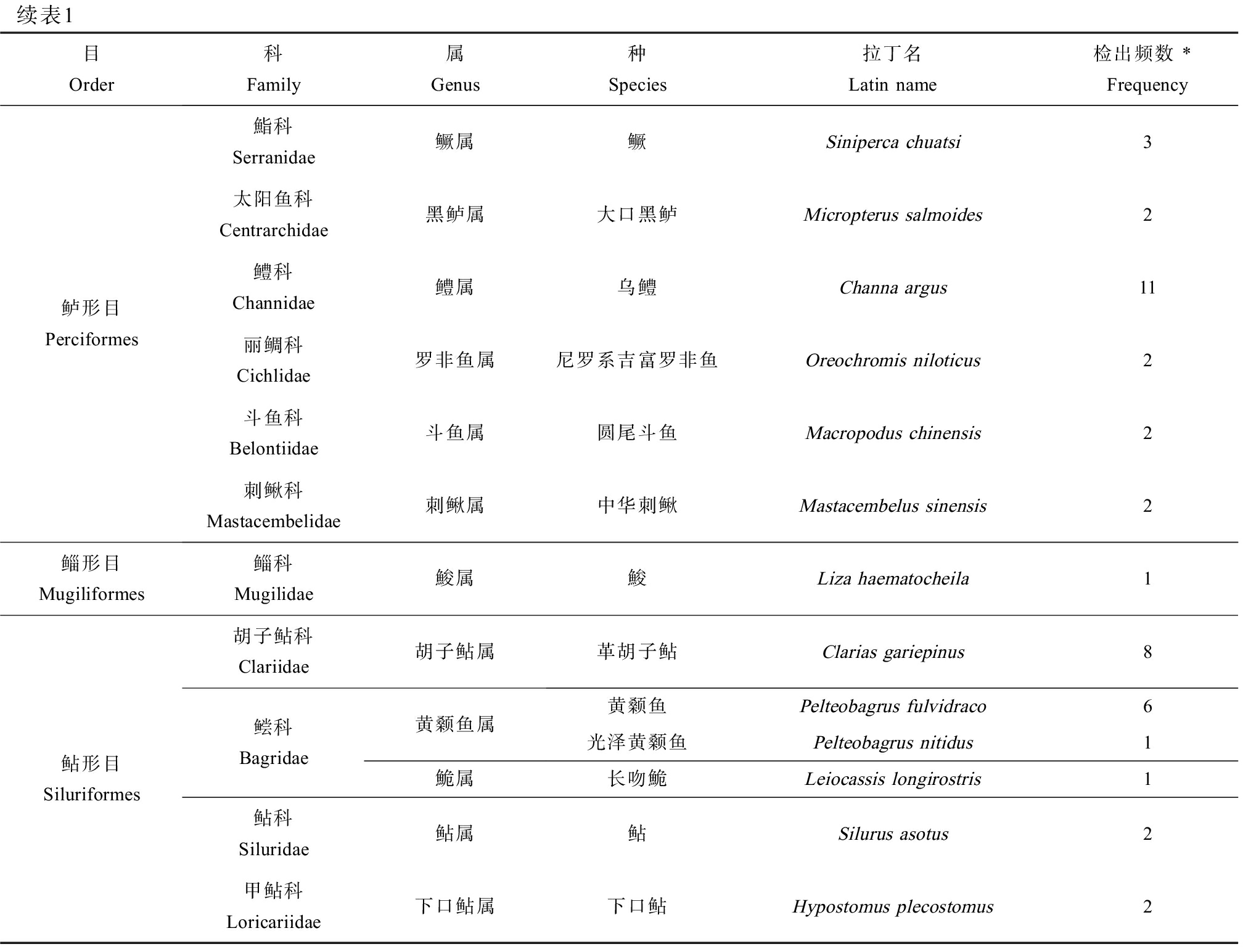
续表1目Order科Family属Genus种Species拉丁名Latin name检出频数 *Frequency鲈形目Perciformes鮨科Serranidae鳜属鳜Siniperca chuatsi3太阳鱼科Centrarchidae黑鲈属大口黑鲈Micropterus salmoides2鳢科Channidae鳢属乌鳢Channa argus11丽鲷科Cichlidae罗非鱼属尼罗系吉富罗非鱼Oreochromis niloticus2斗鱼科Belontiidae斗鱼属圆尾斗鱼Macropodus chinensis2刺鳅科Mastacembelidae刺鳅属中华刺鳅Mastacembelus sinensis2鲻形目Mugiliformes鲻科Mugilidae鮻属鮻Liza haematocheila1鲇形目Siluriformes胡子鲇科Clariidae胡子鲇属革胡子鲇Clarias gariepinus8鲿科Bagridae黄颡鱼属鮠属黄颡鱼Pelteobagrus fulvidraco6光泽黄颡鱼Pelteobagrus nitidus1长吻鮠Leiocassis longirostris1鲇科Siluridae鲇属鲇Silurus asotus2甲鲇科Loricariidae下口鲇属下口鲇Hypostomus plecostomus2
注:*表示总频数为16。
Note: *means a total frequency of 16.
物种在16个点位均有检出,主要为鲫(Carassius auratus)、蒙古鲌(Chanodichthys mongolicus)、刀鲚(Coilia nasus)、鲤(Cyprinus carpio)、鳙(Hypophthalmichthys nobilis)、鲢(Hypophthalmichthys molitrix)、麦穗鱼(Pseudorasbora parva);7种鱼类似刺鳊鮈(Paracanthobrama guichenoti)、中华鳑鲏(Rhodeus sinensis)、长吻鮠(Leiocassis longirostris)、光泽黄颡鱼(Pelteobagrus nitidus)、花 (Hemibarbus maculatus)、鳡(Elopichthys bambusa)和鮻(Liza haematocheila)仅在1个位点有检出。
(Hemibarbus maculatus)、鳡(Elopichthys bambusa)和鮻(Liza haematocheila)仅在1个位点有检出。
2.3 eDNA与传统形态学鱼类多样性调查方法对比
对比分析1次eDNA与5次传统形态学鱼类多样性调查结果发现,2种方法共同检出鱼类37种,占eDNA检出鱼类物种数的68.51%,占鱼类检出序列数的91.89%。
eDNA调查中,未共同检出的17种鱼类中,9种在太湖鱼类志中有记载,分别为寡鳞飘鱼(Pseudolaubuca engraulis)、唇 (H. labeo)、福建小鳔鮈(Microphysogobio fukiensis)、赤眼鳟(Squaliobarbus curriculus)、圆吻鲴(Distoechodon tumirostris)、马口鱼(Opsariichthys bidens)、长吻鮠(Leiocassis longirostris)、鳡(Elopichthys bambusa)和鮻(Liza haematocheila);3种为未记录过的物种,为尖头塘鳢(Eleotris oxycephala)、黏皮髭虾虎鱼(Mugilogobius myxodermus)和温州光唇鱼(Acrossocheilus wenchowensis);另外5种为外来物种,有食蚊鱼(Gambusia affinis)、大口黑鲈(Micropterus salmoides)、尼罗系吉富罗非鱼(Oreochromis niloticus)、革胡子鲶(Clarias gariepinus)、下口鲇(Hypostomus plecostomus)。5次传统形态学调查中,单独调查到的鱼类共19种,其中18种在太湖鱼类志中有记载,另外1种花鳗鲡(Anguilla marmorata)为新发现物种。eDNA调查与传统形态学共同发现的纹缟虾虎鱼(Tridentiger trigonocephalus)在太湖鱼类志未有记载。
(H. labeo)、福建小鳔鮈(Microphysogobio fukiensis)、赤眼鳟(Squaliobarbus curriculus)、圆吻鲴(Distoechodon tumirostris)、马口鱼(Opsariichthys bidens)、长吻鮠(Leiocassis longirostris)、鳡(Elopichthys bambusa)和鮻(Liza haematocheila);3种为未记录过的物种,为尖头塘鳢(Eleotris oxycephala)、黏皮髭虾虎鱼(Mugilogobius myxodermus)和温州光唇鱼(Acrossocheilus wenchowensis);另外5种为外来物种,有食蚊鱼(Gambusia affinis)、大口黑鲈(Micropterus salmoides)、尼罗系吉富罗非鱼(Oreochromis niloticus)、革胡子鲶(Clarias gariepinus)、下口鲇(Hypostomus plecostomus)。5次传统形态学调查中,单独调查到的鱼类共19种,其中18种在太湖鱼类志中有记载,另外1种花鳗鲡(Anguilla marmorata)为新发现物种。eDNA调查与传统形态学共同发现的纹缟虾虎鱼(Tridentiger trigonocephalus)在太湖鱼类志未有记载。
从物种单次检出总数看,eDNA技术单次检出的鱼类物种数显著高于传统形态学。12个位点eDNA调查发现的鱼类物种数均高于传统形态学调查,传统形态学每个点位鱼类物种数为8.00~14.20种,eDNA调查发现的鱼类物种数为10.20~19.00种(图3(b))。表明,鱼类多样性调查中,单次、单位点物种检出数eDNA调查方法显著高于传统形态学调查方法(P<0.05)。
分析eDNA和传统形态学共同检出的鱼类物种质量占比与序列占比(图4(a)),2种方法共同检出的鱼类物种组成相似性极高(r2=0.70,P<0.001),表明2种方法物种检出一致性较高。共同检出物种在各点位物种总丰度均高于85%(S15点位传统形态学物种检出丰度除外),且2种方法在各点位物种检出丰度上一致性较高(图4(b))。
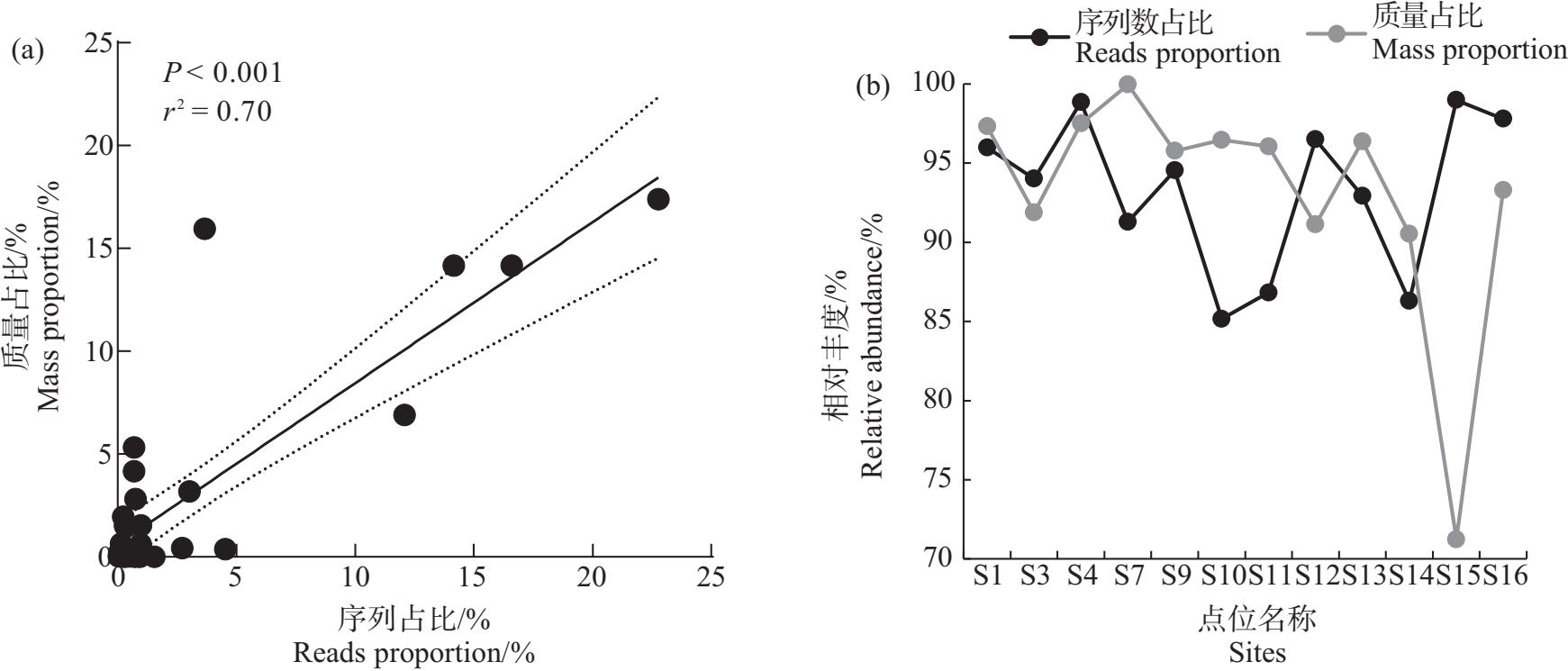
图4 属水平上eDNA技术(序列占比)与传统调查方法(质量占比)共同检出结果对比分析
Fig. 4 Comparison of fish species abundance in genera level between the results based on eDNA metabarcoding and the fishing survey
2.4 eDNA与传统形态学生态类型对比
按鱼类的生活习性洄游类型、栖息水层和食性比较分析2种方法检出鱼类的生态类型(图5),鱼类洄游类型2种方法检出结果均以定居性为主;鱼类栖息水层分析发现,占比最高的鱼类主要为生活在中上层的鱼类,2种检出方法结果一致;鱼类食性分析结果表明,太湖鱼类以杂食性和肉食性为主,2种方法检出结果较一致。对2种方法检出的鱼类洄游性、栖息水层和食性对比分析表明,生物量与序列之间存在显著相关性(P<0.001, r2=0.81)。
3 讨论(Discussion)
3.1 基于eDNA的太湖鱼类多样性检测方法
随着eDNA技术的发展,越来越多的学者利用eDNA技术调查湖泊、河流的鱼类多样性[15-18],本研究利用eDNA技术调查太湖16个采样位点的鱼类组成,并对其鱼类多样性进行初步分析,调查共发现鱼类54种,根据Shannon-Wiener多样性指数(H)进一步研究物种分布(图6),Shannon-Wiener多样性指数在1.7~3.0之间,太湖东部和太湖南部鱼类多样性相对较高,太湖北部和太湖西部相对较低,与2019—2020年传统形态学方法的鱼类群落调查结果类似。这可能与太湖生态状况有关,太湖西北部湖区富营养化程度较高,太湖东南部湖区水草茂盛,水体透明度较高、水质较好[19-20],后续可针对这些点位的水生态状况与鱼类多样性的关系开展进一步研究。
3.2 太湖eDNA生物多样性检测与传统鱼类检测方法比较
太湖鱼类志记载鱼类物种107种[21],基于eDNA技术检出的鱼类中,有45种鱼类在太湖鱼类志有记载,占检出鱼类序列数的95.09%;其余为未记录过的本土物种(4种)和外来物种(5种)。与2019—2020年传统形态学调查方法对比发现,eDNA方法与传统形态学调查方法鱼类物种检出一致率较高。共同检出的37种鱼类序列数占eDNA检测序列数的91.89%。就共同检出的物种在属水平上比较,2种调查方法在各检测点位检出的物种基本吻合,2种调查方法在各点位物种属水平检出一致率达65.63%。
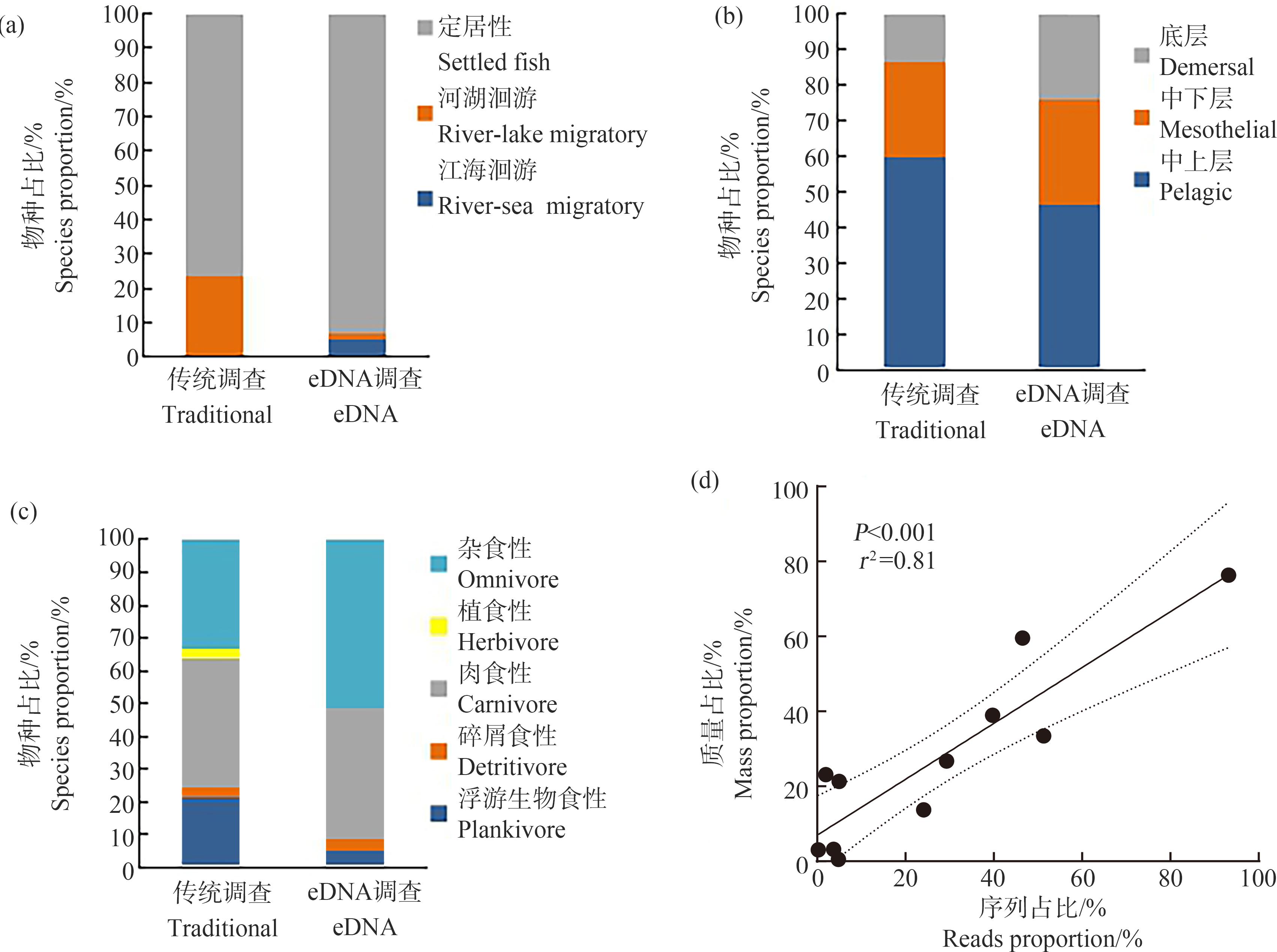
图5 eDNA方法与传统形态学方法鱼类生态类型对比分析
注:(a) 鱼类洄游类型;(b) 鱼类栖息水层;(c) 鱼类食性;(d) 生活习性。
Fig. 5 Comparison of fish ecological types between the results based on eDNA metabarcoding and the fishing survey
Note: (a) Migratory; (b) Habit water layer; (c) Food habits; (d) Habits of life.

图6 基于Shannon-Wiener多样性指数的鱼类多样性分布
Fig. 6 Distribution of fish diversity based on Shannon-Wiener Index in Lake Taihu
eDNA调查技术物种检出效率显著高于传统形态学[22]。2020年eDNA技术单频次调查物种检出数为54种,而传统形态学单频次调查检出物种数在32~42种之间,eDNA单频次调查总物种检出数高于传统形态学;调查的12个位点中,eDNA调查发现的鱼类物种数均高于传统形态学调查,传统形态学调查各点位鱼类物种数为8.00~14.20种,eDNA调查发现的鱼类物种数为10.20~19.00种,单个点位检出种类数eDNA调查高于传统形态学调查。另外,eDNA技术能够检测出一些生物量较低、而被传统形态学方法漏检的物种[19]。如鳡、鮻2种鱼类在本次eDNA技术中有检出,但并未在刘燕山等[14]的5次传统形态学调查中检出,这表明eDNA鱼类调查方法灵敏度高于传统形态学调查方法,尤其是对低丰度鱼类检出,eDNA调查方法更加灵敏。
对太湖鱼类生态类型的检测,2种调查方法检出一致率较高,表明eDNA调查方法可靠性较高。栖息水层中,均以中上层鱼类为主,而食性均以杂食性和肉食性为主,均有鲫、刀鲚,可能与太湖饵料资源现状有关[23]。2种方法检出的鱼类主要以定居性为主,与历年太湖鱼类调查结果一致;洄游性鱼类较少,可能与20世纪太湖通江水道修建水闸,隔断太湖与长江的联系有关[24]。
3.3 eDNA生物多样性检测应用前景
随着生物多样性监测需求的提升,以及eDNA技术的不断优化,基于eDNA技术的生物多样性调查和研究工作陆续开展[25-28]。传统的鱼类监测耗时耗力,人员成本较高,且会对生态环境造成一定程度的影响。eDNA技术在生物多样性监测方面具有显著优势,用该方法调查鱼类多样性与传统形态学调查方法的一致率较高,在不破坏生态系统环境的同时可实现生物快速监测,适合全流域水生生物多样性的全面普查工作。同时eDNA技术可以发现传统监测中未捕捞到的鱼类,灵敏度高,可用于珍稀濒危物种或入侵物种的研究或预警[29-30]。目前,eDNA技术属于半定量研究,与传统鱼类监测方法相比各具优势[31],可以根据研究目的选择合适的调查方法。
[1] Deiner K, Bik H M, Mächler E, et al. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities [J]. Molecular Ecology, 2017, 26(21): 5872-5895
[2] Dejean T, Valentini A, Duparc A, et al. Persistence of environmental DNA in freshwater ecosystems [J]. PLoS One, 2011, 6(8): e23398
[3] Yoccoz N G. The future of environmental DNA in ecology [J]. Molecular Ecology, 2012, 21(8): 2031-2038
[4] 舒璐, 林佳艳, 徐源, 等. 基于环境DNA宏条形码的洱海鱼类多样性研究[J]. 水生生物学报, 2020, 44(5): 1080-1086
Shu L, Lin J Y, Xu Y, et al. Investigating the fish diversity in Erhai Lake based on environmental DNA metabarcoding [J]. Acta Hydrobiologica Sinica, 2020, 44(5): 1080-1086 (in Chinese)
[5] Evans N T, Olds B P, Renshaw M A, et al. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding [J]. Molecular Ecology Resources, 2016, 16(1): 29-41
[6] Thomsen P F, Kielgast J, Iversen L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA [J]. Molecular Ecology, 2012, 21(11): 2565-2573
[7] Fujii K, Doi H, Matsuoka S, et al. Environmental DNA metabarcoding for fish community analysis in backwater lakes: A comparison of capture methods [J].PLoS One, 2019, 14(1): e0210357
[8] 徐念, 常剑波. 长江中下游干流环境DNA样本鱼类物种检测的初步研究[J]. 水生态学杂志, 2016, 37(5): 49-55
Xu N, Chang J B. Preliminary study on fish species detection in the middle and lower Yangtze River using environmental DNA [J]. Journal of Hydroecology, 2016, 37(5): 49-55 (in Chinese)
[9] 孙晶莹, 杨江华, 张效伟. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究[J]. 生态毒理学报, 2018, 13(5): 76-86
Sun J Y, Yang J H, Zhang X W. Identification and biomass monitoring of zooplankton Cladocera species witheDNA metabarcoding technology [J]. Asian Journal of Ecotoxicology, 2018, 13(5): 76-86 (in Chinese)
[10] 唐晟凯, 张彤晴, 沈振华, 等. 太湖鱼类学调查及渔获物分析[J]. 江苏农业科学, 2010, 38(2): 376-379
[11] 李其芳, 严云志, 储玲, 等. 太湖流域河流鱼类群落的时空分布[J]. 湖泊科学, 2016, 28(6): 1371-1380
Li Q F, Yan Y Z, Chu L, et al. Spatial and temporal patterns of stream fish assemblages within Taihu Basin [J]. Journal of Lake Sciences, 2016, 28(6): 1371-1380 (in Chinese)
[12] 杨江华, 张效伟. 一种淡水鱼类线粒体12S通用宏条形码扩增引物及其应用方法: CN109943645A [P]. 2019-06-28.
[13] Yang J H, Zhang L J, Mu Y W, et al. Small changes make big progress: A more efficient eDNA monitoring method for freshwater fish [J]. Environmental DNA, 2023, 5(2): 363-374
[14] 刘燕山, 李大命, 朱明胜, 等. 太湖鱼类群落现状及其多样性[J]. 长江流域资源与环境, 2022, 31(9): 1906-1917
Liu Y S, Li D M, Zhu M S, et al. Current status of fish community and its diversity in Lake Taihu, China [J]. Resources and Environment in the Yangtze Basin, 2022, 31(9): 1906-1917 (in Chinese)
[15] Valdez-Moreno M, Ivanova N V, Elías-Gutiérrez M, et al. Using eDNA to biomonitor the fish community in a tropical oligotrophic lake [J]. PLoS One, 2019, 14(4): e0215505
[16] Thomsen P F, Kielgast J, Iversen L L, et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples [J]. PLoS One, 2012, 7(8): e41732
[17] 唐晟凯, 刘燕山, 王华, 等. 环境DNA技术在邵伯湖鱼类资源监测中的应用[J]. 水产科学, 2022, 41(6): 1007-1016
Tang S K, Liu Y S, Wang H, et al. Application of environmental DNA in monitoring of fish resources in Shaobo Lake [J]. Fisheries Science, 2022, 41(6): 1007-1016 (in Chinese)
[18] 李晓玲, 刘洋, 王丛丛, 等. 基于环境DNA技术的夏季东海鱼类物种多样性研究[J]. 海洋学报, 2022, 44(4): 74-84
Li X L, Liu Y, Wang C C, et al. Study on fish species diversity in the East China Sea in summer based on environmental DNA technology [J]. Acta Oceanologica Sinica, 2022, 44(4): 74-84 (in Chinese)
[19] 张翔, 周国栋, 王雷. 太湖鱼类生物完整性指数构建与健康评价[J]. 水产学杂志, 2020, 33(1): 25-32
Zhang X, Zhou G D, Wang L. Construction of index of biotic integrity and health evaluation of fish in Taihu Lake [J]. Chinese Journal of Fisheries, 2020, 33(1): 25-32 (in Chinese)
[20] 张翔, 沈伟, 周国栋. 2018—2020年太湖鱼类群落结构及其环境因子典范对应分析[J]. 生态与农村环境学报, 2021, 37(5): 674-680
Zhang X, Shen W, Zhou G D. Status of the fish community and canonical correspondence analysis of environmental factors in Lake Taihu from 2018 to 2020 [J]. Journal of Ecology and Rural Environment, 2021, 37(5): 674-680 (in Chinese)
[21] 倪勇, 朱成德. 太湖鱼类志[M]. 上海: 上海科学技术出版社, 2005: 23
Ni Y, Zhu C D. Fishes of the Taihu Lake [M]. Shanghai: Shanghai Scientific and Technical Publishers, 2005: 23
[22] Bohmann K, Evans A, Gilbert M T, et al. Environmental DNA for wildlife biology and biodiversity monitoring [J]. Trends in Ecology &Evolution, 2014, 29(6): 358-367
[23] 刘燕山, 谷先坤, 唐晟凯, 等. 东太湖鱼类群落结构特征及其与环境因子的关系[J]. 生态学报, 2021, 41(2): 769-780
Liu Y S, Gu X K, Tang S K, et al. Fish community structure and its relationship with main environmental variables in the East Taihu Lake [J]. Acta Ecologica Sinica, 2021, 41(2): 769-780 (in Chinese)
[24] 刘恩生, 刘正文, 陈伟民, 等. 太湖鱼类产量、组成的变动规律及与环境的关系[J]. 湖泊科学, 2005, 17(3): 251-255
Liu E S, Liu Z W, Chen W M, et al. Changes in the yield and composition of the fish catches and their relation to the environmental factors in Lake Taihu [J]. Journal of Lake Science, 2005, 17(3): 251-255 (in Chinese)
[25] 刁曹鋆, 王闻, 线薇薇, 等. 环境DNA技术在渔业资源生物量评估中的研究进展: 现状与展望[J]. 海洋科学, 2022, 46(2): 135-144
Diao C Y, Wang W, Xian W W, et al. Role of the environmental DNA technology application in the biomass assessment of the fishery resource: Current status and future perspectives [J]. Marine Sciences, 2022, 46(2): 135-144 (in Chinese)
[26] Li M, Shan X J, Wang W J, et al. Qualitative and quantitative detection using eDNA technology: A case study of Fenneropenaeus chinensis in the Bohai Sea [J]. Aquaculture and Fisheries, 2020, 5(3): 148-155
[27] 赵彦伟, 陈家琪, 董丽, 等. 环境DNA技术在水生态领域应用研究进展[J]. 农业环境科学学报, 2021, 40(10): 2057-2065
Zhao Y W, Chen J Q, Dong L, et al. Advances in the application of environmental DNA in aquatic ecosystems [J]. Journal of Agro-Environment Science, 2021, 40(10): 2057-2065 (in Chinese)
[28] Imamura A, Hayami K, Sakata M K, et al. Environmental DNA revealed the fish community of Hokkaido Island, Japan, after invasion by rainbow trout [J]. Biodiversity Data Journal, 2020, 8: e56876
[29] 林渊源, 赵峥. 环境DNA技术在水生入侵生物监测中的应用[J]. 生态毒理学报, 2021, 16(6): 1-12
Lin Y Y, Zhao Z. Application of environmental DNA technologies in monitoring aquatic invasive species [J]. Asian Journal of Ecotoxicology, 2021, 16(6): 1-12 (in Chinese)
[30] 吴昀晟, 唐永凯, 李建林, 等. 环境DNA在长江江豚监测中的应用[J]. 中国水产科学, 2019, 26(1): 124-132
Wu Y S, Tang Y K, Li J L, et al. The application of environmental DNA in the monitoring of the Yangtze finless porpoise,Neophocaena phocaenoides asaeorientalis [J]. Journal of Fishery Sciences of China, 2019, 26(1): 124-132 (in Chinese)
[31] Evans N T, Lamberti G A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool [J]. Fisheries Research, 2018, 197: 60-66