抗生素自问世以来被广泛用于治疗人类或动物的传染性疾病、预防细菌传播疾病以及促进家畜生长[1-2]。氟喹诺酮类抗生素(fluoroquinolone antibiotics, FQs)作为人畜共用抗生素在全球生产和使用量中占据第3位[3]。70%的FQs会以未代谢的形式排到环境中,而FQs对生物降解具有抵抗性,并且具有高吸附亲和力,这导致了其在环境中的持久性。据报道,FQs在土壤中的半衰期长达580 d,在地表水中的半衰期为10.6 d[4]。环境中的FQs很可能会与其他FQs或污染物以混合物的形式存在,而混合物的累积毒性与相互作用使其具有更大的潜在毒性效应[5]。环境中的污染物或混合物很可能会通过食物链传递的方式进入人体中,对人体健康造成威胁。因此,研究FQs混合污染物对环境中生物有机体的毒性作用具有重要的实际环境意义。
群体感应(quorum sensing, QS)是一种常见的依赖于细菌密度的沟通协调机制,它控制着细菌的生长、增殖、致病性、生物被膜的形成以及相关的群体活动[6]。以QS系统为靶点的群体感应抑制剂(quorum sensing inhibitors, QSIs)通过干扰细菌信号分子的合成、信号分子的失活或酶促破坏,防止积累到阈值、干扰细菌细胞中信号受体的结合或与信号分子(受体类似物)的竞争阻断本应由QS信号触发的靶基因等方法来减弱其致病性[7]。有研究表明,QSIs能阻断铜绿假单胞菌的毒力和生物膜的形成[8]。此外,QSIs能特异性地干扰细菌的QS系统,降低细菌对抗生素的耐药性,如近期有研究者发现一种新型群体感应抑制剂能抑制金黄色葡萄球菌在体内的活性而不产生显著耐药性[9]。因此,QS抑制剂被认为是抗生素替代品,并被建议与抗生素联合使用[10-11],以减少抗生素的使用,增强抗菌效果[12-13]。目前,已有研究发现溴化呋喃酮[14]、金缕梅单宁[15]与抗生素联合使用能增加细菌生物膜对抗生素的敏感性,6-姜酚和环丙沙星联合使用能提高抗生素对铜绿假单胞菌的抗菌能力[16]。因而,开展抗生素与QSIs联合作用的研究不仅有助于探索“提高抗生素抗菌能力、减少抗生素使用量”的方法,同时有助于了解其进入环境后是否对生态环境产生影响及影响程度,而相关的研究较少。
综上,本文拟选择氟喹诺酮类抗生素包括氧氟沙星(ofloxacin, OFL)、左氧氟沙星(levofloxacin, LEV)和群体感应抑制剂4-羟基-2,5-二甲基-3(2H)呋喃酮(4-hydroxy-2,5-dimethyl-3(2H)-furanone, HDMF)为研究对象,以大肠杆菌(Escherichia coli, E. coli)为受试生物,采用直接均分射线法(direct equipartition ray, EquRay)设计3个二元混合体系(OFL-LEV、LEV-HDMF、OFL-HDMF)和均匀设计射线法(uniform design ray, UD-Ray)设计1个三元混合体系(HDMF-OFL-LEV),采用时间毒性微板分析法测定分析3种药物对E. coli在不同暴露时间的浓度效应数据,利用拟合归零法对混合物相互作用类型和强度进行分析,采用分子对接研究单个抗生素、QSIs及其靶蛋白之间的相互作用机理。研究结果可为解析抗生素和群体感应抑制剂的联合作用以及环境风险评价提供参考。
1 材料与方法(Materials and methods)
1.1 菌种培养与实验药剂
实验菌种大肠杆菌(E. coli)购自上海麦克林生化科技有限公司,E. coli的培养与保存方法参见文献[17]。氧氟沙星(OFL,分子量361.37)、左氧氟沙星(LEV,分子量361.37)、4-羟基-2,5-二甲基-3(2H)呋喃酮(HDMF,分子量128.13)纯度均>97%,购自国药集团化学试剂有限公司。药物的储备液用Milli-Q水配制,保存在棕色试剂瓶中,并置于4 ℃冰箱中保存,基本理化性质见表1。
1.2 微板毒性分析法
实验载体采用透明96孔微板,微板毒性分析方法参见文献[18]。3种药物对E. coli抑制率计算公式如下。
(1)
式中:E表示药物对E. coli抑制率;I0表示空白对照组的OD600平均值;I表示各浓度梯度OD600的平均值。
1.3 混合物实验设计
采用直接均分射线法(EquRay)[19]设计3个二元混合物体系(OFL-HDMF、LEV-HDMF、OFL-LEV),用均匀设计法(UD-Ray)[20]设计1个三元混合体系(HDMF-OFL-LEV),每组混合物体系安排了5条不同浓度比的混合物射线(R1、R2、R3、R4、R5),具体浓度比见表2。
表1 3种药物的理化性质及函数的拟合参数
Table 1 Basic physicochemical properties of three drugs and fitting parameters of the function
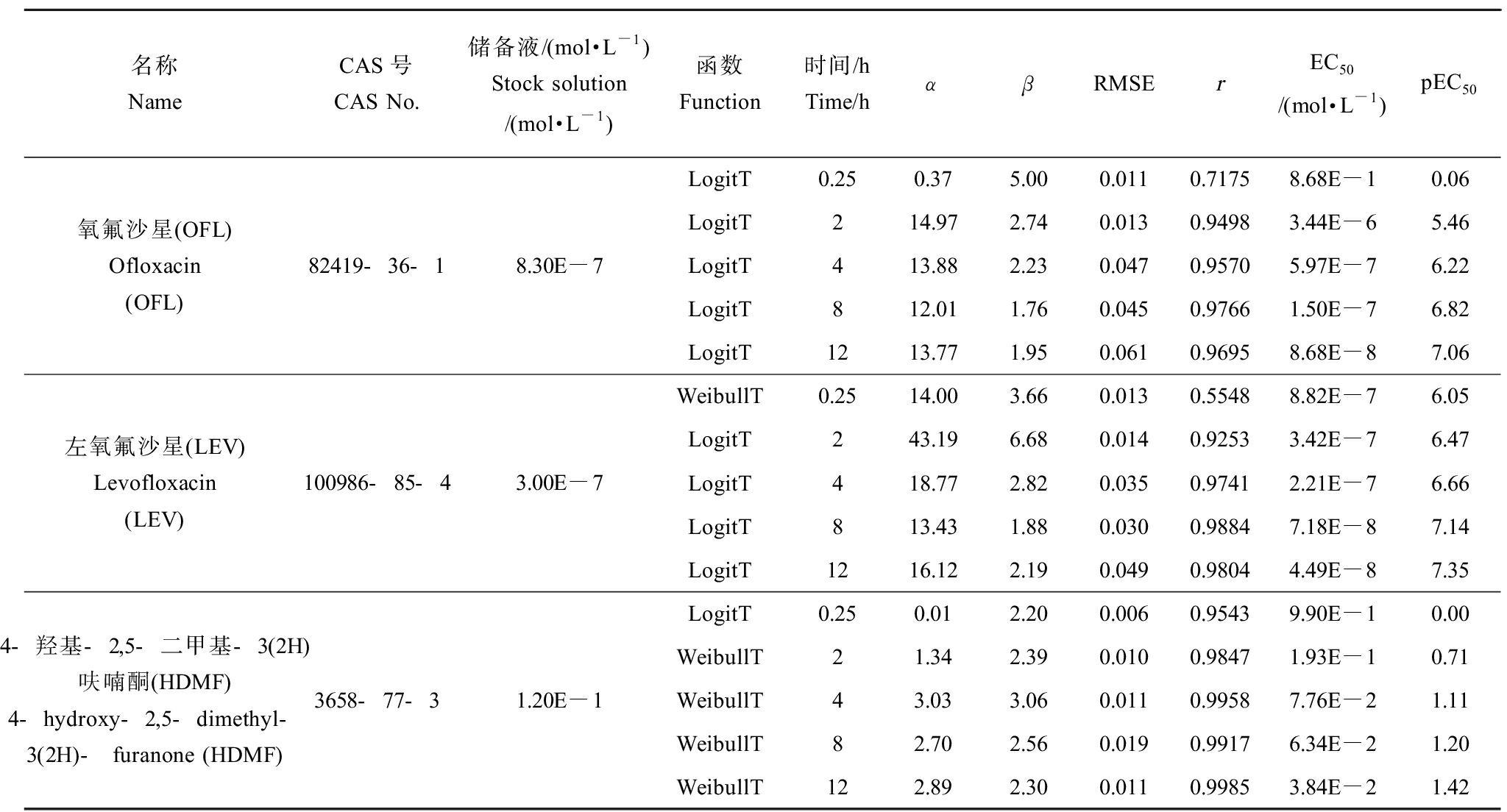
名称NameCAS号CAS No.储备液/(mol·L-1)Stock solution/(mol·L-1)函数Function时间/hTime/hαβRMSErEC50/(mol·L-1)pEC50氧氟沙星(OFL)Ofloxacin(OFL)82419-36-18.30E-7LogitT0.250.375.000.0110.71758.68E-10.06LogitT214.972.740.0130.94983.44E-65.46LogitT413.882.230.0470.95705.97E-76.22LogitT812.011.760.0450.97661.50E-76.82LogitT1213.771.950.0610.96958.68E-87.06左氧氟沙星(LEV)Levofloxacin(LEV)100986-85-43.00E-7WeibullT0.2514.003.660.0130.55488.82E-76.05LogitT243.196.680.0140.92533.42E-76.47LogitT418.772.820.0350.97412.21E-76.66LogitT813.431.880.0300.98847.18E-87.14LogitT1216.122.190.0490.98044.49E-87.354-羟基-2,5-二甲基-3(2H)呋喃酮(HDMF)4-hydroxy-2,5-dimethyl-3(2H)- furanone (HDMF)3658-77-31.20E-1LogitT0.250.012.200.0060.95439.90E-10.00WeibullT21.342.390.0100.98471.93E-10.71WeibullT43.033.060.0110.99587.76E-21.11WeibullT82.702.560.0190.99176.34E-21.20WeibullT122.892.300.0110.99853.84E-21.42
注:RMSE表示均方根误差,r表示相关系数。
Note: RMSE stands for root mean square error; r represents the correlation coefficient.
表2 混合物体系组分的浓度配比
Table 2 Concentration ratio of each component in mixture system
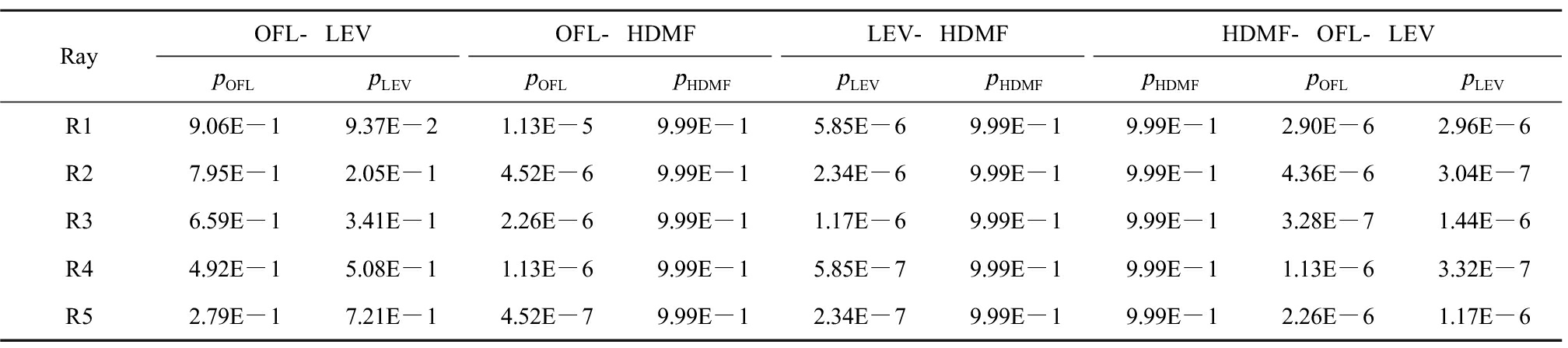
RayOFL-LEVOFL-HDMFLEV-HDMFHDMF-OFL-LEVpOFLpLEVpOFLpHDMFpLEVpHDMFpHDMFpOFLpLEVR19.06E-19.37E-21.13E-59.99E-15.85E-69.99E-19.99E-12.90E-62.96E-6R27.95E-12.05E-14.52E-69.99E-12.34E-69.99E-19.99E-14.36E-63.04E-7R36.59E-13.41E-12.26E-69.99E-11.17E-69.99E-19.99E-13.28E-71.44E-6R44.92E-15.08E-11.13E-69.99E-15.85E-79.99E-19.99E-11.13E-63.32E-7R52.79E-17.21E-14.52E-79.99E-12.34E-79.99E-19.99E-12.26E-61.17E-6
1.4 浓度-效应曲线拟合
对于不同暴露时间对应的浓度-效应数据应用APTox软件中的Weibull函数或Logit函数进行非线性拟合[21]。Weibull、Logit函数公式如下:
E=1-exp[-exp(α+βlgc)]
(2)
(3)
式中:E表示抑制率;α和β分别表示位置和斜率参数;c表示单个化合物或混合物的浓度。
1.5 混合物毒性相互作用评估
采用浓度加和(concentration addition, CA)模型来评估混合物之间的相互作用[22],其表达式为:
(4)
式中:m表示混合物组分数,ci表示混合物产生x%效应时第i组分的浓度;ECx,i为i组分单独存在时产生等效应x%时的效应浓度。当CA预测线在整个95%置信区间以上、之间、以下时,可以认为混合物的毒性分别为拮抗、加和和协同作用。
CA模型能够评估混合物的毒性相互作用,但相互作用强度无法准确评定,因此采用绝对残差(deviation from CA model, dCA)模型来表征混合物毒性相互作用的强度[23]。dCA模型的表达式如式(5)所示,在置信区间外,dCA的绝对值越大表示混合物的相互作用(协同/拮抗作用)越强。
dCAi,C=EOBS-EPRE
(5)
式中:EOBS代表实验观测值,EPRE代表预测值。
1.6 分子对接
以大肠杆菌的DNA回旋酶(DNA gyrase)、DNA拓扑异构酶Ⅳ(DNA topoisomerase Ⅳ)和群体感应(QS)受体SdiA蛋白和LsrR蛋白作用靶点为研究对象,对DNA回旋酶(PDB ID:6YD9)、DNA拓扑异构酶Ⅳ(PDB ID:3FV5)、SdiA蛋白(PDB ID:4LGW)和LsrR蛋白(PDB ID:4L4Y)4个靶点蛋白作为受体,OFL、LEV和HDMF作为配体,使用AutoDock Vina对蛋白的活性点位与配体进行分子对接,采用结合能(binding affinity, kJ·mol-1)作为评价参数,最大负值即为最稳定对接构象[24]。
2 结果(Results)
2.1 2种FQs与QSIs对E. coli的毒性
采用非线性函数(Logit函数或Weibull函数)对数据进行拟合,选取拟合效果较好的函数来绘制CRC曲线,拟合结果如表1所示,3种药物的时间-浓度-效应曲线绘于图1中。
从表1可知,3种药物对E. coli除了在0.25 h拟合数据较差外,在其余暴露时间抑菌数据均能被函数较好地拟合(r>0.90, RMSE<0.10),以半数效应浓度负对数(pEC50)为毒性指标,其毒性大小顺序为:2 h时,LEV(pEC50=6.42)>OFL(pEC50=5.46)>HDMF(pEC50=0.71);在12 h,毒性大小顺序为LEV(pEC50=7.35)>OFL(pEC50=7.06)>HDMF(pEC50=1.42)。其中,HDMF的毒性明显低于抗生素,可能是由于QSIs主要针对细菌的QS通信系统,并不直接导致细菌死亡。这与Ning等[25]的发现相似,即抗生素对E. coli的毒性显著高于QSIs的。
3种药物的时间-浓度-效应(t-CRC)曲线如图1所示,由图1可知,3种药物对E. coli的t-CRC均为“S”型,说明其抑菌性有明显的时间依赖性。在0.25 h时,3种药物均无明显抑菌性;2 h时,t-CRC曲线开始出现轻微的弯曲,这表明OFL和LEV出现轻微的抑菌性,4~12 h时,t-CRC曲线开始出现很明显的弯曲,最终呈现“S”型,说明OFL和LEV在4~12 h对E. coli抑菌作用大幅增强;而HDMF在2~12 h就表现出很明显的抑菌性。在浓度方面,3种药物在2~12 h的t-CRC随着浓度的升高抑制率也在升高,说明3种药物对E. coli的抑菌性具有浓度依赖性。
2.2 2种FQs与QSIs混合体系对E. coli的毒性
2种抗生素和1种群体感应抑制剂的3组二元混合体系(OFL-HDMF、LEV-HDMF、OFL-LEV)共15条射线在不同暴露时间的毒性结果(pEC50值)见图2。
由图2可知,2~12 h时,OFL-HDMF和LEV-HDMF的毒性随时间增加均增强,说明这2组二元混合物具有时间依赖毒性,混合射线中各暴露点的毒性大小顺序为12 h-pEC50>8 h-pEC50>4 h-pEC50>2 h-pEC50。而OFL-LEV的毒性在2~8 h时随时间增加在增强,但是在12 h时,毒性略有降低,毒性大小顺序为8 h-pEC50>12 h-pEC50>4 h-pEC50>2 h-pEC50。
结合组分比(表2)来看,3种药物混合物只有OFL-LEV和LEV-HDMF具有组分比依赖毒性,对于OFL-LEV体系,随着OFL组分的减少和LEV组分的增加会使药物的抑菌性逐渐增强(R1
同样地,三元混合物体系5条射线在各暴露时间的毒性变化规律用pEC50热图展示,结果如图2所示。由图2可知,2~8 h时,三元混合物的毒性随时间的延长在增强,各暴露点的毒性顺序为2 h-pEC50<4 h-pEC50<8 h-pEC50,12 h时,三元混合物的毒性相较于8 h时略有降低,毒性大小顺序为8 h-pEC50>12 h-pEC50>4 h-pEC50>2 h-pEC50。
为了进一步探究不同暴露时间的三元混合物的毒性与组分比的相关性,用Pearson相关系数检验变量间相关性的强弱,结果如图3所示。在HDMF-OFL-LEV混合体系中,OFL和LEV pEC50值与组分浓度比之间的Pearson相关系数r<0.9,显著系数P>0.05,这说明OFL和LEV的组分比与pEC50无显著相关性,HDMF在长暴露时间(8~12 h)内表现出与pEC50存在显著负相关(r=0.91,P≤0.05);8 h-pEC50与12 h-pEC50显著正相关(r=0.97,P≤0.05),也就说明了HDMF-OFL-LEV三元混合物的毒性与组分比依赖性不是很显著。
2.3 2种FQs与QSIs混合体系毒性相互作用及强度分析
2.3.1 2种FQs与QSIs的二元混合物的联合作用类型及强度分析
2种FQs与QSIs的二元混合物的相互作用结果见图4。由图4可知LEV-HDMF、OFL-LEV和OFL-HDMF混合体系,其作用强度均随时间和浓度的变化而变化。
对于OFL-LEV混合体系,在同一浓度作用下,随着暴露时间的延长,在中、低浓度区域,表征拮抗作用的红色在逐渐变深,即拮抗作用逐渐变强;在高浓度区域,表征协同作用的蓝色由深变浅,即协同作用先变强后减弱。在同一暴露时间下,5条混合射线的红色随着OFL浓度的减小在逐渐变浅,即拮抗作用随着OFL浓度的减小在减弱。
对于LEV-HDMF混合体系,在短暴露时间(0.25~2 h)所有混合射线的dCA曲面均位于置信区间上下曲面之内;2~8 h时,dCA曲面在中、高浓度区域逐渐变成蓝色,随着浓度的变化蓝色在由深变浅,即随着时间的变化中、高浓度区域出现协同作用,随着浓度的变化协同作用在减弱;8~12 h时,dCA曲面蓝色变浅,低、中浓度区域出现红色并且在逐渐变强,随着浓度的变化红色再逐渐变浅最终消失蓝色开始出现,即随着时间的延长协同作用转变为拮抗作用,到最终节点拮抗达到最大,随着浓度的变化,拮抗再逐渐减弱最终转变成协同作用。
对于OFL-HDMF混合体系,在同一浓度作用下,随着暴露时间的延长,对于R1、R2射线,表征拮抗作用的红色在逐渐变深,即拮抗作用逐渐变强;对于R3、R4和R5射线,表征协同作用的蓝色由深变浅,即协同作用先变强后减弱。在同一暴露时间下,5条混合射线的红色随着OFL浓度的减小在逐渐变浅最终转变成蓝色,即拮抗作用随着OFL浓度的减小在变弱最终转变成协同作用。
2.3.2 3种药物的三元混合物的联合作用类型及强度分析
HDMF-OFL-LEV的dCA结果如图5所示,由图5可知,三元混合物5条射线都存在明显的协同作用,且协同作用主要集中在混合物的中、高浓度区域。其中R1~R5随着时间的延长协同作用也在增强,在8 h时协同作用达到最强,在最终暴露节点(12 h)R1、R2协同作用消失转变为加和作用,而R3、R4、R5协同作用减弱,dCA曲面在置信曲面下方。5条射线在同一暴露时间下,随浓度的改变,表征协同作用的蓝色先变深后变浅,即dCA值随着混合物浓度的变化先变大而后变成小,即协同作用先变强后减弱;在置信曲面外,5条混合射线在同一浓度作用下,蓝色在8 h达到最深,随着时间的延长到最终暴露时间(12 h)蓝色开始变浅,随着时间的延长,表征协同作用的蓝色先变深后变浅,即dCA值随着时间的增加而先增大后下降,即协同作用先变强后减弱。在三元混合物HDMF-OFL-LEV中,R4射线在8 h的协同作用最强,其混合物浓度为2.10E-2 mol·L-1,dCA绝对值的最大值为0.35。
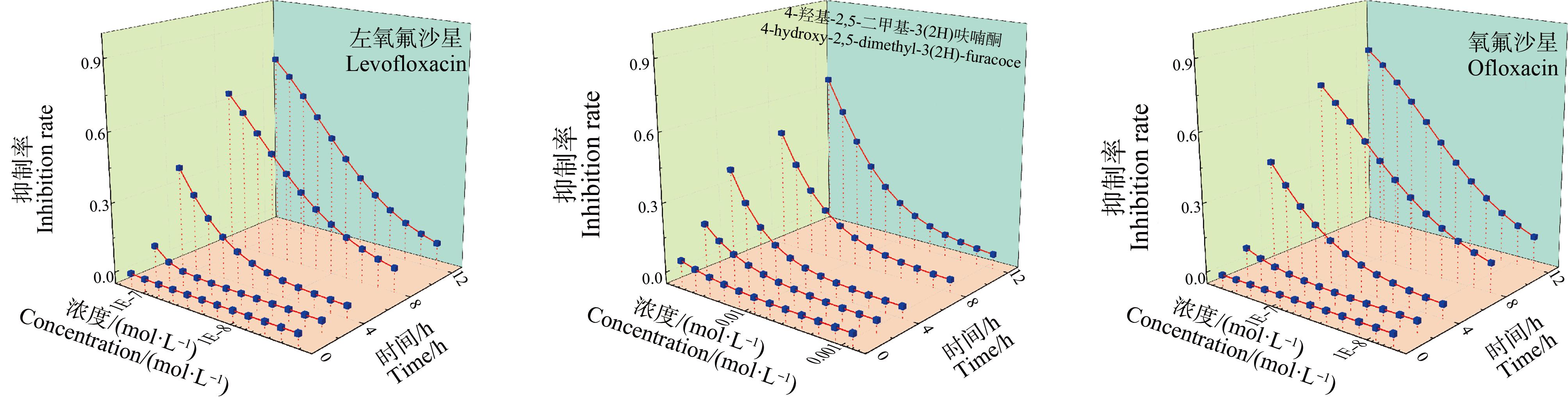
图1 3种药物对E. coli的时间-浓度-效应曲线(t-CRCs)
Fig. 1 Effect of three drugs on time-concentration-response curves (t-CRCs) of E. coli
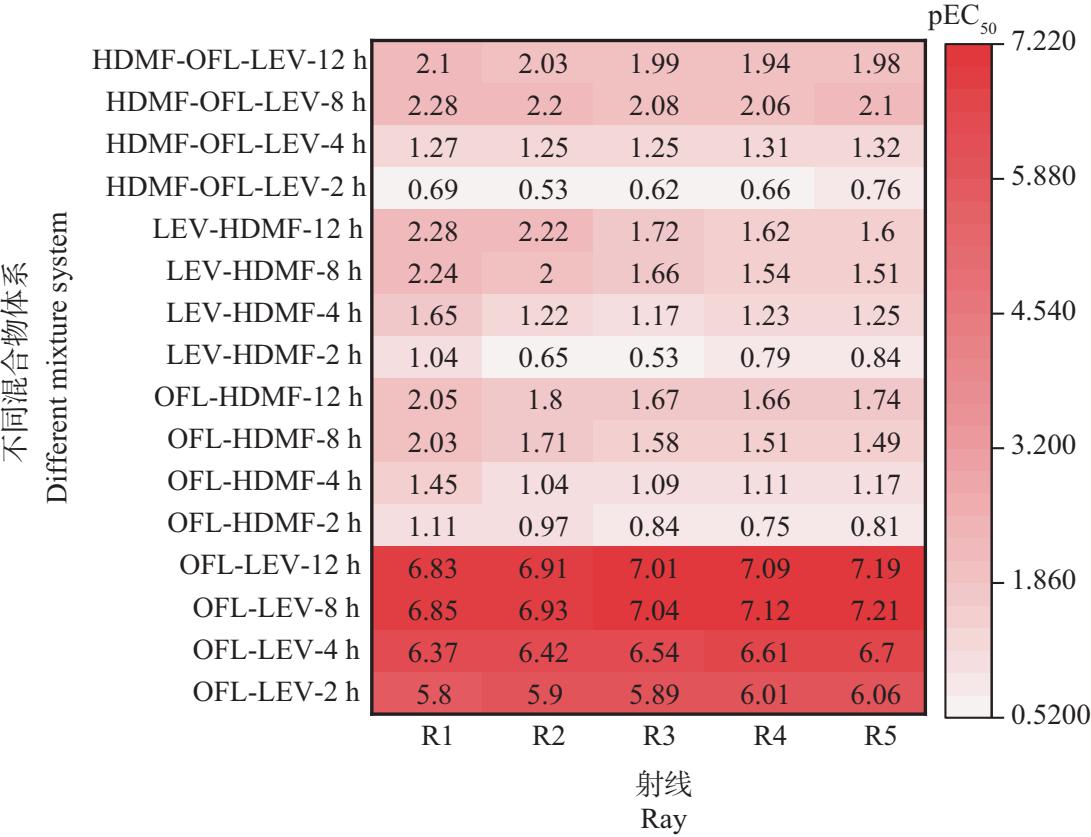
图2 3种药物混合物体系各射线pEC50随时间变化的热图
Fig. 2 Heat map of pEC50 values for each ray in the three binary mixture systems of the three drugs with time
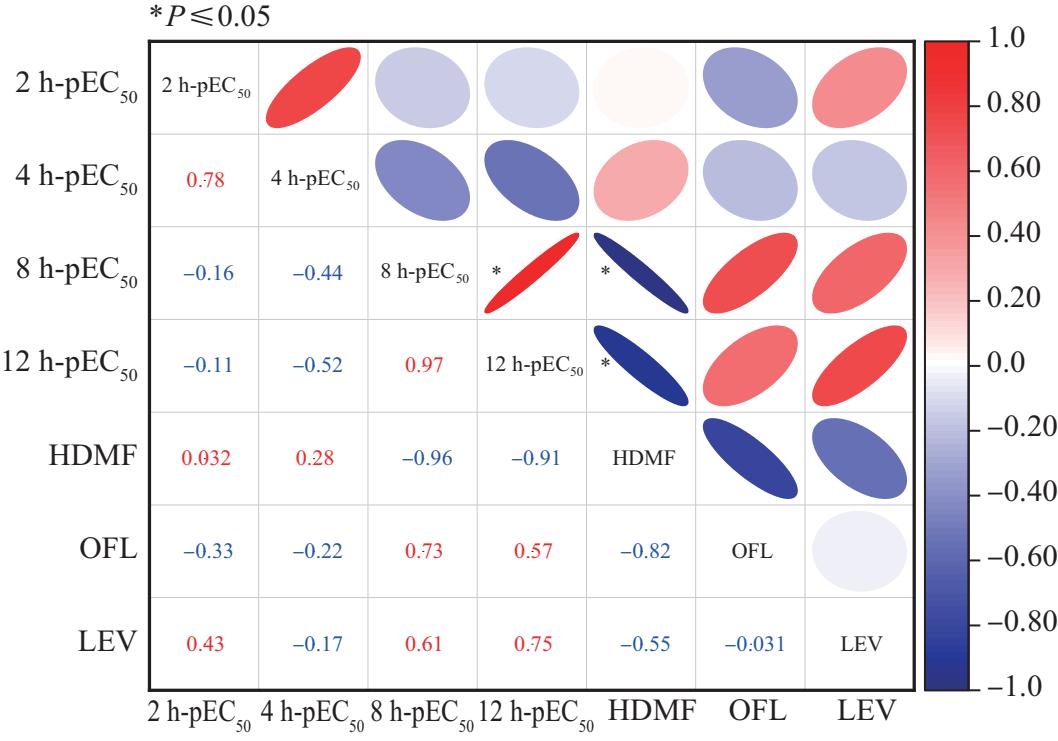
图3 三元混合物在不同暴露时间的抑菌毒性与组分比的相关性图
注:*表示显著性。
Fig. 3 Correlation between the toxicity and component ratios for ternary mixtures at different exposure times
Note: *stands for significance.

图4 3种药品二元混合物的dCA随时间变化曲面图
Fig. 4 Surface plots of dCA with time for binary mixtures of the three drugs
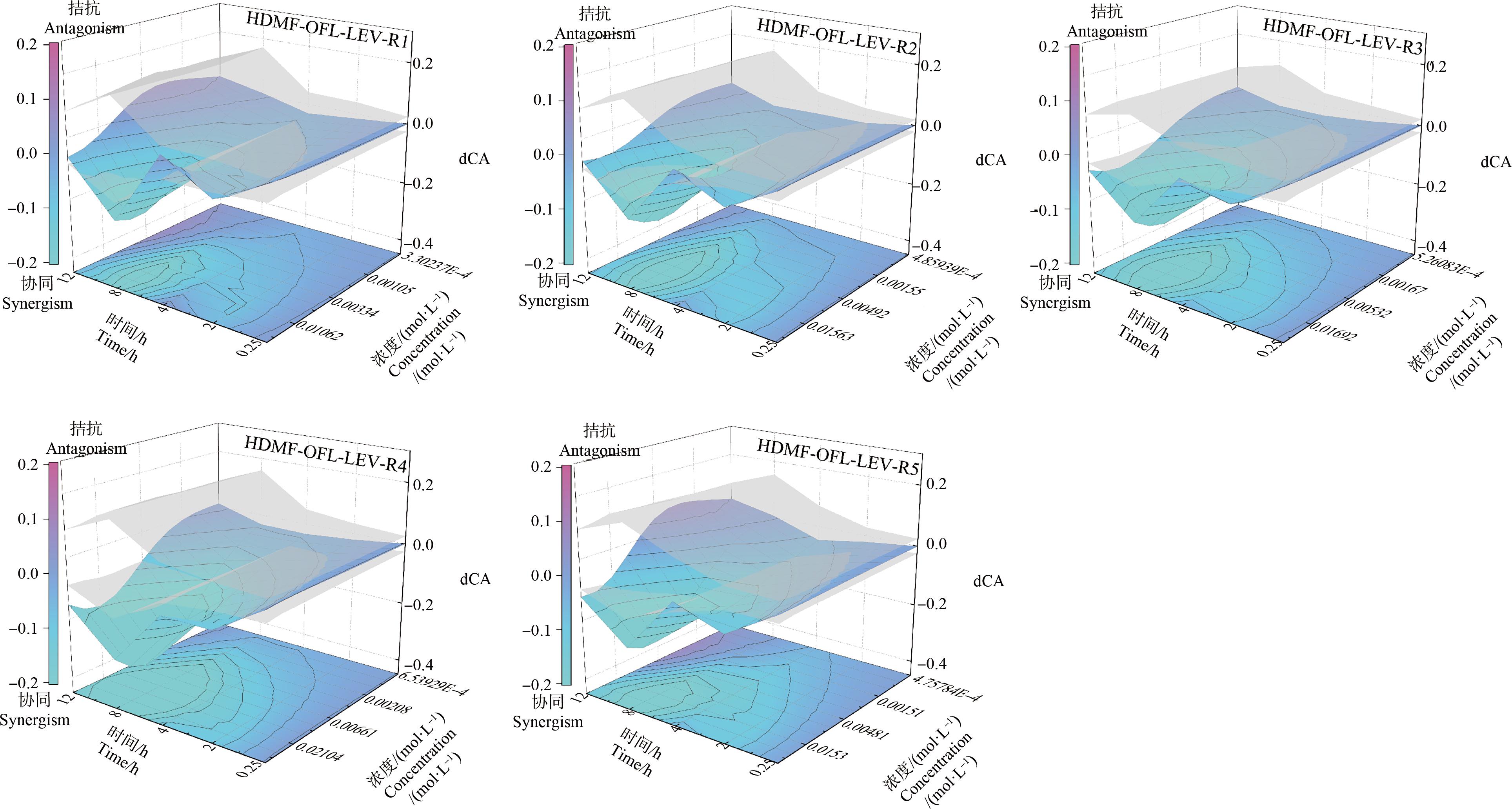
图5 HDMF-OFL-LEV三元混合物的dCA随时间变化曲面图
Fig. 5 Surface plots of dCA with time for HDMF-OFL-LEV ternary mixture
2.4 FQs和QSIs对E. coli毒性的作用机制初探
2.4.1 FQs毒性的作用机制
FQs抗生素作用机制图如图6所示,FQs抗生素通过靶向2种关键酶(DNA回旋酶Ⅱ和拓扑异构酶Ⅳ)来抑制细菌的DNA合成/复制。这些酶分离细菌DNA的双螺旋链,通过在断裂处插入另一条DNA链,使链降级[26]。DNA回旋酶将负超螺旋引入DNA链,这对于染色体凝结、缓解扭转应变和促进转录的起始具有至关重要的作用[27]。拓扑异构酶Ⅳ分解阴性和阳性的超螺旋以及结节(在重组过程中形成),并保持染色体超螺旋密度。FQs通过切割连接活性位点与切割复合物的可逆非共价结合来抑制DNA链降级,从而阻碍了这一重要过程[28]。这种相互作用最终导致氟喹诺酮-酶-DNA三元复合物的形成,阻碍了细菌DNA的复制,最终导致细菌死亡[29]。
2.4.2 QSIs毒性的作用机制
QSIs作用机制如图6所示,E. coli中的2个QS系统,分别由AI-1和AI-2信号分子调控[30]。其中E. coli不能自行合成AI-1信号分子,但是可以接受其他物种的AI-1信号分子,从而激活相关基因的表达。AI-2分子可由LuxR蛋白产生并在细菌中积累,当细胞外的AI-2浓度达到阈值时,分子将通过LsrACBD转运回细菌,随后被LsrK磷酸化。磷酸化的AI-2可以与LsrR结合,开启相关基因的表达[31],这些基因中的大多数具有生物膜调控[32]、运动性和鞭毛合成的功能[33]。QSIs作为信号分子的结构类似物,可以分别与信号分子竞争结合SdiA和LsrR,从而干扰E. coli的QS系统,最终表现出对E. coli的抑制作用[34]。
2.4.3 FQs和QSIs联合毒性的作用机制
由图6和图7可知,FQs和QSIs的二元混合物中OFL和LEV能与DNA拓扑异构酶Ⅳ(PDB ID:3FV5)更好地结合,2种药物在键位的形成上具有高度相似性,例如LEV和SER43、ASP69形成氢键,OFL则和ASN42形成氢键,而SER43和ASN42两者在蛋白质链中的位置相邻,LEV和ARG72、GLY73形成卤素键而OFL同样也和ARG72、GLY73形成卤素键,过于相似的结合点位会造成2种药物的竞争结合。在暴露前期,OFL和LEV能够快速渗透细胞膜进入细胞内部[35],高浓度的抗生素会迅速杀死E. coli,进而造成高浓度区域微弱的协同作用。随着时间的延长,存活下来的E. coli在FQs引起的DNA损伤下可能初始化SOS反应,SOS反应能抵消和修复DNA损伤,同时也能通过诱导靶基因(gyrA)突变进一步触发FQs耐药性[36],另外,OFL和LEV会竞争结合蛋白质上的点位,这也许是OFL和LEV在最终时间节点形成拮抗作用的原因。
QSIs进入细胞内部后会和AI-1和AI-2竞争结合SdiA和LsrR,而AI-2在E. coli生物膜形成中起主要作用,它能介导细菌向生长中的细菌聚集体趋化,从而增强E. coli菌落的形成,随后在群落中形成生物膜[37],由于QSIs的竞争结合,细菌的生物膜无法有效形成,FQs更容易接触细菌造成损伤,这可能是暴露初期形成协同作用的原因。随着时间的延长,进入细菌内的FQs会对DNA造成损伤,这可能会减少SdiA和LsrR蛋白的表达,缺乏靶蛋白会减轻QSIs对QS系统的抑制,另外,FQs在造成DNA损伤的同时还会刺激细菌活性氧(ROS)的产生[38],大量的ROS产生可能会攻击核苷酸的碱基部分和糖磷酸骨架,从而产生双螺旋的单链和双链断裂[39-41],以此为对应,细菌会紧急激活容易出错的SOS反应以抵消和修复DNA损伤,而QSIs与LsrR蛋白的结合同样也会增加SOS基因表达[42],进一步刺激细菌修复自身。因此FQs和QSIs在联合抑制E. coli生长时表现出拮抗作用。OFL作为LEV的同分异构体,其最终时间节点的相互作用关系与LEV的相互关系相似,有区别的是相互作用的强度,由分子对接的结合能可知LEV的结合能(32.635 kJ·mol-1)要高于OFL的结合能(30.543 kJ·mol-1),也就是说LEV要比OFL更容易和作用靶蛋白结合,这有可能是LEV-HDMF体系相互作用强于OFL-HDMF体系相互作用的原因。
三元混合物中,起主要的抑制作用的是FQs,因为3种抗菌药物各自发挥抑菌作用的情况下,FQs能直接抑制细菌生长,而QS系统不直接参与蛋白质合成、DNA复制和修复以及细胞壁合成等基本细胞过程。因此与二元联合毒性作用相比,三元联合毒性作用模式没有发生改变,但二元混合物的相互作用由拮抗/加和作用转变成三元混合物的协同/加和作用,这可能与混合物的混合比和浓度水平有关。
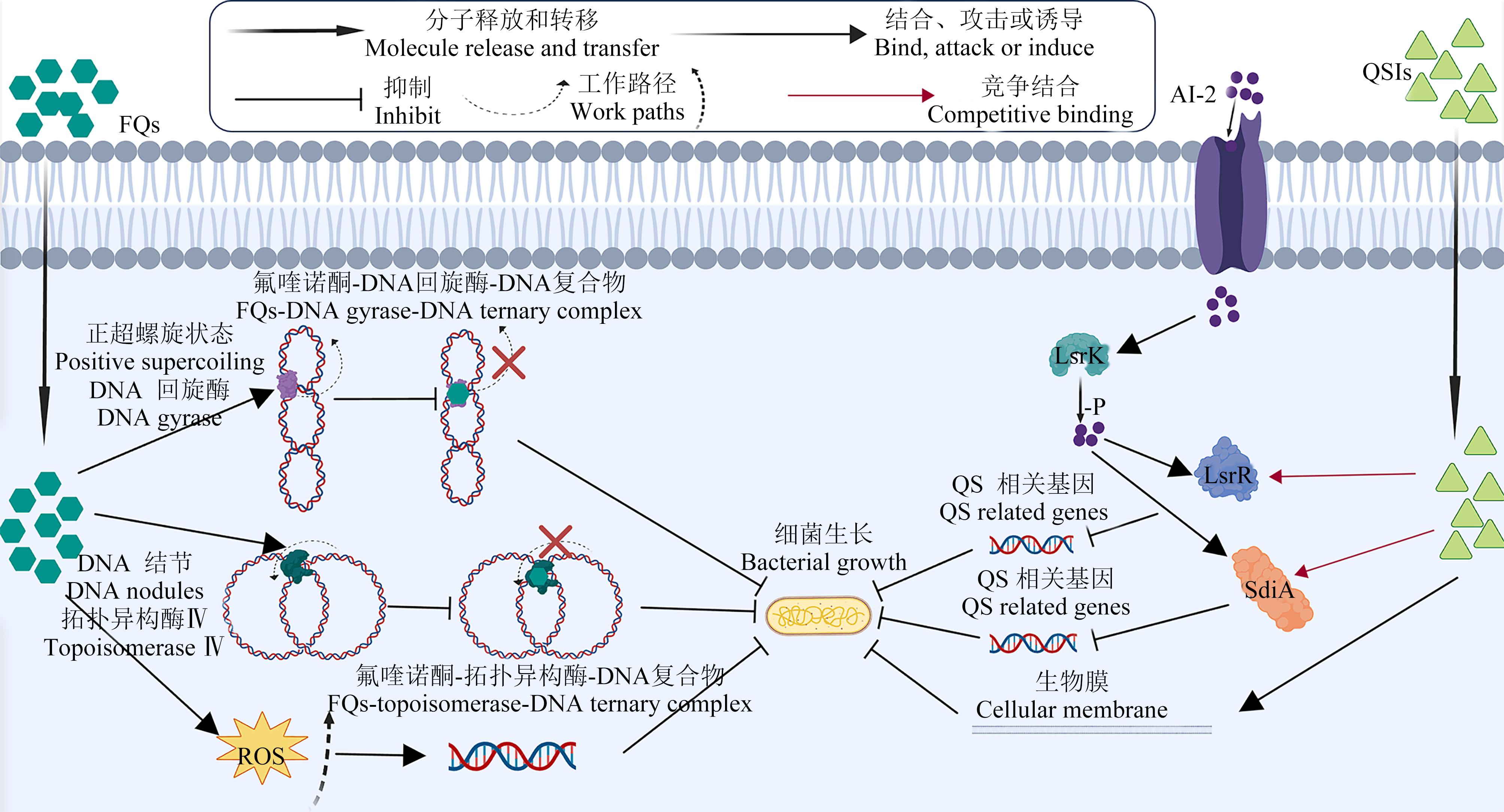
图6 2种药物作用于E. coli的机理图
Fig. 6 Mechanism diagram of two drugs acting on E. coli
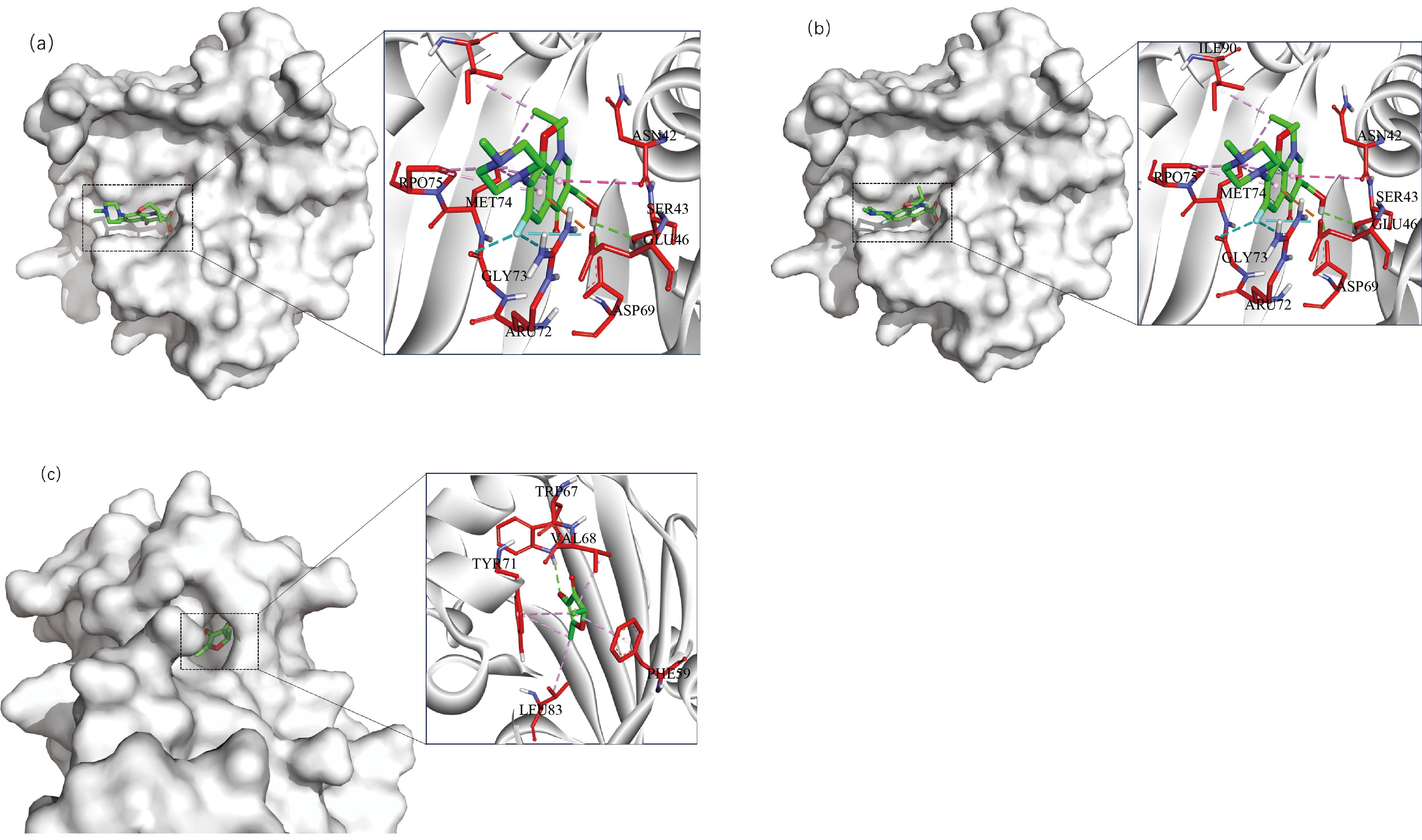
图7 蛋白结构及其与化合物的分子对接图
注:(a) 蛋白与左氧氟沙星(levofloxacin)对接模型;(b) 蛋白与氧氟沙星(ofloxacin)对接模型;(c) SdiA蛋白与4-羟基-2,5-二甲基-3(2H)呋 喃酮(HDMF)对接模型;图中绿色虚线表示氢键相互作用,蓝色虚线表示卤素(氟)相互作用,粉色虚线表示Pi-烷基相互作用。
Fig. 7 Protein structure and its macromolecular docking diagram with compounds
Note: (a) Protein and levofloxacin docking model; (b) Protein and ofloxacin docking model; (c) SdiA protein and 4-hydroxy-2,5-dimethyl-3(2H)-furanone docking model; the hydrogen-bonding interactions are shown in green dashed lines, halogen (fluorine) interactions in blue dashed lines and Pi-Alkyl interactions in pink dashed lines in the figure.
综上所述:(1)3种药物对E. coli毒性表现出较好的浓度-效应关系,对E. coli具有浓度依赖毒性和时间依赖毒性。3种药物不同暴露时间毒性大小不同,毒性大小顺序:LEV(pEC50=7.35)>OFL(pEC50=7.06)>HDMF(pEC50=1.42)。
(2)3种药物的二元混合体系中OFL-HDMF和LEV-HDMF具有时间依赖毒性,OFL-LEV和LEV-HDMF具有组分比依赖毒性,三元体系中毒性的时间、组分比依赖性不明显。
(3)3种药物的二元、三元混合物体系对E. coli的相互作用主要表现为协同/拮抗作用,并且随着暴露时间和浓度不同,协同/拮抗作用、强度也不同,其中HDMF-OFL-LEV-R4射线的协同作用最明显,dCA绝对值为0.31。
(4)混合体系中OFL和LEV产生拮抗的原因是2种药物竞争结合蛋白点位,而LEV-HDMF体系中LEV对DNA的作用会损伤SdiA和LsrR蛋白的表达进而产生2种药物拮抗的原因;LEV作为OFL的同分异构体,其与相应受体蛋白结合能力要强于OFL,这有可能是LEV-HDMF体系相互作用强于OFL-HDMF体系相互作用的原因。
[1] Sodhi K K, Singh D K. Insight into the fluoroquinolone resistance, sources, ecotoxicity, and degradation with special emphasis on ciprofloxacin [J]. Journal of Water Process Engineering, 2021, 43: 102218
[2] Zhi S, Zhou J, Yang F, et al. Systematic analysis of occurrence and variation tendency about 58 typical veterinary antibiotics during animal wastewater disposal processes in Tianjin, China [J]. Ecotoxicology and Environmental Safety, 2018, 165: 376-385
[3] Hamad B. The antibiotics market [J]. Nature Reviews Drug Discovery, 2010, 9(9): 675-676
[4] Van Doorslaer X, Dewulf J, Van Langenhove H, et al. Fluoroquinolone antibiotics: An emerging class of environmental micropollutants [J]. Science of the Total Environment, 2014, 500-501: 250-269
[5] 张静, 张瑾, 宋崇崇, 等. 3种四环素类抗生素对绿藻联合毒性作用评估[J]. 环境科学与技术, 2023, 46(4): 1-10
Zhang J, Zhang J, Song C C, et al. Assessment of the combined toxic effects of three tetracycline antibiotics on green algae [J]. Environmental Science &Technology, 2023, 46(4): 1-10 (in Chinese)
[6] Kalia V C. Quorum sensing inhibitors: An overview [J]. Biotechnology Advances, 2013, 31(2): 224-245
[7] Bhardwaj S, Bhatia S, Singh S, et al. Growing emergence of drug-resistant Pseudomonas aeruginosa and attenuation of its virulence using quorum sensing inhibitors: A critical review [J]. Iranian Journal of Basic Medical Sciences, 2021, 24(6): 699-719
[8] O’Loughlin C T, Miller L C, Siryaporn A, et al. A quorum-sensing inhibitor blocks Pseudomonas aeruginosa virulence and biofilm formation [J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(44): 17981-17986
[9] Mahdally N H, George R F, Kashef M T, et al. Staquorsin: A novel Staphylococcus aureus agr-mediated quorum sensing inhibitor impairing virulence in vivo without notable resistance development [J]. Frontiers in Microbiology, 2021, 12: 700494
[10] Khayyat A N, Hegazy W A H, Shaldam M A, et al. Xylitol inhibits growth and blocks virulence in Serratia marcescens [J]. Microorganisms, 2021, 9(5): 1083
[11] Abbas H A, Hegazy W A H. Repurposing anti-diabetic drug Sitagliptin as a novel virulence attenuating agent in Serratia marcescens [J]. PLoS One, 2020, 15(4): e0231625
[12] Roy V, Meyer M T, Smith J A, et al. AI-2 analogs and antibiotics: A synergistic approach to reduce bacterial biofilms [J]. Applied Microbiology and Biotechnology, 2013, 97(6): 2627-2638
[13] Vadekeetil A, Alexandar V, Chhibber S, et al. Adjuvant effect of cranberry proanthocyanidin active fraction on antivirulent property of ciprofloxacin against Pseudomonas aeruginosa [J]. Microbial Pathogenesis, 2016, 90: 98-103
[14] Li J Q, Wei X F, Song Y S, et al. In vitro study of the effect of inhibition of quorum sensing by brominated furanone on peritoneal dialysis-associated peritonitis associated with Escherichia coli infection [J]. Current Microbiology, 2022, 79(11): 337
[15] Brackman G, Cos P, Maes L, et al. Quorum sensing inhibitors increase the susceptibility of bacterial biofilms to antibiotics in vitro and in vivo [J]. Antimicrobial Agents and Chemotherapy, 2011, 55(6): 2655-2661
[16] Shukla A, Parmar P, Patel B, et al. Breaking bad: Better call gingerol for improving antibiotic susceptibility of Pseudomonas aeruginosa by inhibiting multiple quorum sensing pathways [J]. Microbiological Research, 2021, 252: 126863
[17] 骆纵纵, 张瑾, 周娜娜, 等. 3种青霉素类抗生素对大肠杆菌的时间毒性微板分析法建立及其联合抑菌作用[J]. 生态毒理学报, 2022, 17(2): 189-199
Luo Z Z, Zhang J, Zhou N N, et al. Establishment of time-dependent microplate toxicity analysis method for combined antibacterial effects of three penicillin antibiotics to Escherichia coli [J]. Asian Journal of Ecotoxicology, 2022, 17(2): 189-199 (in Chinese)
[18] 曹家乐, 张瑾, 杜士林, 等. 不同pH值对铜和硫酸阿米卡星的青海弧菌时间依赖毒性相互作用的影响[J]. 环境化学, 2022, 41(10): 3378-3389
Cao J L, Zhang J, Du S L, et al. Effects of different pH values on the time-dependent toxic interactions of copper and Amikacin sulfate in Vibrio qinghaiensis sp.-Q67 [J]. Environmental Chemistry, 2022, 41(10): 3378-3389 (in Chinese)
[19] Dou R N, Liu S S, Mo L Y, et al. A novel direct equipartition ray design (EquRay) procedure for toxicity interaction between ionic liquid and dichlorvos [J]. Environmental Science and Pollution Research International, 2011, 18(5): 734-742
[20] Liu S S, Xiao Q F, Zhang J, et al. Uniform design ray in the assessment of combined toxicities of multi-component mixtures [J]. Science Bulletin, 2016, 61(1): 52-58
[21] 骆纵纵, 张瑾, 周娜娜, 等. 3种青霉素类抗生素对大肠杆菌的时间毒性微板分析法建立及其联合抑菌作用[J]. 生态毒理学报, 2022, 17(2): 189-199
Luo Z Z, Zhang J, Zhou N N, et al. Establishment of time-dependent microplate toxicity analysis method for combined antibacterial effects of three penicillin antibiotics to Escherichia coli [J]. Asian Journal of Ecotoxicology, 2022, 17(2): 189-199 (in Chinese)
[22] 刘树深, 张瑾, 张亚辉, 等. APTox: 化学混合物毒性评估与预测[J]. 化学学报, 2012, 70(14): 1511-1517
Liu S S, Zhang J, Zhang Y H, et al. APTox: Assessment and prediction on toxicity of chemical mixtures [J]. Acta Chimica Sinica, 2012, 70(14): 1511-1517 (in Chinese)
[23] 刘树深. 化学混合物毒性评估与预测方法[M]. 北京: 科学出版社, 2017: 57-68
[24] Trott O, Olson A J. AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading [J]. Journal of Computational Chemistry, 2010, 31(2): 455-461
[25] Ning Q, Wang D L, You J. Joint effects of antibiotics and quorum sensing inhibitors on resistance development in bacteria [J]. Environmental Science Processes &Impacts, 2021, 23(7): 995-1005
[26] Jin M K, Zhang Q, Zhao W L, et al. Fluoroquinolone antibiotics disturb the defense system, gut microbiome, and antibiotic resistance genes of Enchytraeus crypticus [J]. Journal of Hazardous Materials, 2022, 424(Pt C): 127509
[27] Blondeau J M. Fluoroquinolones: Mechanism of action, classification, and development of resistance [J]. Survey of Ophthalmology, 2004, 49(2): S73-S78
[28] Correia S, Poeta P, Hébraud M, et al. Mechanisms of quinolone action and resistance: Where do we stand? [J]. Journal of Medical Microbiology, 2017, 66(5): 551-559
[29] Kohanski M A, Dwyer D J, Collins J J. How antibiotics kill bacteria: From targets to networks [J]. Nature Reviews Microbiology, 2010, 8(6): 423-435
[30] Wang Y S, Bian Z R, Wang Y. Biofilm formation and inhibition mediated by bacterial quorum sensing [J]. Applied Microbiology and Biotechnology, 2022, 106(19-20): 6365-6381
[31] Rasmussen T B, Givskov M. Quorum-sensing inhibitors as anti-pathogenic drugs [J]. International Journal of Medical Microbiology, 2006, 296(2-3): 149-161
[32] Reen F J, Gutiérrez-Barranquero J A, Parages M L, et al. Coumarin: A novel player in microbial quorum sensing and biofilm formation inhibition [J]. Applied Microbiology and Biotechnology, 2018, 102(5): 2063-2073
[33] Ren D C, Bedzyk L A, Ye R W, et al. Differential gene expression shows natural brominated furanones interfere with the autoinducer-2 bacterial signaling system of Escherichia coli [J]. Biotechnology and Bioengineering, 2004, 88(5): 630-642
[34] Li Q Q, Mao S, Wang H, et al. The molecular architecture of Pseudomonas aeruginosa quorum-sensing inhibitors [J]. Marine Drugs, 2022, 20(8): 488
[35] Taghavi Z, Hassanshahian M, Hassanshahi G. Study of antimicrobial effect of a new fluoroquinolone derivative against pathogenic bacteria in planktonic form and biofilm [J]. Proceedings of the National Academy of Sciences, India Section B: Biological Sciences, 2023, 93(1): 137-145
[36] Liang H B, Zhang J Y, Hu J H, et al. Fluoroquinolone residues in the environment rapidly induce heritable fluoroquinolone resistance in Escherichia coli [J]. Environmental Science &Technology, 2023, 57(12): 4784-4795
[37] Laganenka L, Sourjik V. Autoinducer 2-dependent Escherichia coli biofilm formation is enhanced in a dual-species coculture [J]. Applied and Environmental Microbiology, 2018, 84(5): e02638-e02617
[38] Goswami M, Mangoli S H, Jawali N. Involvement of reactive oxygen species in the action of ciprofloxacin against Escherichia coli [J]. Antimicrobial Agents and Chemotherapy, 2006, 50(3): 949-954
[39] Memar M Y, Yekani M, Celenza G, et al. The central role of the SOS DNA repair system in antibiotics resistance: A new target for a new infectious treatment strategy [J]. Life Sciences, 2020, 262: 118562
[40] Cabiscol E, Tamarit J, Ros J. Oxidative stress in bacteria and protein damage by reactive oxygen species [J]. International Microbiology: The Official Journal of the Spanish Society for Microbiology, 2000, 3(1): 3-8
[41] Dwyer D J, Kohanski M A, Collins J J. Role of reactive oxygen species in antibiotic action and resistance [J]. Current Opinion in Microbiology, 2009, 12(5): 482-489
[42] Li X F, Liu Y Y, Wang Y J, et al. Resistance risk induced by quorum sensing inhibitors and their combined use with antibiotics: Mechanism and its relationship with toxicity [J]. Chemosphere, 2021, 265: 129153