-
含氮有机污染物按照含氮官能团种类可分为氨基类、硝基类、偶氮类、杂环氮类以及包含上述不同基团的复合类[1-2]。其中,部分含氮有机污染物不仅母体化合物具有较高毒性、其分解的中间产物毒性甚至超过母体化合物,如5-氯-2-甲基苯胺及其中间产物3-氯苯胺都具有三致效应与遗传毒性[3];喹啉在经过氧化处理后产生的中间产物如吲哚、吡啶等对硝化和反硝化细菌以及四膜虫DNA产生的毒性高于喹啉自身对其的毒性[4]。近年来,以磺胺二甲基嘧啶为例的抗生素类含氮有机污染物已在黄浦江中被检测出来(2.05—623.27 ng·L−1),若处理不当,会增强细菌的抗性基因并对生态系统和人类健康构成严重威胁[5]。因此对于废水中含氮有机污染物的去除显得至关重要。
传统的水处理技术包括物理法、生物法、化学法以及它们的组合法。然而,这些方法存在易产生二次污染、处理效果不稳定、效率低和成本高等问题[6-7],而超临界水氧化技术(SCWO)作为高级氧化的一种特殊方式(压力>22.1 MPa,温度>374.3 ℃),其具备去除效率高、耗时短、反应速率快等特点。近年来,SCWO技术已被广泛应用于水体中含氮有机污染物的去除。如Shin等采用SCWO技术考察了丙烯腈工厂废水的去除效果,结果表明,在温度552 ℃、压力25 MPa和250%过氧量条件下,丙烯腈的去除效率15 s即可达到97%[8];Liu等研究吲哚时发现,在碳酸钾催化作用下,经过30 min,目标物中碳、氮元素的转化率在750 ℃、反应时间为30 min时可达到100%与80%[9]。因此,超临界水氧化技术(SCWO)被认为是去除水体中有机污染物的有效措施。然而,由于SCWO过程中需要高温、高压条件,且在使用过程中存在易腐蚀、易堵塞等现象,导致SCWO在使用过程中存在成本高、能源消耗大等问题。因此,利用模型预测含氮有机污染物在SCWO中的降解效果和规律对于评估含氮有机污染物在SCWO体系中的去除不仅可以节约成本,同时可为其他有机污染物的去除提供理论指导。
定量构效关系(QSAR)模型近年来已广泛应用于揭示有机污染物的降解规律以及预测有机污染物的降解行为。其基本思想是通过有机物分子结构信息预测有机污染物的环境行为,为新生或缺乏实验数据的有机污染物的环境行为提供初始评估[10-11]。如Jia等对Fenton氧化体系中的反应速率常数进行了QSAR建模,结果显示反应速率常数与表面积、福井指数以及前线轨道能密切相关[12]。Zhu等建立了在酸性条件和中性条件下具有统计学意义的臭氧氧化有机污染物的反应速率常数的QSAR模型,模型结果显示臭氧体系氧化速率常数与福井指数关系密切[13-14]。Luo等成功建立了526种有机污染物在•OH氧化条件下的QSAR模型,并展现出良好的预测效果[15]。Xiao等对85个痕量级有机污染物在硫酸自由基氧化条件下建立了最优QSAR模型,发现分子中氧原子与碳原子个数比以及分子前线轨道能在硫酸自由基氧化过程中占据主导地位[16]。然而,对于含氮有机污染物去除效果的QSAR模型却鲜有研究,因此,建立含氮有机污染物在SCWO中去除效果的QSAR模型,对于评估含氮有机污染物的去除效果和研究其去除规律均具有重要意义。
本研究以41种含氮有机污染物在SCWO中去除效果为研究对象,采用多元线性回归法建立去除效果与有机污染物分子量子化学参数之间的构效关系,并结合QSAR模型验证方法对所建模型进行评估和检验,通过模型中量子化学参数解释含氮有机污染物在SCWO中去除规律,旨在为含氮有机污染物的在SCWO中的去除提供初始评估和理论指导。
-
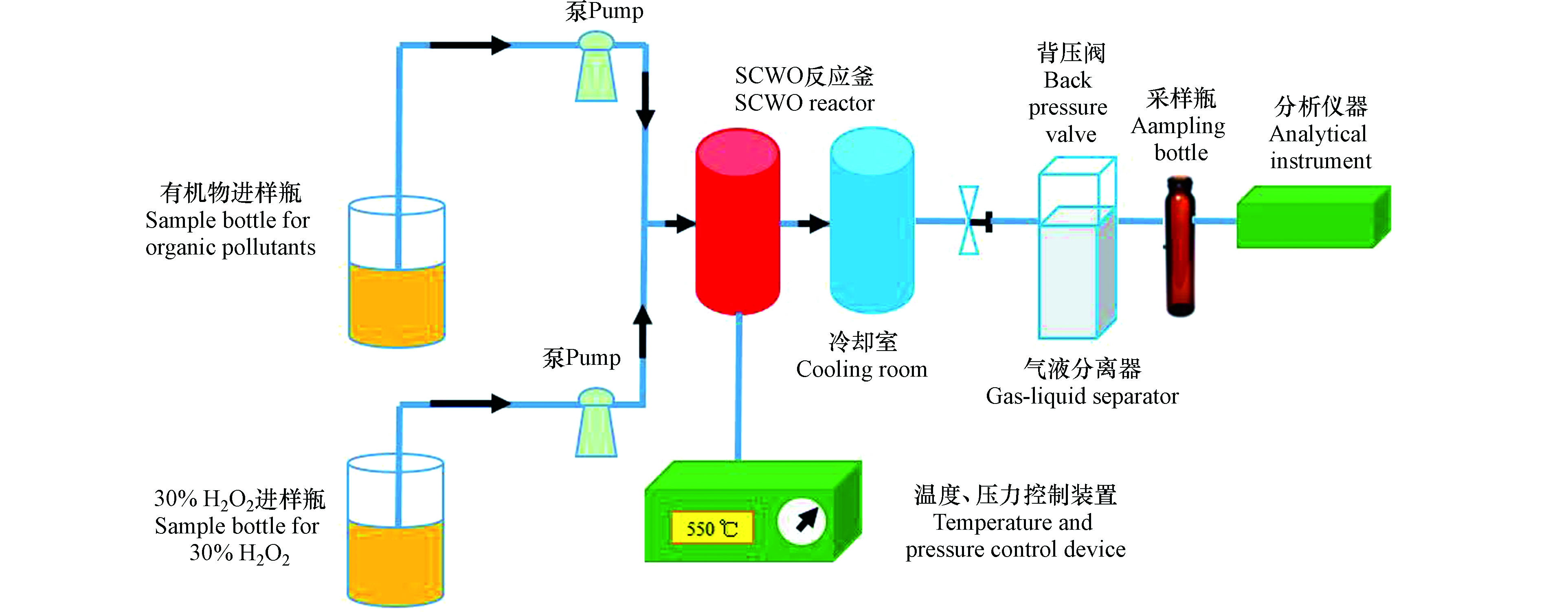
本实验搭建的超临界氧化装置(图1)包括:有机物和氧化剂进样装置、平流泵、温度与压力控制装置、反应釜、冷却室、背压阀、气液分离器、采样系统及分析系统。其中,反应釜由内径2.5 mm、长度2 m的哈氏合金构成,容积、温度与压力上限分别为10 mL、600 ℃与40 MPa。首先,分别将已充分溶解的目标有机物和氧化剂通过两台平流泵注入反应装置;然后,为了探索所选有机污染物在超临界氧化体系中的脱氮效果,同时保证反应釜的稳定与安全性,反应釜通过温控装置加热至反应温度(通过热电偶监测)550 ℃;随后,从反应釜流出的尾液通过冷却水室冷却至室温,再经由气液分离装置进行气液分离;最后,尾液则由40 mL玻璃试管收集待测。此外,整个反应系统在实验前需经过30 min预热,以期达到温度、压力平衡。
由于个别目标有机污染物含有氯离子和硫酸根离子会导致超临界水氧化系统中的反应釜受腐蚀,故此实验中目标有机污染物的初始浓度为1 mmol·L−1。同样地,将配制好的目标有机污染物溶液置于2 L烧杯中备用。待超临界水氧化设备预热30 min后,将温度和压力调节至550 ℃和24 MPa。将目标有机污染物和氧化剂(30%双氧水)同时通过泵打入反应系统。此实验的采样时间点分别为0.5、1、1.5、2、2.5、3 min。各时间点样品经过固液分离系统后将液体收集进行下一步测定分析。
-
总氮(TN)采用德国耶拿公司的multi N/C-3100仪器进行分析测定,分别对有机污染物的初始浓度和经SCWO后出水浓度进行总氮测定,经过计算,得出TN去除效率(注:亚硝态氮和NOx在全过程中并未检出,因此,本研究中的总氮包括有机氮和无机氮中的氨氮、硝态氮和氮气含量),41种有机污染物的TN去除率见表1,其计算公式如下:
其中,
$ {\mathrm{T}\mathrm{N}}_{0} $ 为有机污染物初始TN浓度,$ {\mathrm{T}\mathrm{N}}_{t} $ 为反应时间t时刻出水的TN浓度,$ \mathrm{T}\mathrm{N}\mathrm{\%} $ 为总氮去除效率。 -
本试验采用Gaussian 09[17]和Material Studio 7.0[18]两款软件对含氮化合物进行分子结构优化与量子化学计算,并得到目标物的相关量化参数,再结合TN去除效率,采用SPSS 22.0中多元线性回归法得到含氮化合物的
$ \mathrm{T}\mathrm{N}\mathrm{\%} $ 与量化参数的QSAR模型。其中, 41种有机污染物的各量子化学参数具体数值见表1,所选取的量子化学参数及其释义见表2。 -
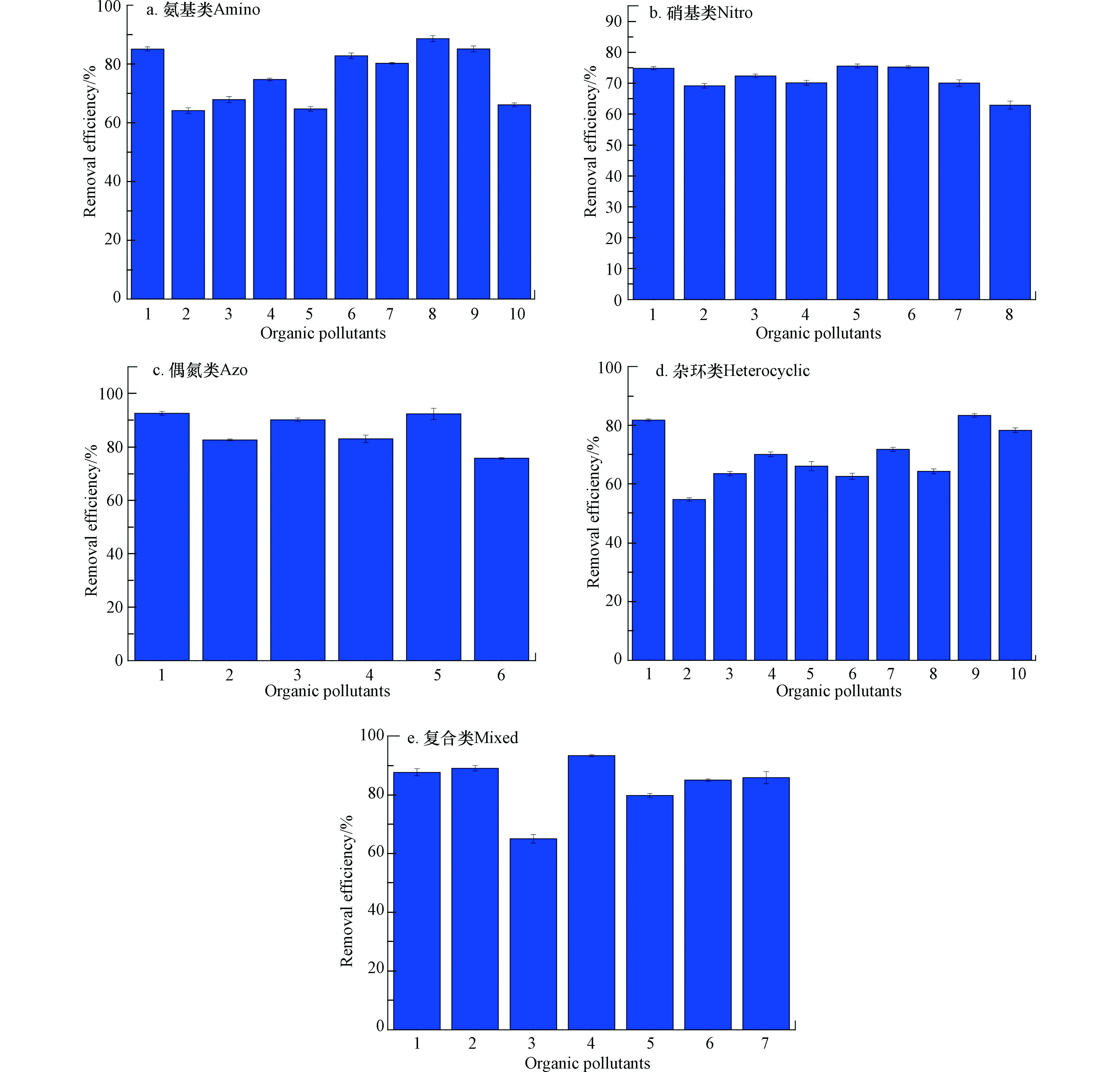
图2为41种含氮化合物的TN去除率图,可以看出当反应温度为550 ℃时,大部分物质的TN去除率达到60%以上,表明在该条件下SCWO可以有效地对所选含氮有机物进行脱氮。上述结果与Pinto等[19]的研究结果相似,其在温度525 ℃和压力25 MPa下,喹啉在SCWO中TN去除率达到了50%[19]。同时,图2也显示出不同的含氮化合物的不同TN去除率,这种差异源于两点:一是因为化合物自身的氧化等级,由于SCWO会产生剧烈的氧化环境,这使得原本就带有O元素的基团,如硝基(-NO2),难以被还原;其二可能与含氮产物,如铵根离子、硝酸根离子等分布相关。其中,总氮去除率最高的物质是邻硝基苯胺(93.324%),最低的物质是三聚氰酸(54.746%),41中物质平均去除率为76.030%±10.746%。
-
QSAR模型的建立采用多元线性回归法中的逐步回归进行建模。首先,将41个有机物的TN去除率按照训练集与测试集样本数量比近似4:1的要求分为训练集和测试集,同时保证所选的测试集均匀分布在整体数据集之中,且保证测试集化合物包含5个不同种类。本研究中,选择表1中*标记的化合物为测试集化合物,随后以训练集中TN%为因变量,19种量子化学参数为自变量进行QSAR建模,其结果见表3。模型的验证采用了模型的可决系数(
$ {R}^{2} $ )、标准差(SD)、均方根误差(RMSE)、显著性检验(Sig.)、Fisher检验(F检验)、留一法内部验证($ {q}^{2} $ )、外部验证($ {Q}_{\mathrm{e}\mathrm{x}\mathrm{t}}^{2} $ )、t检验、变量共线性检验(VIF)以及Y随机检验。当且仅当模型的检验指数满足以下标准时,方可认为模型具备良好的稳定性和较高的预测能力,即(1)$ {R}^{2} $ 大于0.6;(2)显著性检验小于0.01;(3)$ {q}^{2} $ 和$ {Q}_{ext}^{2} $ 均大于0.5;(4)F值大于F表中对应的临界值;(5)模型中各自变量VIF值大于1且小于5。根据逐步回归法依次选取不同变量进行先行拟合,直到模型具备良好的统计学结果,建模过程中,尽量保证用最少的自变量解释因变量,基于此原则,建立了包含4个自变量的最优QSAR模型,同时为了对比说明,表3列出了从1个自变量到4个自变量的QSAR模型及其统计学参数。如表3所示,4个QSAR模型的可决系数
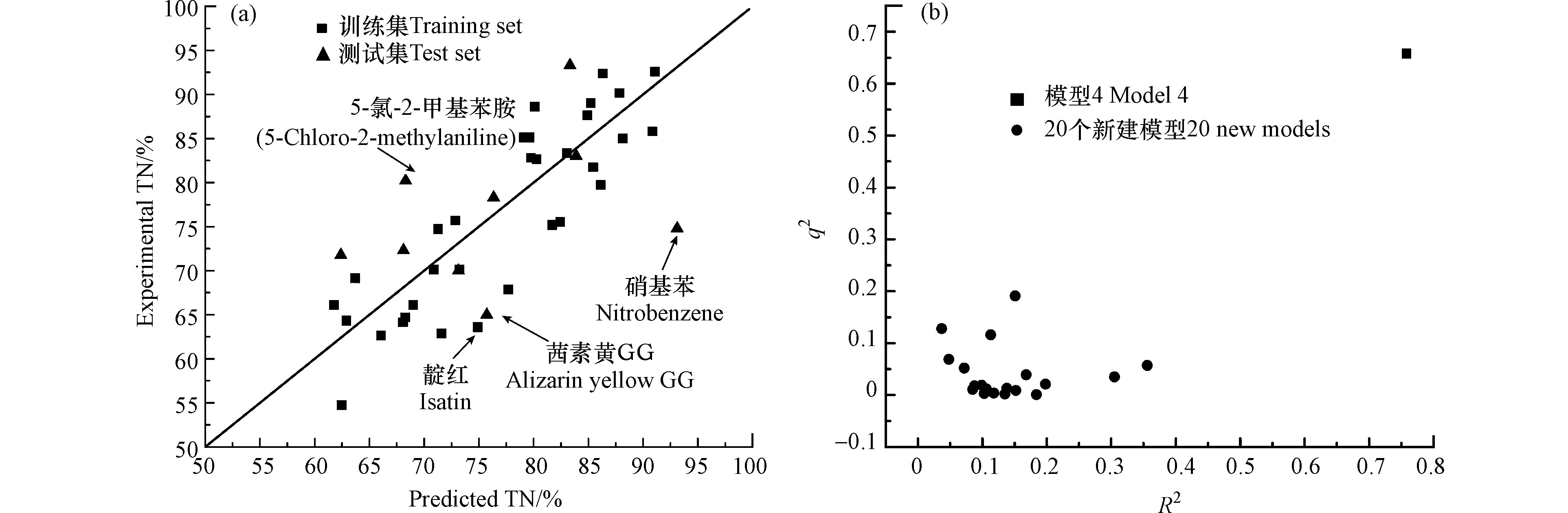
$ {R}^{2} $ 随着模型中自变量的数量增加而增加,其中模型4具有最高的$ {R}^{2} $ 值(0.701)。模型的标准差SD以及RMSE随着模型中自变量数量的增加而减小,说明模型引入的自变量数量越多,其对训练集中TN%值的预测越准。模型的F检验值均大于其检验的临界值2.53,而显著性检验结果均小于0.01,说明模型中各自变量之间差异性显著。综合上述验证标准,4个模型中仅有模型4满足所有验证标准,因此,模型4被认为是含氮有机污染物在SCWO的总氮去除率的最优QSAR模型。模型4对41种含氮有机物的TN%值预测值与实验值的对比图见图3(a),如图所示,黑色点代表32种训练集化合物,红色点表示9种测试集化合物,黑色实线表示回归线1∶1,图中数据点越接近该回归线,表明模型对TN%预测值越接近实验值。图中除了靛红、茜素黄GG、5-氯-2-甲基苯胺和天青B硝基苯外,其他各点均能较好地分布在回归线周围,表明该模型对于其他37种含氮有机污染物的预测较准确。而偶氮胂1、吡啶、铬黑T和天青B的预测值与实验值之差分别为11.303%、10.720%、−11.927%和18.305%。此外,从图中可以看出测试集的选择能够较均匀的分布在整个数据集之中,说明测试集的选择是合理的。图3(b)为模型4经过20次循环的Y随机验证图,图中红色点代表本章所建立的最优QSAR模型的$ {R}^{2} $ 和$ {q}^{2} $ 值,圆点代表经过20次Y随机检验的$ {R}^{2} $ 和$ {q}^{2} $ 值,从中可以发现所有新建模型中的$ {R}^{2} $ 和$ {q}^{2} $ 值远远低于最优模型的原始$ {R}^{2} $ 和$ {q}^{2} $ 值。因此,可以判定本章所建立的最优QSAR模型并非偶然所得并展现了良好的稳健性。表4为模型4中各参数的统计学检验数据,可以看出所有自变量参数的t值绝对值都大于检验标准临界值2.037,显著性检验结果均小于0.05且VIF检验值均大于1并远小于5,表明模型中各自变量具有显著的统计学意义且各自变量之间无多重共线性的可能。
最优QSAR模型中各量子化学参数具有明显的化学意义,其中,f(-)为福井函数的一个指标,其代表了化学反应过程中的亲电攻击点位的选择[20],表明了最高占据轨道的电子贡献能力,其值越大,表示该区域易失去电子而化学反应。模型中所包含的f(-)n代表了在亲电反应中有机污染物分子内各原子电子变化的最小值,它与TN%呈反比例关系。相反,f(+)代表了化学反应过程中的亲核攻击点位的选择,其可表明最低未占据轨道的接受电子能力[21],其值越小,说明该区域越易得到电子而发生化学反应,其作用机理与f(-)相反,从而导致f(+)与TN%呈正比例关系。Egap值是QSAR研究中常用到的量化参数[16,21],Egap越大表明分子的最高占据轨道能越小(越不易失电子)或最低未占据轨道能越大(越不易得电子),因此其值越大,表明有机污染物分子惰性越大且越不易与氧化剂发生反应[22]。偶极矩μ表示分子的极性性质,其值越大,表明分子极性越大,其与TN%呈正比例关系。综述所述,含氮有机污染物在SCWO中去除率受以上4个量子化学参数的影响。
-
本研究采用欧氏距离(Euclid Distance)来定义最优QSAR模型的应用域(APD)。欧氏距离通常为在m维空间空两个点之间的真实距离。应用在QSAR定义域方向则是测试集中数据样本与其最近的训练集样本之间的距离应小于APD。APD的计算公式如(2)[23—24]:
< d >的计算步骤如下:
(1) 计算出训练集数据样本中任意两个数据之间的欧氏距离的平均值;
(2) 统计训练集中小于(1)中平均值的欧几里得距离的数值,并组成新的数据集;
(3) (2)中新的数据集的平均值即为< d >,σ为新数据集中的标准偏差值,公式如(3):
其中,n为新数据集中样本个数,
$ {x}_{i} $ 为新数据集样本的值,$ \stackrel{~}{x} $ 为新数据集样本的平均值,Z一般取0.5。表5为模型4的应用域欧氏距离及测试集化合物距离与其最近的训练集化合物间的距离。如表5所示,本研究所建立的最优QSAR模型的APD距离为11.244。而测试集中9个有机污染物分子距离与其最近的训练集之间的距离都远远小于11.244,说明利用此QSAR模型对测试集化合物的预测值是可靠的。而对于其他新的化合物,当且仅当其与训练集中最近的化合物之间的欧几里得距离小于11.244时,方可采用此模型进行预测。
-
(1)本研究采用多元线性回归法针对含氮有机污染物分子在SCWO中TN去除率构建了其与有机污染物分子量子化学参数之间的构效关系。其最优QSAR模型为TN%=84.20−374.34f(-)n−99.74Egap+1.80μ+180.498f(+)n,模型的拟合度(
$ {R}^{2} $ )、内部验证($ {q}^{2} $ )和外部验证($ {Q}_{test}^{2} $ )结果分别为0.758、0.658和0.581,表明模型具有良好的预测能力。其他统计学验证结果表明模型具备较好的统计学意义和稳定性。(2)最优模型参数分析显示f(-)n、Egap、μ和f(+)是决定含氮有机污染物分子在SCWO中TN去除的关键因素,四个量子化学参数代表了有机污染物分子降解氧化位点的选择和分子电子的转移。通过使用该最优模型,可为含氮有机污染物在SCWO中TN去除规律研究提供初始评估和理论指导。
含氮有机污染物在超临界水氧化(SCWO)中去除的QSAR模型
Quantitative-structure-activity-relationship (QSAR) models for the removal of nitrogenous organic contaminants in supercritical water oxidation (SCWO)
-
摘要: 超临界水氧化(SCWO)作为一项高效的去除水体中有机污染物的技术已得到了广泛的应用。为了更好地理解含氮有机物污染物在SCWO中总氮(TN)去除的规律,本研究以定量构效关系(QSAR)模型为方法,构建了41种含氮有机污染物在SCWO中TN%与有机污染物分子量子化学参数之间的QSAR模型。其最优QSAR模型结果为TN%=84.20-374.34f(-)n−99.74Egap+1.80μ+180.498f(+)n,模型的验证结果表明,所建的最优QSAR模型对于预测TN%展现了良好的稳定性和预测能力。模型中量子化学参数分析结果表明,亲核及亲电攻击福井指数、偶极矩以及分子的前线轨道能量差是影响含氮有机污染物分子在SCWO中TN去除的内在量子因素,该模型的建立可为含氮有机污染物在SCWO中TN的去除效果和规律研究提供初始评估。Abstract: Supercritical water oxidation (SCWO) is an effective method for wastewater treatment. To gain a better understanding of the total nitrogen removal rate (TN%) for nitrogenous organic contaminants in SCWO, a Quantitative-Structure-Activity-Relationship (QSAR) model was applied to establish the relationship between TN% and quantum chemical descriptors of 41 nitrogenous organics. The optimal QSAR model was TN%=84.20-374.34f(-)n−99.74Egap+1.80μ+180.498f(+)n. Validation tests demonstrated that the developed QSAR model was stable and had good predictive ability. Based on the quantum chemical descriptors in the model, nucleophilic and electrophilic attacks, dipole moment and the difference between lowest unoccupied and highest occupied molecular orbital energies are the intrinsic factors influencing the TN removal of nitrogenous organics during SCWO process. The proposed QSAR model provides a preliminary evaluation for studying the TN removal rate and rules of nitrogenous organic contaminants in SCWO process.
-
Key words:
- SCWO /
- Nitrogenous organic contaminants /
- TN removal rate /
- QSAR /
- Quantum chemical descriptor
-

-
表 1 41种有机污染物的总氮去除率与19种量子化学参数数值表(*为测试集化合物)
Table 1. The specific values for TN removal rate and 19 quantum chemical parameters of 41 nitrogenous contaminants (* stands for the compounds in the test set)
有机污染物
Organic
pollutantsTN% μ
(Debye)$ q({H)}^{+} $
(e)$ q({CH)}_{x}^{+} $
(e)$ q({CH)}_{n}^{+} $
(e)$ q({{C}^{-})}_{x} $
(e)$ q({{C}^{-})}_{n} $
(e)$ q\left(N\right) $
(e)$ {E}_{LUMO} $
(a.u.)$ {E}_{HOMO} $
(a.u.)$ {E}_{gap} $
(a.u.)$ {E}_{B3LYP} $
(a.u.)$ {BO}_{x} $ $ {BO}_{n} $ $ f({+)}_{x} $
(e)$ f({+)}_{n} $
(e)$ f({-)}_{x} $
(e)$ f({-)}_{n} $
(e)$ f({0)}_{x} $
(e)$ f({0)}_{n} $
(e)Aniline 85.106 7.133 0.329 0.199 0.194 0.181 −0.256 −0.792 0.002 −0.198 0.200 −289.380 1.845 1.268 0.124 0.047 0.127 0.017 0.107 0.093 3,4-DCA 64.135 4.561 0.429 0.22 0.205 0.199 −0.273 −0.771 −0.03 −0.222 0.192 −1206.92 1.515 1.142 0.123 0.034 0.092 0.038 0.101 0.049 N,N-DMBA 67.873 0.651 0.215 0.203 0.161 −0.029 −0.348 −0.499 −0.008 −0.218 0.21 −407.109 1.397 0.973 0.082 −0.013 0.016 −0.035 0.042 −0.034 SN 74.721 5.455 0.385 0.237 0.199 0.21 −0.32 −0.758 −0.027 −0.231 0.204 −889.704 1.963 0.927 0.091 0.028 0.123 0.025 0.096 0.026 SA 64.706 5.839 0.491 0.229 0.198 0.209 −0.337 −0.701 −0.037 −0.239 0.202 −907.498 1.441 1.268 0.089 0.05 0.091 0.047 0.081 0.06 4-DMABA 82.811 6.43 0.226 0.221 0.091 0.219 −0.349 −0.668 −0.044 −0.213 0.169 −482.59 1.667 1.037 0.151 0.017 0.099 0.007 0.094 0.021 RB 88.627 9.211 0.391 0.279 0.188 0.454 −0.569 −0.674 −0.011 −0.225 0.214 −1879.86 1.53 0.962 0.063 −0.005 0.038 −0.007 0.062 −0.006 CV 85.115 13.449 0.469 0.284 0.179 0.259 −0.592 −0.631 −0.005 −0.243 0.238 −1589.34 1.503 1.103 0.053 0.002 0.05 0.007 0.051 0.007 BG 66.102 11.221 0.511 0.29 0.191 0.267 −0.586 0.499 −0.005 −0.195 0.19 −1847.93 1.491 0.846 0.104 −0.103 0.058 −0.004 0.066 0.001 3,5-DA 69.129 3.759 0.512 0.28 0.271 0.808 −0.189 0.541 −0.147 −0.309 0.162 −911.357 1.639 0.792 0.131 0.012 0.141 0.047 0.12 0.019 2-NT 70.109 4.262 0.239 0.239 0.2 0.059 −0.587 0.519 −0.089 −0.276 0.187 −481.209 1.604 0.925 0.175 0.015 0.116 0.013 0.143 0.023 4-NP 75.539 5.302 0.462 0.241 0.21 0.369 −0.279 0.551 −0.091 −0.262 0.171 −509.761 1.836 1.296 0.116 0.045 0.087 0.007 0.096 0.066 3-NBA* 70.012 3.674 0.492 0.276 0.262 0.814 −0.157 0.518 −0.128 −0.32 0.192 −930.041 1.911 0.911 0.122 0.018 0.23 0.011 0.139 0.011 3,5-DNBA 62.859 5.729 0.479 0.244 0.201 0.377 −0.291 0.522 −0.048 −0.261 0.213 −509.869 1.438 1.213 0.083 0.046 0.125 0.031 0.089 0.037 EBBR 92.593 8.489 0.204 0.248 0.176 0.453 −0.151 −0.551 −0.039 −0.176 0.137 −1807.77 1.732 1.187 0.046 0.001 0.039 −0.019 0.046 0.007 OG 82.653 3.935 0.461 0.257 0.183 0.348 −0.263 −0.279 −0.07 −0.196 0.126 −2381.37 1.514 1.198 0.063 0.01 0.047 0.005 0.055 0.006 ACBK 90.164 9.628 0.427 0.246 0.206 0.411 −0.303 −0.116 −0.095 −0.212 0.117 −3299.97 1.95 0.881 0.111 −0.002 0.063 0 0.072 0.003 SR23 92.391 5.829 0.269 0.235 0.2 0.332 −0.218 −0.127 −0.099 −0.207 0.108 −1142.39 1.822 0.939 0.144 −0.008 0.075 −0.01 0.069 −0.002 1-MLD 75.694 5.137 0.199 0.221 0.175 0.216 −0.239 −0.388 0.022 −0.227 0.249 −262.231 1.667 0.964 0.166 0.035 0.199 0.009 0.155 0.04 1,10-Phen 81.746 3.196 0.211 0.211 0.179 0.188 −0.245 −0.643 −0.063 −0.241 0.178 −569.74 1.703 0.097 0.084 0.019 0.049 −0.025 0.068 0.033 Cyanuric acid 54.746 6.89 0.489 0 0 0.451 0.311 0.481 −0.019 −0.295 0.276 −511.756 1.513 0.888 0.159 0.036 0.261 0.041 0.169 0.037 Isatin 63.587 4.622 0.409 0.219 0.206 0.217 −0.254 −0.659 −0.105 −0.249 0.144 −513.2 1.342 0.878 0.119 0.026 0.076 0.027 0.096 0.025 Quinoline 92.391 2.029 0.218 0.213 0.151 0.18 −0.247 −0.457 −0.063 −0.242 0.179 −411.928 1.576 0.985 0.106 0.019 0.11 0.016 0.096 0.018 Pyridine 75.758 3.19 0.302 0.222 0.179 0.059 −0.559 −0.449 −0.031 −0.259 0.228 −251.864 1.444 0.986 0.161 0.057 0.131 0.053 0.138 0.06 Diazine 81.746 5.25 0.292 0.224 0.189 0.03 −0.199 −0.231 −0.059 −0.239 0.18 −277.352 1.493 0.984 0.165 0.056 0.181 0.061 0.173 0.058 EBT 54.746 13.641 0.489 0.277 0.199 0.361 −0.629 −0.319 −0.025 −0.217 0.192 −2109.67 1.785 0.898 0.079 0.005 0.09 0.012 0.084 0.004 Azure B* 63.587 8.382 0.322 0.228 0.131 0.262 −0.293 −0.678 −0.01 −0.286 0.276 −1505.09 1.559 1.312 0.046 0.015 0.032 −0.007 0.038 0.015 2,4-DNPH 70.115 8.983 0.339 0.258 0.199 0.569 −0.601 −0.681 −0.137 −0.255 0.118 −2000.15 1.531 0.886 0.053 −0.003 0.08 0.005 0.076 0.001 AYGG* 66.102 7.867 0.408 0.269 0.239 0.217 −0.439 −0.659 −0.103 −0.283 0.18 −749.251 1.46 1.35 0.139 0.019 0.107 0.023 0.109 0.027 Melamine * 62.634 2.861 0.271 0.241 0.198 0.206 −0.266 −0.883 −0.086 −0.231 0.145 −502.426 1.678 1.201 0.079 0.021 0.121 −0.009 0.077 0.039 2-NA 71.795 4.008 0.469 0.189 −0.251 0.245 0.215 −0.889 −0.092 −0.232 0.14 −488.532 1.671 0.916 0.188 0.021 0.171 −0.012 0.123 0.033 3-NA 64.341 7.093 0.476 0.251 0.211 0.226 −0.207 −0.909 −0.122 −0.141 0.019 −489.267 1.386 1.109 1.664 0.019 1.664 0.028 1.664 0.034 5-Cl-2-MA* 83.333 3.011 0.254 0.229 0.202 −0.007 −0.549 −0.848 −0.024 −0.245 0.221 −825.951 1.379 1.003 0.117 0.026 0.116 0.019 0.085 0.024 NB* 78.298 5.142 0.341 0.241 0.206 0.057 −0.288 0.016 −0.138 −0.273 0.135 −440.784 1.601 1.319 0.091 0.036 0.058 −0.021 0.079 0.015 2,6-DNT* 87.64 2.53 0.241 0.242 0.214 0.088 −0.607 −0.016 −0.107 −0.289 0.182 −679.907 1.613 0.905 0.121 0.011 0.104 0.022 0.111 0.021 2-NP 75.188 5.259 0.485 0.26 0.214 0.814 −0.191 0.357 −0.105 −0.298 0.193 −625.493 1.929 0.909 0.184 0.012 0.221 −0.013 0.14 0.019 Arsenzao 1* 65 5.729 0.417 0.258 0.213 0.442 −0.249 −0.192 −0.118 −0.236 0.12 −4592.27 1.959 0.733 0.063 0.001 0.157 −0.002 0.11 0.002 Indole* 93.324 2.199 0.401 0.229 0.189 0.159 −0.29 −0.541 −0.019 −0.211 0.192 −359.058 1.601 0.971 0.119 0.024 0.118 0.042 0.117 0.026 MO 79.734 0.494 0.488 0.39 0.385 0.667 0.667 −0.779 0.02 −0.234 0.254 −439.558 1.394 0.956 0.13 0.01 0.12 0.009 0.121 0.027 AO74 84.984 13.83 0.492 0.239 0.172 0.481 −0.301 −0.773 −0.014 −0.27 0.266 −1612.54 1.684 1.037 0.041 0.013 0.044 −0.011 0.047 0.009 4-NA 85.799 7.848 0.471 0.243 0.203 0.757 −0.225 −0.289 −0.12 −0.226 0.106 −1199.97 1.642 0.778 0.143 −0.006 0.233 −0.015 0.135 −0.003 表 2 量子化学结构参数
Table 2. Instruction of Quantum-Chemical Descriptors
缩写
Abbreviations含义
Meaning单位
Unitμ 有机污染物分子在真空状态下的偶极距 Debye $ q({H)}^{+} $ 有机污染物分子氢原子局部最大电荷 e $ q({CH)}_{x/n}^{+} $ 有机污染物分子与碳原子相连的氢原子的局部最大/最小电荷 e $ q({{C}^{-})}_{x/n} $ 有机污染物分子优化后碳原子的局部最大电荷 e $ {E}_{B3LYP} $ 有机污染物分子在B3LYP方法下的分子能量 a.u. $ {E}_{LUMO} $ 前线轨道理论中电子最低未占据轨道能量 a.u. $ {E}_{HOMO} $ 前线轨道理论中电子最高占据轨道能量 a.u. $ {E}_{gap} $ 最低未占据轨道能量与最高占据轨道能量差 a.u. $ f({+)}_{x/n} $ 有机污染物分子亲核攻击指数最大/小值 e $ f({-)}_{x/n} $ 有机污染物分子亲电攻击指数最大/小值 e $ f({0)}_{x/n} $ 有机污染物分子优化后亲自由基攻击指数最大/小值 e $ {BO}_{x/n} $ 有机污染物分子键级的最大/小值 表 3 41种含氮有机污染物TN%QSAR模型一览表
Table 3. Regression Models for Calculating TN% 41 Nitrogenous Organic Compounds
No. Model $ {R}^{2} $ SD $ {q}^{2} $ F sig. RMSE $ {Q}_{ext}^{2} $ 1 TN%=79.226−259.854 f(-)n 0.372 8.654 0.247 15.843 0.000 73.737 0.326 2 TN%=93.470−240.967f(-)n−77.632Egap 0.510 7.655 0.407 15.074 0.000 57.151 0.270 3 TN%=88.514−224.469f(-)n−81.264Egap+0.891μ 0.595 6.954 0.472 13.693 0.000 48.934 0.443 4 TN%=87.261−317.511f(-)n−92.963Egap+1.223μ+151.457f(+)n 0.701 5.940 0.575 15.819 0.000 37.443 0.519 表 4 模型4的统计学检验
Table 4. Statistical Checking Values for Model 4
Variables Regression coefficients t Sig. VIF Constant 84.200 ± 4.212 19.241 0.000 f(-)n −374.340 ± 54.660 −6.302 0.000 1.358 Egap −99.740 ± 20.307 −4.654 0.000 1.050 Model 4 μ 1.794 ± 0.380 4.315 0.000 1.150 f(+)n 180.498 ± 48.016 3.372 0.002 1.521 Criterion |t|>2.037 <0.05 1~5 表 5 模型4的APD及测试集中化合物到其临近训练集的距离
Table 5. The APD and distances of test compounds to neighbors in the training set for model 4
测试集化合物
Test compounds欧式距离
Euclid DistanceThe euclid distance of model 4 11.244 5-Cl-2-MA 7.181 NB 10.925 2,6-DNT 3.174 3-NBA 0.818 Arsenazo I 2.277 Indole 5.805 Cyanuric acid 5.458 Azure B 2.530 AYGG 5.121 -
[1] 董建威. 高浓度含氮废水处理技术研究 [J]. 广东化工, 2014, 41(15): 166-167. doi: 10.3969/j.issn.1007-1865.2014.15.085 DONG J W. Research rrogress in the treatment of high ammonia content wastewater [J]. Guangdong Chemical Industry, 2014, 41(15): 166-167(in Chinese). doi: 10.3969/j.issn.1007-1865.2014.15.085
[2] 李兰娟, 刘福强, 凌盼盼, 等. 含氮废水处理技术和工艺研究进展 [J]. 安全与环境学报, 2009, 9(5): 46-51. doi: 10.3969/j.issn.1009-6094.2009.05.011 LI L J, LIU F Q, LING P P, et al. Review on the technology of nitrogen containing wastewater treatment [J]. Journal of Safety and Environment, 2009, 9(5): 46-51(in Chinese). doi: 10.3969/j.issn.1009-6094.2009.05.011
[3] 徐向阳, 郑昱. 序批式气提生物反应器(SABR)处理氯苯胺类有机废水好氧污泥颗粒化研究 [J]. 环境科学学报, 2005, 11: 20-28. XU X Y, ZHENG Y. Granulation of aerobic sludges in sequencing airlift bioreactor (SABR) for the treatment of chloroanilines-containing organic wastewater [J]. Journal of Environmental Science, 2005, 11: 20-28(in Chinese).
[4] LUO Z, LI X, ZHAI J. Kinetic investigations of quinoline oxidation by ferrate (VI) [J]. Environmental Technology, 2016, 37(10): 1249-1256. doi: 10.1080/09593330.2015.1111424 [5] JIANG L, HU X, YIN D, et al. Occurrence, distribution and seasonal variation of antibiotics in the Huangpu River, Shanghai, China [J]. Chemosphere, 2011, 82(6): 822-828. doi: 10.1016/j.chemosphere.2010.11.028 [6] AKPAN U G, HAMEED B H. Parameters affecting the photocatalytic degradation of dyes using TiO2-based photocatalysts: a review [J]. Journal of Hazardous Materials, 2009, 170: 520-529. doi: 10.1016/j.jhazmat.2009.05.039 [7] MURALI V, ONG S A, HO L N, et al. Evaluation of integrated anaerobic-aerobic biofilm reactor for degradation of azo dye methyl orange [J]. Bioresource Technology, 2013, 143: 104-111. doi: 10.1016/j.biortech.2013.05.122 [8] SHIN Y H, SHIN N C, VERIANSYAH B, et al. Supercritical water oxidation of wastewater from acrylonitrile manufacturing plant [J]. Journal of Hazardous Materials, 2009, 163(2-3): 1142-1147. doi: 10.1016/j.jhazmat.2008.07.069 [9] LIU S, JIN H, WEI W, et al. Gasification of indole in supercritical water: Nitrogen transformation mechanisms and kinetics [J]. International Journal of Hydrogen Energy, 2016, 41(36): 15985-15997. doi: 10.1016/j.ijhydene.2016.04.219 [10] QU R, LIU H, FENG M, et al. Investigation on intramolecular hydrogen bond and some thermodynamic properties of polyhydroxylated anthraquinones [J]. Journal of Chemical and Engineering Data, 2012, 57: 2442-2455. doi: 10.1021/je300407g [11] MAHMOUD W M, TOOLARAM A P, MENZ J, et al. Identification of phototransformation products of thalidomide and mixture toxicity assessment: an experimental and quantitative structural activity relationships (QSAR) approach [J]. Water Research, 2014, 49: 11-22. doi: 10.1016/j.watres.2013.11.014 [12] JIA L, SHEN Z, GUO W, et al. QSAR models for oxidative degradation of organic pollutants in the Fenton process [J]. Journal of the Taiwan Institute of Chemical Engineers, 2015, 46: 140-147. doi: 10.1016/j.jtice.2014.09.014 [13] ZHU H, GUO W, SHEN Z, et al. QSAR models for degradation of organic pollutants in ozonation process under acidic condition [J]. Chemosphere, 2015, 119: 65-71. doi: 10.1016/j.chemosphere.2014.05.068 [14] ZHU H, SHEN Z, TANG Q, et al. Degradation mechanism study of organic pollutants in ozonation process by QSAR analysis [J]. Chemical Engineering Journal, 2014, 255: 431-436. doi: 10.1016/j.cej.2014.05.073 [15] LUO X, YANG X, QIAO X, et al. Development of a QSAR model for predicting aqueous reaction rate constants of organic chemicals with hydroxyl radicals [J]. Environmental Science:Processes & Impacts, 2017, 19(3): 350-356. [16] XIAO R, YE T, WEI Z, et al. Quantitative structure-activity relationship (QSAR) for the oxidation of trace organic contaminants by sulfate radical [J]. Environmental Science & Technology, 2015, 49(22): 13394-13402. [17] FRISCH M J, TRUCKS G W, SCHLEGEL H B, et al. Gaussian[CP]. Inc Wallingford, CT, 2009. [18] MODULE C C. Material Studio, version 7.0[CP]. Accelrys Inc. , San Diego, CA. , 2013. [19] PINTO L D S, DOS SANTOS L M F, AL-DURI B, et al. Supercritical water oxidation of quinoline in a continuous plug flow reactor—part 1: effect of key operating parameters [J]. Journal of Chemical Technology & Biotechnology, 2006, 81(6): 912-918. [20] FUENTEALBA P, FLOREZ E, TIZNADO W. Topological analysis of the Fukui function [J]. Journal of Chemical Theory and Computation, 2010, 6: 1470-1478. doi: 10.1021/ct100022w [21] HU J Y, MORITA T, MAGARA Y, et al. Evaluation of reactivity of pesticides with ozone in water using the energies of frontier molecular orbitals [J]. Water Research, 2000, 34(8): 2215-2222. doi: 10.1016/S0043-1354(99)00385-1 [22] SAJAN D, JOSEPH L, VIJAYAN N, et al. Natural bond orbital analysis, electronic structure, non-linear properties and vibrational spectral analysis of L-histidinium bromide monohydrate: a density functional theory [J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2011, 81(1): 85-98. doi: 10.1016/j.saa.2011.05.052 [23] MELAGRAKI G, AFANTITIS A. Enalos KNIME nodes: Exploring corrosion inhibition of steel in acidic medium [J]. Chemometrics and Intelligent Laboratory Systems, 2013, 123: 9-14. doi: 10.1016/j.chemolab.2013.02.003 [24] ZHANG S, GOLBRAIKH A, OLOFF S, et al. A novel automated lazy learning QSAR (ALL-QSAR) approach: method development, applications, and virtual screening of chemical databases using validated ALL-QSAR models [J]. Journal of Chemical Information and Modeling, 2006, 46: 1984-1995. doi: 10.1021/ci060132x -



 下载:
下载: