-
汞(Hg)是一种生物非必需的重金属元素,由于其具有长距离迁移性、生物累积性、高毒性等特点,已经被联合国环境规划署(UNEP)列为全球性的污染物[1]。在自然环境中,汞元素以无机汞(如零价汞单质、二价汞盐等)和有机汞(如甲基汞、乙基汞等)形态存在,并且在环境条件下会发生相互转化。不同化学形态汞的物理化学性质和生物毒性差异显著,甲基汞(MeHg)是对人类和环境健康威胁最大的含汞化合物。甲基汞具有极强的神经毒性,在食物链和生物圈中易发生富集、放大效应[2-3],海产品和农作物中的甲基汞严重威胁食品安全和人类健康[4-5]。正确认识自然环境中甲基汞的生成和累积过程是汞污染研究领域的热点和难点,同时也是有效防治全球性汞污染的核心环节之一。
食物链中累积的甲基汞主要来自于环境中广泛分布的厌氧菌所驱动的无机汞甲基化过程[6]。生物膜(biofilm)是水生食物链的重要基础,代表着细菌和古菌在自然环境中的主要生存方式[7],驱动了众多元素的生物地球化学循环过程[8]。在环境汞循环过程中,生物膜参与汞的还原、氧化、甲基化和去甲基化等形态转化过程[9-10]。进入到生物膜中的二价汞可经由微生物的还原作用形成零价汞后扩散、挥发进入周围环境;在Desulfovibrio desulfuricans ND132等微生物的作用下,零价汞也会被氧化为二价汞[11];同时,生物膜是驱动零价、二价无机汞与甲基汞之间相互转化的重要环境介质。由于生物膜是食物链中甲基汞的重要来源,Branfireun等将生物膜添加到了最新总结的淡水生态系统汞的循环模型中[9],但是目前关于生物膜在汞的环境转化过程,特别是甲基化过程中的作用仍缺乏系统认识。
在甲基汞微生物合成机制的前期研究中,大多采用了浮游态细菌培养物进行实验,然而有研究表明,生物膜内部微生物细胞的生活习性、生理代谢途径等明显不同于浮游态细胞,生物膜中发生的生化过程无法通过研究浮游态细胞进行准确预测[12]。此外,研究发现,沉积物、周丛生物、水生植物等附着的生物膜中甲基汞占总汞的比例约为10%,明显高于外部水环境[10]。与厌氧区域的沉积物和含水层相比,生物膜中汞甲基化速率往往高出1—2个数量级[13-14]。这些表明生物膜中可能存在不同于浮游态微生物的汞甲基化作用机制。生物膜是甲基汞生成的重要环境介质和甲基汞在食物链中富集的起点,明确生物膜中汞甲基化过程的作用机制是有效保障食品安全,控制汞污染健康风险的重要前提,甲基汞在生物膜中的生成机制亟待进一步系统研究。
-
国际纯粹与应用化学联合会(IUPAC)对生物膜的定义:生物膜是粘附于固体表面并被微生物细胞自身产生的胞外多聚物(extracellular polymeric substances, EPS)所包裹的微生物细胞的聚集体[15]。自然环境中绝大多数微生物,特别是活性最强的群体,多以生物膜的形式存在[16],生物膜的平均细胞密度大约为1014 cells·dm−3 [17]。作为汞甲基化最为活跃的环境介质之一,海洋表层沉积物中以生物膜形式附着生长的细胞丰度高出浮游态2—3个数量级[7]。Cleckner等报道了周丛生物附着生物膜是沼泽湿地地区中甲基汞生成的新位点 [18]。研究发现,当硫酸盐还原菌(Desulfovibrio desulfuricans)形成生物膜后,其将无机汞转化为甲基汞的速率比浮游态细菌高出近10倍[14]。这些研究表明生物膜是自然环境中一个潜在的汞甲基化热点区域。
后续调查研究发现,不同生物膜中甲基汞的累积程度差异较大,例如美国地质调查局(USGS)和美国环境保护局(USEPA)曾联合开展一项关于河流中汞循环的调查研究发现,表层沉积物附着生物膜中甲基汞的平均浓度大约是卵石和沉木附着生物膜的20倍[19]。不同生物膜中计算得到的汞甲基化速率常数(km)和甲基化效率(MeHg/THg)也存在较大差异,如表1所示。造成这种差异的一个可能原因是不同生物膜中主导甲基汞产生的微生物种类不同,例如湖滨岩石带生物膜中硫酸盐还原菌对甲基汞产量贡献了约60% [20],而湖泊水生植物附着的生物膜中产甲烷菌在甲基汞生成过程中起关键作用[21],此外附着生物膜中的藻类微生物被发现具有增强厌氧菌汞甲基化的能力[22]。
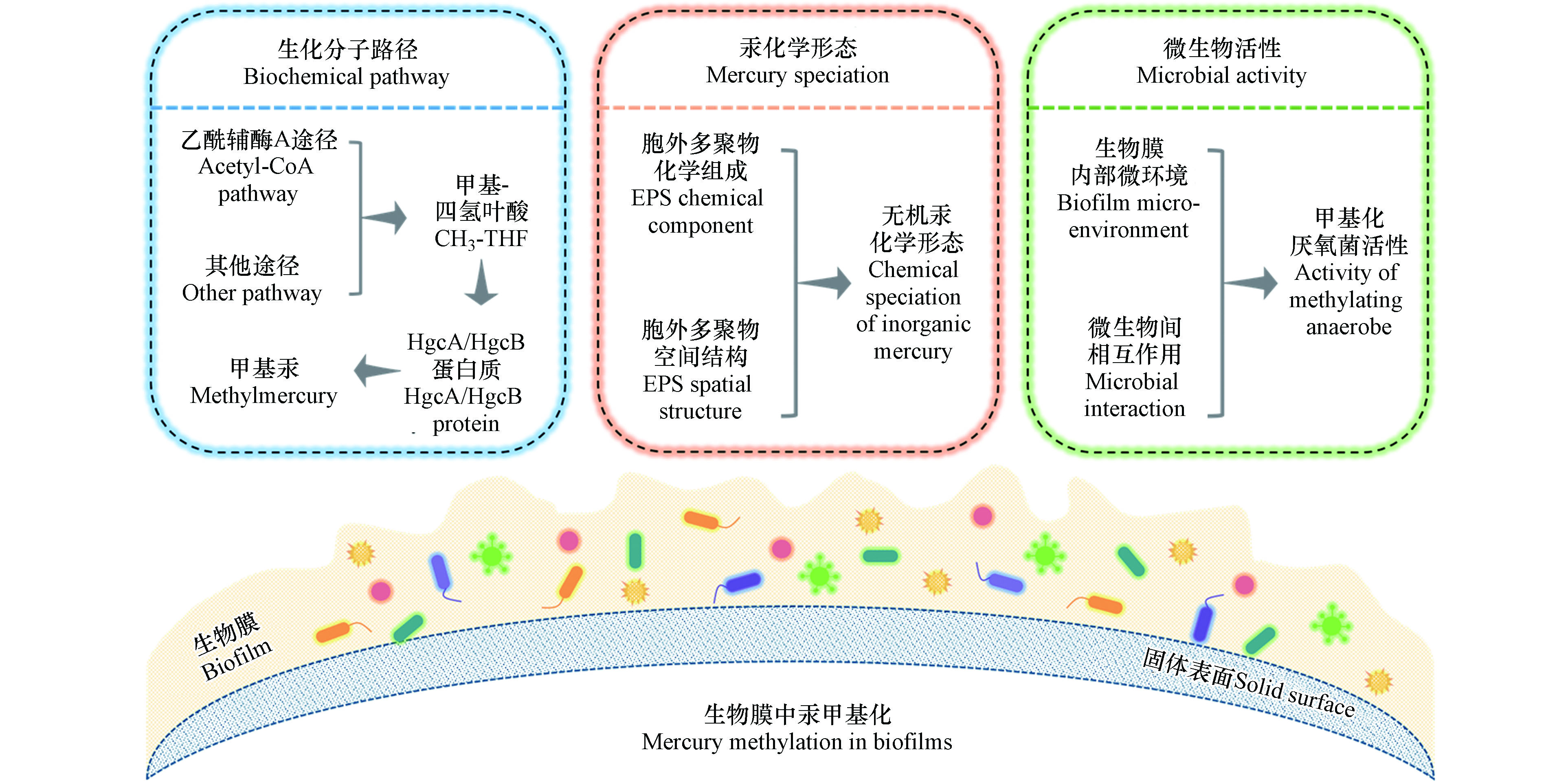
上述调查和研究结果表明,生物膜中微生物汞甲基化反应机制可能不同于浮游态微生物。进一步的研究发现,季节、温度、光照、溶解氧、有机质、营养盐等外部环境条件均会影响生物膜中汞甲基化过程[30-33, 36]。除了外部环境条件外,微生物汞甲基化的生化分子路径、无机汞的化学形态和甲基化微生物的活性是决定微生物汞甲基化过程的三个关键内部因素[35, 37]。因此,系统地阐明这三个关键因素如何影响汞甲基化过程,是生物膜中微生物汞甲基化研究的重点(图1)。
-
Jensen和Jernelöv首次通过实验验证了沉积物中汞甲基化作用主要是由微生物导致的[38]。随后陆续发现了产甲烷菌、硫酸盐还原菌、铁还原菌等具有汞甲基化能力的微生物[39-41]。早期研究发现,完全氧化型硫酸盐还原菌利用乙酰辅酶A途径将甲基-四氢叶酸(CH3-THF)的甲基转移到一种类咕啉蛋白质上,然后通过一氧化碳脱氢酶(CODH)催化进行汞甲基化反应[42-43]。然而,非完全氧化型硫酸盐还原菌不利用乙酰辅酶A途径也可以进行汞甲基化反应,这表明硫酸盐还原菌可能存在多种汞甲基化生化路径[44-45]。起初,不同种群的微生物的汞甲基化分子机制并不清楚,直到Parks等首次报道了微生物汞甲基化基因hgcA和hgcB才揭示了汞的微生物甲基化遗传机制[46]。hgcA基因编码一种类咕啉蛋白(HgcA),hgcB基因则编码一种铁氧化还原蛋白(HgcB)共同参与汞甲基化反应。推测的甲基化过程:CH3-THF将甲基转移给Co(Ⅰ)-HgcA形成CH3-Co(Ⅲ)-HgcA,随后CH3-Co(Ⅲ)-HgcA将甲基提供给Hg(Ⅱ)完成汞甲基化,自身则接收HgcB蛋白提供的电子还原为Co(Ⅰ)-HgcA,重新参与到反应中。hgcA和hgcB基因在目前已知的的汞甲基化微生物中均有发现,这也让其成为微生物汞甲基化的必要条件[47]。
在相同的环境条件下,与浮游态微生物相比,生物膜中与细胞生理功能和代谢途径相关的基因可能会显著上调,从而导致生物膜与浮游态微生物具有明显的表型差异[48-49]。尽管目前浮游态微生物汞甲基化的分子机制已经相对清晰,但是作为环境中微生物普遍生存方式的生物膜中汞甲基化的生化路径却少有研究。目前,仅有Lin等利用非完全氧化型硫酸盐还原菌Desulfovibrio desulfuricans M8和ND132形成生物膜,对比考察了生物膜形态和浮游形态的汞甲基化生化路径的差异[35]。在抑制了乙酰辅酶A途径后,浮游态微生物汞甲基化速率未受显著影响,而生物膜中的汞甲基化速率降低了50%。而且,生物膜中编码CODH酶的cooS基因表达量是浮游态细菌的4倍,进一步证明了乙酰辅酶A途径是生物膜中汞甲基化的重要机制。乙酰辅酶A途径的存在可能是导致生物膜中汞甲基化速率比浮游态细菌更高的原因之一。
生物膜并不是单个微生物的简单堆砌聚集,生存在生物膜中的微生物具有明显的社会性,微生物细胞之间会进行复杂的通讯交流。群体感应、第二信使分子等信号调节系统会启动或抑制相关基因的表达,调控微生物多种重要生理功能,如调节中心代谢、影响生物膜形成、控制芽胞发育等以适应环境条件的改变,从而使微生物群体实现单一微生物无法完成的生理功能或行为[50-51]。但是,在生物膜的汞甲基化过程中,群体感应、第二信使分子等信号调节系统是否会起作用,是否会导致生物膜中厌氧菌采取不同于浮游态细胞的甲基化生化路径有待于进一步的研究。
-
生物膜细胞周围包裹着大量由细胞自身产生的生物大分子组成的聚合物,称为胞外多聚物(EPS)。胞外多聚物对于生物膜的存在至关重要,起到粘附支撑生物膜、保持水分、提供防护屏障、吸附有机和无机物质,维持营养等作用[52]。胞外多聚物主要含有蛋白质、多糖和胞外核酸等生物大分子,其组成和结构具有高度的异质性,受微生物种类、生长阶段、营养条件、温度、pH值等多种因素的影响[53]。胞外多聚物具有芳香结构及羧基、羟基、醛基、巯基等丰富的官能团,会通过化学络合、静电作用等方式从水中快速富集Hg(Ⅱ)、Pb(Ⅱ)、Cd(Ⅱ)、Zn(Ⅱ)、Cu(Ⅱ)等重金属元素[54-56],对重金属环境行为产生显著影响。
传统的预测甲基汞生物合成的量化模型中,只有溶解态、电中性、憎水性形态的含汞络合物(Hg(HS)20和HgS0(aq))可以作为甲基汞的前体物质进入微生物细胞内部参与甲基汞的生物合成[57-58]。然而最新研究发现,除了溶解态络合物,当无机汞以纳米颗粒物[59-60]和零价汞[11, 61]形态存在时,其也可以被厌氧菌利用产生甲基汞,甲基化效率低于溶解态络合物。无机汞的化学形态直接决定了汞的生物可利用性。
胞外多聚物如何影响无机汞的吸附、还原、沉淀等形态转化过程目前尚缺乏深入认识。研究发现,90%添加到培养物中的无机二价汞离子可以被细菌的胞外多聚物吸附,且胞外多聚物中的巯基基团是主要的作用基团[62-63]。Leclerc等发现,湖水附着生物膜中与汞具有强络合作用的小分子巯基化合物(如,巯基乙酸、半胱氨酸、谷胱甘肽等)的含量比周围水体高出3个数量级[64]。前期研究发现,小分子巯基化合物能显著增强部分汞甲基化细菌对无机汞的摄取和甲基化能力[65]。因此,生物膜中高含量的小分子巯基化合物可能影响生物膜中无机汞的甲基化过程。天然水环境形成的生物膜中已观测到了无机二价汞离子被还原的过程[66],此外有研究发现,胞外多聚物中的还原性组分(如,胞外多糖中的半缩醛结构)可以还原Ag(Ⅰ)和Au(Ⅲ)形成纳米尺寸的单质Ag(0)和Au(0)[67-68],但是胞外多聚物如何影响汞的氧化和还原过程目前有待研究。生物膜中富集的重金属元素多以纳米颗粒物,特别是硫化金属纳米颗粒物的形态存在,例如,天然水环境中形成的硫酸盐还原菌生物膜中检测到大量的ZnS纳米颗粒物[69]。Zhang等发现,厌氧菌胞外多聚物中的芳香结构基团通过与汞离子相互作用,能显著减缓HgS颗粒的沉淀团聚过程,使得HgS以结构更加无序的纳米颗粒物的形态存在,并且这种胞外多聚物包被的HgS纳米颗粒物可以被微生物利用产生甲基汞[70]。
除了具体组分、官能基团外,胞外多聚物的空间结构也可能在汞的形态转化和传质过程中起作用。Wang等发现,细菌胞外多聚物的空间结构为不完全伸展链组成的无规则卷曲结构,其空间结构受pH影响,在低pH值下倾向于形成更为致密的结构 [71-72]。重金属离子和纳米颗粒物在生物膜中的传质、扩散、迁移等过程受到静电作用、疏水作用、空间位阻等作用控制[16, 73],胞外多聚物空间结构的改变可能会影响溶解态络合物、纳米颗粒物等形态的无机汞在生物膜中的分布、迁移、转化及其甲基化过程。
-
环境中甲基汞的生成主要由厌氧微生物驱动,通过胞内酶促反应将无机汞转化为甲基汞,因此,厌氧微生物的活性是影响微生物汞甲基化过程的重要因素之一。生物膜作为微生物面对环境压力所采取的一种有利于生存的群居措施,其为微生物提供了一个相对稳定的环境以应对外界环境条件的波动[74]。生物膜中相对活性较高的厌氧菌的存在可能是生物膜中汞甲基化速率高于浮游态厌氧菌的原因之一[14]。
在胞外多聚物的包裹下,生物膜内部的环境条件与外部环境存在较大差异,例如,在外部好氧条件下,生物膜内部可形成一个有利于汞甲基化的厌氧微环境,这就为厌氧菌提供了一个额外的生态位[75]。Olsen等通过人为破坏生物膜的三维结构发现,生物膜内部厌氧微区遭到了破坏,汞甲基化微生物活性随之降低,进而导致汞甲基化速率降低[32]。生物膜中藻类等非甲基化微生物分泌的小分子巯基化合物,一方面可能会影响无机汞的化学形态,另一方面也能增强汞甲基化细菌的活性,促进细菌细胞吸收更多无机汞[25]。此外,胞外多聚物中丰富的多糖、蛋白质等有机物也可以被细菌利用进行呼吸代谢,从而增强其活性,促进汞的甲基化[76-77]。生物膜中汞甲基速率受到温度、光照、溶解氧、营养元素等环境因素的影响[10],当这些环境因素发生变化时,生物膜内部的微环境环境条件如何影响甲基化厌氧菌的活性有待于进一步的研究。
除了生物膜内部微环境会影响微生物活性外,生物膜中不同种群微生物之间的相互作用也会影响甲基化厌氧菌的活性。例如,硫酸盐还原菌和硫氧化菌往往共存于生物膜中,后者可以通过将硫氧化生成硫酸盐,为硫酸盐还原菌提供电子受体,从而促进硫酸盐还原菌的汞甲基化[27]。自然环境中汞甲基化过程往往由不同种类的微生物共同驱动,例如,Yu等研究发现,河流沉积物中主要的汞甲基化微生物为硫酸盐还原菌和铁还原菌[78]。Liu等发现,铁还原菌和产甲烷菌共同驱动了稻田土壤中甲基汞的生成过程[79]。通过不同浮游态细菌共培养实验发现,硫酸盐还原菌、产甲烷菌、互营杆菌等厌氧菌之间可以通过氢和乙酸转移方式建立互营共栖关系,进而增强其汞甲基化能力,汞甲基化速率相比单菌增加了2—9倍[80]。特别是在贫硫贫铁环境中,互营共栖的厌氧微生物可能是主要的甲基汞来源。当环境条件发生改变时,生物膜中不同菌种的响应不尽相同,其中甲基化细菌的活性可能也会随之改变,从而引起生物膜驱动的汞甲基化过程发生相应改变。
-
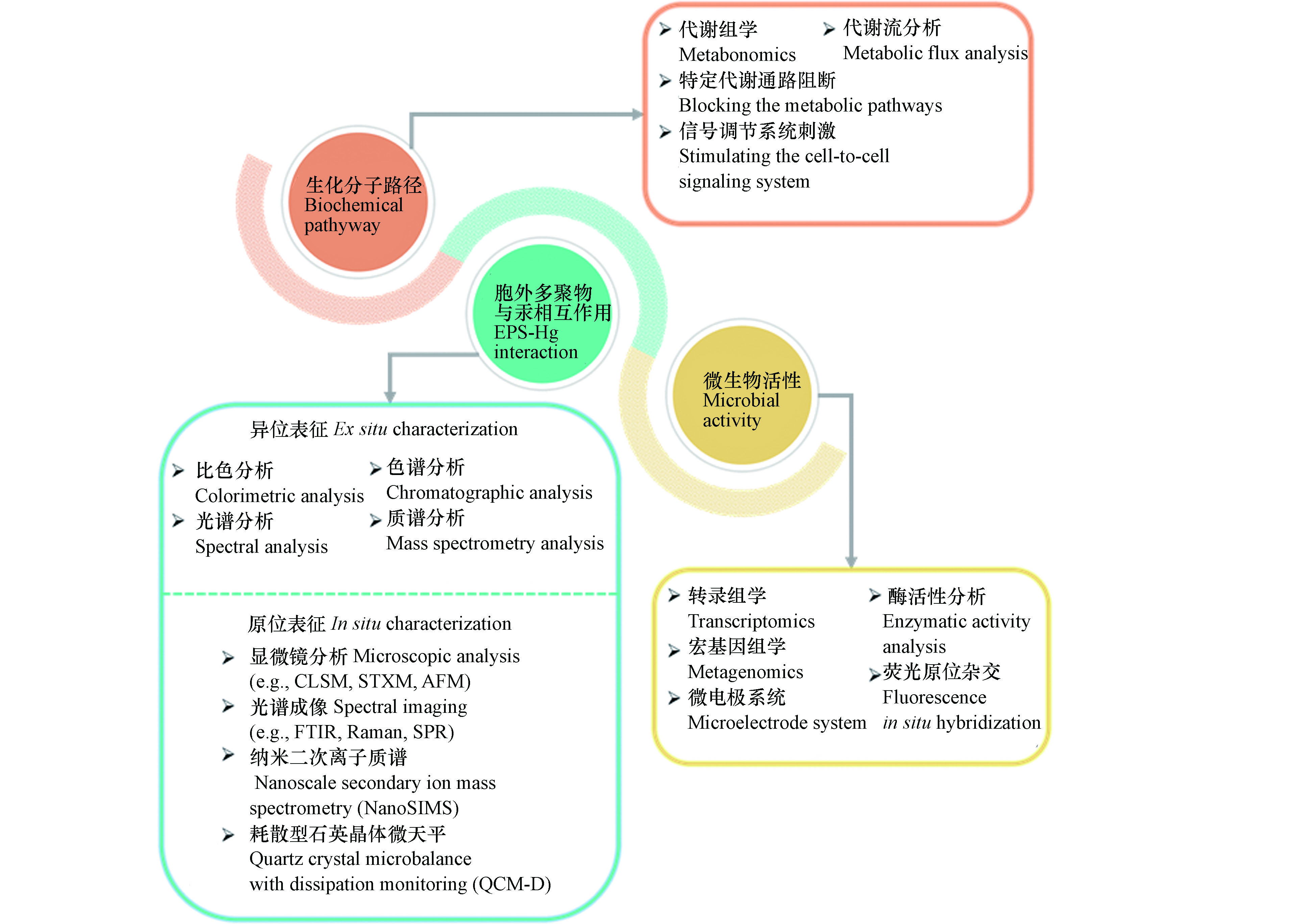
揭示生物膜中汞甲基化过程的作用机制依赖于准确、可靠、高效的仪器表征技术方法。针对生物膜微生物汞甲基化研究中存在的问题,可用于表征生化分子路径、胞外多聚物与汞相互作用、微生物活性的传统和新兴的技术方法,如图2中所示。
-
乙酰辅酶A途径(也称为Wood-Ljungdahl途径)是广泛存在于厌氧细菌和古菌中用于进行物质和能量代谢的通路之一[81]。前期利用硫酸盐还原菌进行的汞甲基化生化分子机制的研究中,普遍认为在合成乙酰辅酶A过程中产生的甲基-四氢叶酸是甲基的来源。在乙酰辅酶A途径中甲酸脱氢酶、乙酰辅酶A合成酶、一氧化碳脱氢酶等是该途径中的关键酶。通过加入相应的抑制剂,抑制关键酶活性,进而考察相应代谢途径是常用的研究方法之一。例如,Ekstrom等利用氯仿抑制一氧化碳脱氢酶,阻断乙酰辅酶A途径,发现完全氧化型硫酸盐还原菌的汞甲基化能力被抑制,而非完全氧化型硫酸盐还原菌影响较小,这表明非完全氧化型硫酸盐还原菌还可以利用其他代谢途径进行汞甲基化反应[44]。
抑制特定代谢通路的方法并不能准确描述具体的生化分子路径。随着代谢组学的发展,代谢流分析技术的出现为更加准确地研究具体代谢通路提供了可能[82]。代谢流分析是利用稳定同位素(如13C、15N等)标记特定化合物,结合核磁共振、色谱-质谱联用等方法,追踪分析下游代谢产物中稳定同位素组成,从而系统地定量特定代谢途径的走向、分布等情况[83]。不同种类汞甲基化微生物可利用的碳源不同,因此可以通过稳定同位素标记特定碳源(如乙酸、乳酸等),利用代谢流分析技术,揭示汞甲基化过程中甲基的来源,从而发现可能的新途径。
微生物在生长过程中会产生一种可以扩散的信号分子,在微生物群体密度较低时,信号分子保持在低浓度,而随着微生物生长,信号分子积累到临界浓度后会诱导目标基因的转录表达,进而表现出不同于浮游态微生物的生活状态和习性[84]。然而,目前关于微生物的群体感应等信号调节系统是否会影响微生物汞甲基化途径尚未见报道。N-酰基高丝氨酸内酯(AHL)是革兰氏阴性菌群体感应中广泛存在的一种通讯信号分子。AHL信号分子的产生依赖于LuxI类AHL合成酶,而其应答依赖于LuxR类受体蛋白[85]。因此,可以通过人为添加AHL信号分子,或者对AHL合成酶或受体蛋白进行抑制,刺激信号调节系统进而研究群体感应是否会影响微生物的汞甲基化。
-
胞外多聚物含有的蛋白质、多糖等生物大分子具有丰富的羧基、羟基、巯基等官能团,准确的表征胞外多聚物的分子组成和官能基团及其与无机汞的相互作用是研究生物膜中无机汞化学形态的基础。传统的方法是利用物理化学的手段将胞外多聚体从生物膜中分离,然后利用化学比色方法测定胞外多聚物的分子组成,例如,BCA法测定蛋白质含量,苯酚-硫酸法测定多糖含量,二苯胺法测定核酸含量等。胞外多聚物中的小分子化合物,可以在消解过后利用气相色谱、液相色谱-质谱联用等方法检测。官能团、芳香结构等则通过红外光谱、紫外吸收光谱、荧光光谱等进行表征。分子量信息通过超滤离心管、尺寸排阻色谱等获得。胞外多聚物的亲疏水性则可以利用亲疏水树脂来进行分离检测[53]。这些传统的表征方法显著提升了对胞外多聚物与重金属相互作用的认识,但是物理化学的提取和保存方法也在一定程度上破坏了胞外多聚物的组成和结构,无法准确预测生物膜中胞外多聚物对无机汞形态的作用。
新表征技术的发展为原位考察胞外多聚物组成及其与重金属相互作用提供了条件。利用荧光染料对特定组分进行染色,通过激光扫描共聚焦显微镜(Confocal laser scanning microscope, CLSM)可以原位地观察生物膜中胞外多聚物特定组分的相对含量和空间分布情况[86]。红外光谱(Fourier transform infrared spectroscopy, FTIR)成像、拉曼光谱(Raman spectroscopy)成像、表面等离子体共振(Surface plasmon resonance, SPR)成像等可以用来表征官能基团的分布。将生物膜样品固定、包埋后,纳米二次离子质谱技术(Nanoscale secondary ion mass spectrometry, NanoSIMS)、基于同步辐射的扫描透射X射线显微镜(Scanning transmission X-ray microscopy, STXM)等先进技术也可以用来原位观察生物膜的物质组成和结构特征[87-88]。此外,耗散型石英晶体微天平(Quartz crystal microbalance with dissipation monitoring, QCM-D)、原子力显微镜(Atomic force microscopy, AFM)等是原位表征生物膜与不同形态无机汞相互作用的有力工具。
胞外多聚物的空间结构对不同形态无机汞在生物膜中的转化、传质等过程可能也会产生影响。光散射技术常用来表征聚合物的空间结构特征,例如,Dogsa等利用小角X射线散射技术(Small angle X-ray scattering, SAXS)研究了不同pH条件下细菌胞外多聚物的结构[89];Benigar等通过SAXS、静态光散射(Static light scattering, SLS)和动态光散射(Dynamic light scattering, DLS)考察了多糖的空间结构 [90]。此外,荧光相关光谱(Fluorescence correlation spectroscopy, FCS)结合共聚焦显微镜常用来考察不同形态重金属在生物膜中的扩散传质过程。FCS通过测定溶液中微区内被荧光探针标记了的各形态重金属因布朗运动而产生的荧光涨落现象,分析荧光涨落的相关函数而获得自扩散相关系数等参数信息[91]。
-
微生物活性表示的是微生物体内生理代谢活动过程的状态水平。通常认为生长速率快的微生物具有较高的代谢活性,在微生物汞甲基化研究中,普遍通过测定光密度(OD)、细胞数、总蛋白量等指标来描述生长速率快慢,进而间接表示甲基化微生物的活性。这些指标技术要求低,易于操作,但是也存在难以区分活性和非活性细胞的问题,不能准确反映活性微生物的代谢活动水平。酶活性也是常用的评估微生物活性的指标之一,常用的指示微生物代谢活性的酶包括脲酶、过氧化氢酶、脱氢酶、磷酸酶等[92],但是微生物酶活性也容易受到提取和测试环境条件的影响。蛋白质是生理代谢活动的主要承担者,核糖体RNA(rRNA)与蛋白质的合成密切相关,因此利用RNA来反映微生物的活性已成为一种重要的研究手段[93]。诸多研究表明,rRNA的含量与微生物的生长速率、代谢活性具有高度的相关性,而且只有活性微生物体内才存在高含量的rRNA[94]。通过转录组学,测定rRNA的含量,除了能获得代谢活性信息,还能获得活性微生物群落结构信息,这对于研究生物膜中微生物的活性具有显著优势。
环境条件显著影响微生物的活性,准确定量地描述生物膜内部微环境条件的变化,是考察生物膜中汞甲基化微生物活性的重要内容。目前,以微电极系统为代表的微传感器已经成为原位、实时、定量表征生物膜内部微环境的有力工具之一[95-97]。微电极系统可以转换为温度微电极、pH微电极、氧化还原电位微电极、氧气微电极等,通过对生物膜进行穿刺,进而获得相应内部微环境的状况。微电极的尖端直径一般在微米级范围,确保穿刺不会破坏生物膜内部微环境,同时微电极系统反应灵敏且检测限低,可以检测生物膜内部微环境的瞬间动态变化。
随着高通量核酸测序技术的发展,以宏基因组学为代表的宏组学已经成为研究微生物种群间相互作用机制的热门技术。宏基因组学是以环境样本中微生物群体基因组为研究对象,通过测序分析、功能基因筛选等手段,研究微生物种群结构、功能活性、相互作用关系等[98]。随着决定汞甲基化能力的hgcAB基因的发现,利用宏基因组学手段研究不同汞甲基化微生物之间群落组成和相互作用成为可能。例如,Liu等结合宏基因组学、PacBio测序和实时定量PCR方法测定了稻田土壤中hgcAB基因的分布丰度及其系统发育关系,发现铁还原菌和产甲烷菌共同驱动了稻田土壤中的甲基汞生成过程[79]。另外,荧光原位杂交技术(Fluorescence in situ hybridization, FISH)由于兼备分子生物学准确性和显微镜可视性的特点,在微生物群落结构、种群相互作用、种群中特定微生物代谢活性等研究中也得到较为广泛的应用[99]。FISH技术是以荧光染料标记的特异寡核苷酸片段为探针,与微生物的DNA或RNA分子进行原位杂交,在特定激发波长下,通过荧光检测系统对目标微生物进行鉴别、跟踪和观察。因此,可利用FISH技术配合激光扫描共聚焦显微镜原位观察不同种群汞甲基化微生物在生物膜中的空间分布和相互作用情况。
-
综上所述,微生物汞甲基化的生化分子路径、无机汞的化学形态和甲基化微生物的活性是影响环境生物膜中汞甲基化的三个关键因素,然而目前对这三个因素的认识仍有较大不足,存在诸多亟待解答的问题:(1)与浮游态微生物相比,生物膜中各种类微生物的汞甲基化生化路径有何区别?微生物之间的群体交流是否会导致厌氧菌汞甲基化的生化路径发生改变?(2)生物膜胞外多聚物的组成和结构如何影响无机汞的络合、还原、沉淀等形态转化过程?环境因素如何影响胞外多聚物的组成和结构?(3)生物膜内部微环境如何影响汞甲基化厌氧菌的活性?不同种类厌氧菌之间的相互作用如何对汞甲基化过程产生影响?
针对生物膜汞甲基化研究中仍然存在的问题,结合目前传统的和新兴的研究方法和技术手段,未来可开展如下几方面的研究:(1)通过阻断代谢途径,利用代谢组学、代谢流分析技术等方法研究生物膜中汞甲基化的生化分子机制。(2)通过异位表征和原位表征相结合的技术手段,考察甲基化微生物胞外多聚物影响无机汞化学形态的机制。(3)利用微电极系统考察生物膜内部微环境,采用宏基因组学、荧光原位杂交技术等研究生物膜内甲基化微生物间相互作用的机制。(4)考察胞外多聚物作用下形成的含汞化合物(如,溶解态络合物、纳米颗粒物、零价单质等)在生物膜中形成甲基汞的作用机制以及环境扰动对甲基化过程的影响。
微生物汞甲基化的作用机制是汞污染研究领域的热点和难点问题,而生物膜这一具有重要环境意义的潜在甲基汞污染源需要给予重点关注。系统、深刻认识生物膜中汞甲基化过程的作用机制和主控因子,有助于实现环境中甲基汞生成和累积过程的准确预测和有效阻控。同时,生物膜是微生物长期进化和适应环境的结果,使得微生物对外部环境变化具有较强的耐受能力。生物膜中厌氧菌驱动的汞甲基化过程能够在不适于浮游态微生物生存的环境甚至极端环境中发生,为探索汞污染可能危及的未知区域提供新的思路。
环境生物膜中的汞甲基化
Mercury methylation in environmental biofilms
-
摘要: 汞是严重危害食品安全及人体健康的全球性重金属污染物,揭示具有强生物毒性的甲基汞的生成和累积机制是汞污染研究领域的热点和难点。生物膜中无机汞的甲基化是环境中甲基汞污染的主要来源和甲基汞进入食物链的起点,但是目前学术界对汞甲基化机制的认知多基于浮游态纯菌实验,无法准确模拟生物膜中汞的甲基化过程。本文从生物膜中汞甲基化的生化分子路径、无机汞的化学形态和甲基化微生物的活性这三个决定汞甲基化过程的关键因素出发,详细阐述了目前生物膜中汞甲基化的研究现状、存在问题以及拟解决问题的技术手段,并对未来在该领域的研究工作进行了展望。生物膜中汞甲基化机制的研究可为深入理解甲基汞的环境累积过程,提高汞污染风险分析的准确性,开发有效的汞污染风险防控技术提供科学依据和数据支撑。Abstract: Mercury is a heavy metal contaminant that endangers food safety and human health globally. Understanding the mechanisms that control the production and accumulation of the potent toxin, methylmercury, has been the research focus of mercury pollution. Methylation of inorganic mercury in biofilms significantly contributes to methylmercury contamination in the natural environment, and serves as the entry point of methylmercury accumulation in the food web. However, current understanding of the mechanisms governing mercury methylation is mainly originated from experiments using planktonic cultures of pure strains, which do not properly simulate mercury methylation processes in biofilms. Here, we synthesized the current understanding and remaining challenges regarding the principal biochemical pathways responsible for mercury methylation, chemical speciation of inorganic mercury and the activity of microorganism in biofilms, the three key factors affecting microbial mercury methylation. Technical approaches for further solving the key questions of mercury methylation in environmental biofilms and future perspectives of this research area were also discussed. Research on mercury methylation in biofilms will help improve the mechanistic understanding of the methylmercury accumulation processes in the environment, and provide insights to accurate risk analysis and effective remediation strategies of mercury pollution.
-
Key words:
- biofilm /
- mercury methylation /
- biochemical pathway /
- mercury speciation /
- microbial activity
-

-
表 1 文献报道的生物膜中汞甲基化速率常数和甲基化效率
Table 1. Mercury methylation rate constant and methylation efficiency in biofilms reported in literature
生物膜类型
Biofilm type生物膜载体
Biofilm carrier甲基化速率常数/d− 1
Methylation rate
constant (km)甲基化效率/%
Methylation efficiency
(MeHg/THg)文献
Reference混菌生物膜 凤眼莲、丁香蓼等 2.32 × 10− 4—3.45 × 10− 4 0.9—8.3 Achá et al., 2011 [23] 穿叶眼子菜、伊乐藻、芦苇等 1.50 × 10− 3—1.80 × 10− 2 0.3—3.5 Hamelin et al., 2011 [21] 狸藻、小莎草等 2.01 × 10− 4—4.15 × 10− 2 0.1—10.0 Cleckner et al., 1999 [18] 穿叶眼子菜、藨草、香蒲等 2.00 × 10− 3—0.14 0.4—23.9 Hamelin et al., 2015 [12] 槐叶萍、凤眼莲、金鱼藻等 1.91 × 10− 2—0.17 0.2—17.0 Mauro et al., 2002 [24] 凤眼莲 2.62 × 10− 2—0.17 5.1—29.2 Lázaro et al., 2013 [22] 芦苇 4.00 × 10− 2—0.18 3.9—23.7 Bouchet et al., 2018 [25] 凤眼莲、膜稃草、丁香蓼等 1.00 × 10− 3—0.18 0.2—36.1 Correia et al., 2012 [26] 凤眼莲 6.30 × 10− 2—0.28 6.1—24.6 Lázaro et al., 2019 [27] 凤眼莲 6.40 × 10− 2—0.29 6.2—25.6 Lázaro et al., 2016 [28] 狐尾藻、凤眼莲 1.61 × 10− 2—0.36 1.6—30.2 Guimarães et al., 2006 [29] 凤眼莲 0.16—0.47 15.3—37.3 Lázaro et al., 2018 [30] 多孔玻璃盘 1.48 × 10− 6—1.08 × 10− 4 < 0.1 Schwartz et al., 2019 [31] 聚丙烯网 2.33 × 10− 5—1.25 × 10− 4 < 0.2 Olsen et al., 2016 [32] 防水木板 5.00 × 10− 5—1.50 × 10− 3 0.3—1.4 Huguet et al., 2010 [33] 岩石 3.84 × 10− 4—1.42 × 10− 2 0.1—0.8 Desrosiers et al., 2006 [20] 岩石 3.50 × 10− 2—0.20 0.2—9.5 Buckman et al., 2015 [34] 纯菌生物膜 玻璃载玻片 3.33 × 10− 2—0.19 6.6—17.3 Lin and Jay, 2007 [14] 玻璃载玻片 6.29 × 10− 2—0.19 3.1—30.6 Lin et al., 2013 [35] -
[1] DRISCOLL C T, MASON R P, CHAN H M, et al. Mercury as a global pollutant: Sources, pathways, and effects [J]. Environmental Science & Technology, 2013, 47(10): 4967-4983. [2] TSUI T M, LIU S, BRASSO R L, et al. Controls of methylmercury bioaccumulation in forest floor food webs [J]. Environmental Science & Technology, 2019, 53(5): 2434-2440. [3] TANG W, LIU Y, GUAN W, et al. Understanding mercury methylation in the changing environment: Recent advances in assessing microbial methylators and mercury bioavailability [J]. Science of the Total Environment, 2020, 714: 136827. doi: 10.1016/j.scitotenv.2020.136827 [4] LIU M, ZHANG Q, CHENG M, et al. Rice life cycle-based global mercury biotransport and human methylmercury exposure [J]. Nature Communications, 2019, 10(1): 5164. doi: 10.1038/s41467-019-13221-2 [5] ZHANG H, FENG X, LARSSEN T, et al. In inland China, rice, rather than fish, is the major pathway for methylmercury exposure [J]. Environmental Health Perspectives, 2010, 118(9): 1183-1188. doi: 10.1289/ehp.1001915 [6] KERIN E J, GILMOUR C C, RODEN E, et al. Mercury methylation by dissimilatory iron-reducing bacteria [J]. Applied and Environmental Microbiology, 2006, 72(12): 7919-7921. doi: 10.1128/AEM.01602-06 [7] FLEMMING H-C, WUERTZ S. Bacteria and archaea on Earth and their abundance in biofilms [J]. Nature Reviews Microbiology, 2019, 17(4): 247-260. doi: 10.1038/s41579-019-0158-9 [8] FLEMMING H C, WINGENDER J, SZEWZYK U, et al. Biofilms: An emergent form of bacterial life [J]. Nature Reviews Microbiology, 2016, 14(9): 563-575. doi: 10.1038/nrmicro.2016.94 [9] BRANFIREUN B A, COSIO C, POULAIN A J, et al. Mercury cycling in freshwater systems-an updated conceptual model [J]. Science of the Total Environment, 2020, 745: 140906. doi: 10.1016/j.scitotenv.2020.140906 [10] DRANGUET P, FAUCHEUR S L, SLAVEYKOVA V I. Mercury bioavailability, transformations, and effects on freshwater biofilms [J]. Environmental Toxicology and Chemistry, 2017, 36(12): 3194-3205. doi: 10.1002/etc.3934 [11] HU H, LIN H, ZHENG W, et al. Oxidation and methylation of dissolved elemental mercury by anaerobic bacteria [J]. Nature Geoscience, 2013, 6(9): 751-754. doi: 10.1038/ngeo1894 [12] KONOPKA A. What is microbial community ecology? [J]. The ISME Journal, 2009, 3(11): 1223-1230. doi: 10.1038/ismej.2009.88 [13] HAMELIN S, PLANAS D, AMYOT M. Mercury methylation and demethylation by periphyton biofilms and their host in a fluvial wetland of the St. Lawrence River (QC, Canada) [J]. Science of the Total Environment, 2015, 512: 464-471. [14] LIN C, JAY J A. Mercury methylation by planktonic and biofilm cultures of Desulfovibrio desulfuricans [J]. Environmental Science & Technology, 2007, 41(19): 6691-6697. [15] VERT M,DOI Y,HELLWICH K,et al. Terminology for biorelated polymers and applications (IUPAC Recommendations 2012) [J]. Pure and Applied Chemistry, 2012, 84(2): 377-410. doi: 10.1351/PAC-REC-10-12-04 [16] BRANDA S S, VIK Å, FRIEDMAN L, et al. Biofilms: The matrix revisited [J]. Trends in Microbiology, 2005, 13(1): 20-26. doi: 10.1016/j.tim.2004.11.006 [17] BUFFLE J, WILKINSON K J, VAN LEEUWEN H P. Chemodynamics and bioavailability in natural waters [J]. Environmental Science & Technology, 2009, 43(19): 7170-7174. [18] CLECKNER L B, GILMOUR C C, HURLEY J P, et al. Mercury methylation in periphyton of the Florida Everglades [J]. Limnology and Oceanography, 1999, 44(7): 1815-1825. doi: 10.4319/lo.1999.44.7.1815 [19] BELL A H, SCUDDER B C. Mercury accumulation in periphyton of eight river ecosystems [J]. Journal of the American Water Resources Association, 2007, 43(4): 957-968. doi: 10.1111/j.1752-1688.2007.00078.x [20] DESROSIERS M, PLANAS D, MUCCI A. Mercury methylation in the epilithon of boreal shield aquatic ecosystems [J]. Environmental Science & Technology, 2006, 40(5): 1540-1546. [21] HAMELIN S, AMYOT M, BARKAY T, et al. Methanogens: principal methylators of mercury in lake periphyton [J]. Environmental Science & Technology, 2011, 45(18): 7693-7700. [22] LÁZARO W L, GUIMARÃES J R D, IGNÁCIO A R A, et al. Cyanobacteria enhance methylmercury production: A hypothesis tested in the periphyton of two lakes in the Pantanal floodplain, Brazil [J]. Science of the Total Environment, 2013, 456: 231-238. [23] ACHÁ D, HINTELMANN H, YEE J. Importance of sulfate reducing bacteria in mercury methylation and demethylation in periphyton from Bolivian Amazon region [J]. Chemosphere, 2011, 82(6): 911-916. doi: 10.1016/j.chemosphere.2010.10.050 [24] MAURO J, GUIMARÃES J, HINTELMANN H, et al. Mercury methylation in macrophytes, periphyton, and water - comparative studies with stable and radio-mercury additions [J]. Analytical and Bioanalytical Chemistry, 2002, 374(6): 983-989. doi: 10.1007/s00216-002-1534-1 [25] BOUCHET S, GOÑI-URRIZA M, MONPERRUS M, et al. Linking microbial activities and low-molecular-weight thiols to Hg methylation in biofilms and periphyton from high-altitude tropical lakes in the Bolivian altiplano [J]. Environmental Science & Technology, 2018, 52(17): 9758-9767. [26] CORREIA R R S, MIRANDA M R, GUIMARÃES J R D. Mercury methylation and the microbial consortium in periphyton of tropical macrophytes: Effect of different inhibitors [J]. Environmental Research, 2012, 112: 86-91. doi: 10.1016/j.envres.2011.11.002 [27] LÁZARO W L, DÍEZ S, BRAVO A G, et al. Cyanobacteria as regulators of methylmercury production in periphyton [J]. Science of the Total Environment, 2019, 668: 723-729. doi: 10.1016/j.scitotenv.2019.02.233 [28] LÁZARO W L, DÍEZ S, SILVA C J D, et al. Waterscape determinants of net mercury methylation in a tropical wetland [J]. Environmental Research, 2016, 150: 438-445. doi: 10.1016/j.envres.2016.06.028 [29] GUIMARÃES J R D, MAURO J B N, MEILI M, et al. Simultaneous radioassays of bacterial production and mercury methylation in the periphyton of a tropical and a temperate wetland [J]. Journal of Environmental Management, 2006, 81(2): 95-100. doi: 10.1016/j.jenvman.2005.09.023 [30] LÁZARO W L, DÍEZ S, SILVA C J D, et al. Seasonal changes in peryphytic microbial metabolism determining mercury methylation in a tropical wetland [J]. Science of the Total Environment, 2018, 627: 1345-1352. doi: 10.1016/j.scitotenv.2018.01.186 [31] SCHWARTZ G E, OLSEN T A, MULLER K A, et al. Ecosystem controls on methylmercury production by periphyton biofilms in a contaminated stream: implications for predictive modeling [J]. Environmental Toxicology and Chemistry, 2019, 38(11): 2426-2435. doi: 10.1002/etc.4551 [32] OLSEN T A, BRANDT C C, BROOKS S C. Periphyton biofilms influence net methylmercury production in an industrially contaminated system [J]. Environmental Science & Technology, 2016, 50(20): 10843-10850. [33] HUGUET L, CASTELLE S, SCHÄFER J, et al. Mercury methylation rates of biofilm and plankton microorganisms from a hydroelectric reservoir in French Guiana [J]. Science of the Total Environment, 2010, 408(6): 1338-1348. doi: 10.1016/j.scitotenv.2009.10.058 [34] BUCKMAN K L, MARVIN-DIPASQUALE M, TAYLOR V F, et al. Influence of a chlor-alkali superfund site on mercury bioaccumulation in periphyton and low-trophic level fauna [J]. Environmental Toxicology and Chemistry, 2015, 34(7): 1649-1658. doi: 10.1002/etc.2964 [35] LIN T Y, KAMPALATH R A, LIN C, et al. Investigation of mercury methylation pathways in biofilm versus planktonic cultures of Desulfovibrio desulfuricans [J]. Environmental Science & Technology, 2013, 47(11): 5695-5702. [36] HAMELIN S, PLANAS D, AMYOT M. Spatio-temporal variations in biomass and mercury concentrations of epiphytic biofilms and their host in a large river wetland (Lake St. Pierre, Qc, Canada) [J]. Environmental Pollution, 2015, 197: 221-230. doi: 10.1016/j.envpol.2014.11.007 [37] BRAVO A G, COSIO C. Biotic formation of methylmercury: A bio-physico-chemical conundrum [J]. Limnology and Oceanography, 2020, 65(5): 1010-1027. doi: 10.1002/lno.11366 [38] JENSEN S, JERNELÖV A. Biological methylation of mercury in aquatic organisms [J]. Nature, 1969, 223(5207): 753-754. doi: 10.1038/223753a0 [39] WOOD J M. Biological cycles for toxic elements in the environment [J]. Science, 1974, 183(4129): 1049-1052. doi: 10.1126/science.183.4129.1049 [40] COMPEAU G C, BARTHA R. Sulfate-reducing bacteria: Principal methylators of mercury in anoxic estuarine sediment [J]. Applied and Environmental Microbiology, 1985, 50(2): 498-502. doi: 10.1128/aem.50.2.498-502.1985 [41] FLEMING E J, MACK E E, GREEN P G, et al. Mercury methylation from unexpected sources: Molybdate-inhibited freshwater sediments and an iron-reducing bacterium [J]. Applied and Environmental Microbiology, 2006, 72(1): 457-464. doi: 10.1128/AEM.72.1.457-464.2006 [42] CHOI S C, CHASE T, BARTHA R. Enzymatic catalysis of mercury methylation by Desulfovibrio desulfuricans LS [J]. Applied and Environmental Microbiology, 1994, 60(4): 1342-1346. doi: 10.1128/aem.60.4.1342-1346.1994 [43] CHOI S C, CHASE T, BARTHA R. Metabolic pathways leading to mercury methylation in Desulfovibrio desulfuricans LS [J]. Applied and Environmental Microbiology, 1994, 60(11): 4072-4077. doi: 10.1128/aem.60.11.4072-4077.1994 [44] EKSTROM E B, MOREL F M M, BENOIT J M. Mercury methylation independent of the acetyl-coenzyme A pathway in sulfate-reducing bacteria [J]. Applied and Environmental Microbiology, 2003, 69(9): 5414-5422. doi: 10.1128/AEM.69.9.5414-5422.2003 [45] EKSTROM E B, MOREL F M M. Cobalt limitation of growth and mercury methylation in sulfate-reducing bacteria [J]. Environmental Science & Technology, 2008, 42(1): 93-99. [46] PARKS J M, JOHS A, PODAR M, et al. The genetic basis for bacterial mercury methylation [J]. Science, 2013, 339(6125): 1332-1335. doi: 10.1126/science.1230667 [47] GILMOUR C C, PODAR M, BULLOCK A L, et al. Mercury methylation by novel microorganisms from new environments [J]. Environmental Science & Technology, 2013, 47(20): 11810-11820. [48] LATTIF A A, CHANDRA J, CHANG J, et al. Proteomics and pathway mapping analyses reveal phase-dependent over-expression of proteins associated with carbohydrate metabolic pathways in Candida albicans biofilms [J]. The Open Proteomics Journal, 2008, 1: 5-26. doi: 10.2174/1875039700801010005 [49] RESCH A, ROSENSTEIN R, NERZ C, et al. Differential gene expression profiling of Staphylococcus aureus cultivated under biofilm and planktonic conditions [J]. Applied and Environmental Microbiology, 2005, 71(5): 2663-2676. doi: 10.1128/AEM.71.5.2663-2676.2005 [50] WHITELEY M, DIGGLE S P, GREENBERG E P. Progress in and promise of bacterial quorum sensing research [J]. Nature, 2017, 551(7680): 313-320. doi: 10.1038/nature24624 [51] YIN W, CAI X, MA H, et al. A decade of research on the second messenger c-di-AMP [J]. FEMS Microbiology Reviews, 2020, 44(6): 701-724. doi: 10.1093/femsre/fuaa019 [52] FLEMMING H C, WINGENDER J. The biofilm matrix [J]. Nature Reviews Microbiology, 2010, 8(9): 623-633. doi: 10.1038/nrmicro2415 [53] SHENG G, YU H, LI X. Extracellular polymeric substances (EPS) of microbial aggregates in biological wastewater treatment systems: A review [J]. Biotechnology Advances, 2010, 28(6): 882-894. doi: 10.1016/j.biotechadv.2010.08.001 [54] WEI L, LI Y, NOGUERA D R, et al. Adsorption of Cu2+ and Zn2+ by extracellular polymeric substances (EPS) in different sludges: Effect of EPS fractional polarity on binding mechanism [J]. Journal of Hazardous Materials, 2017, 321: 473-483. doi: 10.1016/j.jhazmat.2016.05.016 [55] ZHANG D, PAN X, MOSTOFA K M G, et al. Complexation between Hg(II) and biofilm extracellular polymeric substances: An application of fluorescence spectroscopy [J]. Journal of Hazardous Materials, 2010, 175(40546): 359-365. [56] ZHANG P, CHEN Y, PENG M, et al. Extracellular polymeric substances dependence of surface interactions of Bacillus subtilis with Cd2+ and Pb2+: An investigation combined with surface plasmon resonance and infrared spectra [J]. Colloids and Surfaces B: Biointerfaces, 2017, 154: 357-364. doi: 10.1016/j.colsurfb.2017.03.046 [57] BENOIT J M, GILMOUR C C, MASON R P, et al. Sulfide controls on mercury speciation and bioavailability to methylating bacteria in sediment pore waters [J]. Environmental Science & Technology, 1999, 33(6): 951-957. [58] BENOIT J M, MASON R P, GILMOUR C C. Estimation of mercury‐sulfide speciation in sediment pore waters using octanol—water partitioning and implications for availability to methylating bacteria [J]. Environmental Toxicology and Chemistry, 1999, 18(10): 2138-2141. [59] GRAHAM A M, AIKEN G R, GILMOUR C C. Dissolved organic matter enhances microbial mercury methylation under sulfidic conditions [J]. Environmental Science & Technology, 2012, 46(5): 2715-2723. [60] ZHANG T, KIM B, LEVARD C, et al. Methylation of mercury by bacteria exposed to dissolved, nanoparticulate, and microparticulate mercuric sulfides [J]. Environmental Science & Technology, 2012, 46(13): 6950-6958. [61] COLOMBO M J, HA J, REINFELDER J R, et al. Anaerobic oxidation of Hg(0) and methylmercury formation by Desulfovibrio desulfuricans ND132 [J]. Geochimica et Cosmochimica Acta, 2013, 112: 166-177. doi: 10.1016/j.gca.2013.03.001 [62] DASH H R, BASU S, DAS S. Evidence of mercury trapping in biofilm-EPS and mer operon-based volatilization of inorganic mercury in a marine bacterium Bacillus cereus BW-201B [J]. Archives of Microbiology, 2017, 199(3): 445-455. doi: 10.1007/s00203-016-1317-2 [63] DASH H R, DAS S. Interaction between mercuric chloride and extracellular polymers of biofilm-forming mercury resistant marine bacterium Bacillus thuringiensis PW-05 [J]. RSC Advances, 2016, 6(111): 109793-109802. doi: 10.1039/C6RA21069D [64] LECLERC M, PLANAS D, AMYOT M. Relationship between extracellular low-molecular-weight thiols and mercury species in natural lake periphytic biofilms [J]. Environmental Science & Technology, 2015, 49(13): 7709-7716. [65] SCHAEFER J K, MOREL F M. High methylation rates of mercury bound to cysteine by Geobacter sulfurreducens [J]. Nature Geoscience, 2009, 2(2): 123-126. doi: 10.1038/ngeo412 [66] CHENG J, ZHAO W, LIU Y, et al. Adsorption properties and gaseous mercury transformation rate of natural biofilm [J]. Bulletin of Environmental Contamination and Toxicology, 2008, 81(5): 516-520. doi: 10.1007/s00128-008-9526-2 [67] KANG F, ALVAREZ P J J, ZHU D. Microbial extracellular polymeric substances reduce Ag+ to silver nanoparticles and antagonize bactericidal activity [J]. Environmental Science & Technology, 2014, 48(1): 316-322. [68] KANG F, QU X, ALVAREZ P J J, et al. Extracellular saccharide-mediated reduction of Au3+ to gold nanoparticles: New insights for heavy metals biomineralization on microbial surfaces [J]. Environmental Science & Technology, 2017, 51(5): 2776-2785. [69] LABRENZ M, DRUSCHEL G K, THOMSEN-EBERT T, et al. Formation of sphalerite (ZnS) deposits in natural biofilms of sulfate-reducing bacteria [J]. Science, 2000, 290(5497): 1744-1747. doi: 10.1126/science.290.5497.1744 [70] ZHANG Z, SI R, LV J, et al. Effects of extracellular polymeric substances on the formation and methylation of mercury sulfide nanoparticles [J]. Environmental Science & Technology, 2020, 54(13): 8061-8071. [71] WANG L, WANG L, REN X, et al. pH dependence of structure and surface properties of microbial EPS [J]. Environmental Science & Technology, 2012, 46(2): 737-744. [72] WANG L, WANG L, YE X, et al. Spatial configuration of extracellular polymeric substances of Bacillus megaterium TF10 in aqueous solution [J]. Water Research, 2012, 46(11): 3490-3496. doi: 10.1016/j.watres.2012.03.054 [73] IKUMA K, DECHO A W, LAU B L. When nanoparticles meet biofilms—interactions guiding the environmental fate and accumulation of nanoparticles [J]. Frontiers in Microbiology, 2015, 6: 591. [74] HALL-STOODLEY L, COSTERTON J W, STOODLEY P. Bacterial biofilms: From the natural environment to infectious diseases [J]. Nature Reviews Microbiology, 2004, 2(2): 95-108. doi: 10.1038/nrmicro821 [75] ITO T, NIELSEN J L, OKABE S, et al. Phylogenetic identification and substrate uptake patterns of sulfate-reducing bacteria inhabiting an oxic-anoxic sewer biofilm determined by combining microautoradiography and fluorescent in situ hybridization [J]. Applied and Environmental Microbiology, 2002, 68(1): 356-364. doi: 10.1128/AEM.68.1.356-364.2002 [76] BRAVO A G, BOUCHET S, TOLU J, et al. Molecular composition of organic matter controls methylmercury formation in boreal lakes [J]. Nature Communications, 2017, 8: 14255. doi: 10.1038/ncomms14255 [77] GUILLEMETTE F, MCCALLISTER S L, GIORGIO P A D. Selective consumption and metabolic allocation of terrestrial and algal carbon determine allochthony in lake bacteria [J]. The ISME Journal, 2016, 10(6): 1373-1382. doi: 10.1038/ismej.2015.215 [78] YU R, FLANDERS J R, MACK E E, et al. Contribution of coexisting sulfate and iron reducing bacteria to methylmercury production in freshwater river sediments [J]. Environmental Science & Technology, 2012, 46(5): 2684-2691. [79] LIU Y, JOHS A, BI L, et al. Unraveling microbial communities associated with methylmercury production in paddy soils [J]. Environmental Science & Technology, 2018, 52(22): 13110-13118. [80] YU R, REINFELDER J R, HINES M E, et al. Syntrophic pathways for microbial mercury methylation [J]. The ISME Journal, 2018, 12(7): 1826-1835. doi: 10.1038/s41396-018-0106-0 [81] BORREL G, ADAM P S, GRIBALDO S. Methanogenesis and the wood-ljungdahl pathway: An ancient, versatile, and fragile association [J]. Genome Biology and Evolution, 2016, 8(6): 1706-1711. doi: 10.1093/gbe/evw114 [82] CHEAH Y E, YOUNG J D. Isotopically nonstationary metabolic flux analysis (INST-MFA): Putting theory into practice [J]. Current Opinion in Biotechnology, 2018, 54: 80-87. doi: 10.1016/j.copbio.2018.02.013 [83] LONG C P, ANTONIEWICZ M R. High-resolution 13C metabolic flux analysis [J]. Nature Protocols, 2019, 14(10): 2856-2877. doi: 10.1038/s41596-019-0204-0 [84] PAPENFORT K, BASSLER B L. Quorum sensing signal-response systems in Gram-negative bacteria [J]. Nature Reviews Microbiology, 2016, 14(9): 576-588. doi: 10.1038/nrmicro.2016.89 [85] HAO Y, WINANS S C, GLICK B R, et al. Identification and characterization of new LuxR/LuxI-type quorum sensing systems from metagenomic libraries [J]. Environmental Microbiology, 2010, 12(1): 105-117. doi: 10.1111/j.1462-2920.2009.02049.x [86] REICHHARDT C, PARSEK M R. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization [J]. Frontiers in Microbiology, 2019, 10: 677. doi: 10.3389/fmicb.2019.00677 [87] CHADWICK G L, OTERO F J, GRALNICK J A, et al. NanoSIMS imaging reveals metabolic stratification within current-producing biofilms [J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(41): 20716-20724. doi: 10.1073/pnas.1912498116 [88] NEU T R, MANZ B, VOLKE F, et al. Advanced imaging techniques for assessment of structure, composition and function in biofilm systems [J]. FEMS Microbiology Ecology, 2010, 72(1): 1-21. doi: 10.1111/j.1574-6941.2010.00837.x [89] DOGSA I, KRIECHBAUM M, STOPAR D, et al. Structure of bacterial extracellular polymeric substances at different pH values as determined by SAXS [J]. Biophysical Journal, 2005, 89(4): 2711-2720. doi: 10.1529/biophysj.105.061648 [90] BENIGAR E, DOGSA I, STOPAR D, et al. Structure and dynamics of a polysaccharide matrix: Aqueous solutions of bacterial levan [J]. Langmuir, 2014, 30(14): 4172-4182. doi: 10.1021/la500830j [91] HINK M A. Fluorescence correlation spectroscopy [J]. Methods of Molecular Biology, 2015, 1251(1251): 135-150. [92] HUANG D, XUE W, ZENG G, et al. Immobilization of Cd in river sediments by sodium alginate modified nanoscale zero-valent iron: Impact on enzyme activities and microbial community diversity [J]. Water Research, 2016, 106: 15-25. doi: 10.1016/j.watres.2016.09.050 [93] BLAZEWICZ S J, BARNARD R L, DALY R A, et al. Evaluating rRNA as an indicator of microbial activity in environmental communities: Limitations and uses [J]. The ISME Journal, 2013, 7(11): 2061-2068. doi: 10.1038/ismej.2013.102 [94] RAMOS C, MØLBAK L, MOLIN S. Bacterial activity in the rhizosphere analyzed at the single-cell level by monitoring ribosome contents and synthesis rates [J]. Applied and Environmental Microbiology, 2000, 66(2): 801-809. doi: 10.1128/AEM.66.2.801-809.2000 [95] CHENG L, HOUSE M W, WEISS W J, et al. Monitoring sulfide-oxidizing biofilm activity on cement surfaces using non-invasive self-referencing microsensors [J]. Water Research, 2016, 89: 321-329. doi: 10.1016/j.watres.2015.11.066 [96] DHAR B R, SIM J, RYU H, et al. Microbial activity influences electrical conductivity of biofilm anode [J]. Water Research, 2017, 127: 230-238. doi: 10.1016/j.watres.2017.10.028 [97] WANG J, CHEN Y, DONG Y, et al. A new method to measure and model dynamic oxygen microdistributions in moving biofilms [J]. Environmental Pollution, 2017, 229: 199-209. doi: 10.1016/j.envpol.2017.05.062 [98] BAHRAM M, HILDEBRAND F, FORSLUND S K, et al. Structure and function of the global topsoil microbiome [J]. Nature, 2018, 560(7717): 233-237. doi: 10.1038/s41586-018-0386-6 [99] WAGNER M, HAIDER S. New trends in fluorescence in situ hybridization for identification and functional analyses of microbes [J]. Current Opinion in Biotechnology, 2012, 23(1): 96-102. doi: 10.1016/j.copbio.2011.10.010 -



 下载:
下载: