-
阴离子型聚丙烯酰胺(HPAM)是由单体丙烯酰胺(AM)和丙烯酸(AA)在碱性条件下共聚而成的一种线性水溶性聚合物,分子量在300万—2000万区间[1 − 2]. 低浓度HPAM水溶液具有较高黏度[3 − 4]. HPAM作为絮凝剂而广泛用于煤泥水澄清处理[5],所以选煤循环水中长期累积了大量的HPAM,这将降低煤泥对浮选药剂的吸附能力,不利于浮选[6]. 另外,含HPAM循环水外排可能会造成环境污染[7]. 因此,急需研发对煤泥水中HPAM脱除的有效方法. 生物降解是脱除HPAM的有效方法,具有环境友好、条件温和、无二次污染等优点[8]. 但生物降解难点在于获得能够分泌高活性降解酶优势菌株,目前从聚合物驱水样[9]、土壤、活性淤泥[10]等长期受HPAM污染环境中分离菌株分泌的降解酶活性和稳定性普遍都偏低,这将导致降解效率欠佳[11]. HPAM降解效率可能受到酶与底物相互作用的影响,并且HPAM的结构也会影响酶降解[12]. 因此,需要深入了解生物酶如何与底物相互作用,为定向突变、基因重组等实验提供理论参考,同时拓宽对生物酶催化机制的认识. 分子对接和分子动力学(MD)模拟已被证明是在原子及分子水平上探究酶与底物微观相互作用的可靠技术[13 − 14],尤其MD可探索酶-底物复合物随模拟时间的动态变化.
在真核生物中只有黄孢原毛平革菌(P. chrysosporium)已被证明可以使用HPAM作为碳源. 在营养物质有限的条件下由P. chrysosporium产生的胞外过氧化物酶可以从各种环境有机污染物中提取一个电子,从而引发自由基介导的许多难降解化合物的非特异性降解[15]. Sutherland等[16]利用14C标记底物研究了P. chrysosporium对不溶性交联HPAM的降解,将2,3-(14C)-AA和2,3-(14C)-AM掺入聚合物中,通过测量释放CO2的放射性来监测矿化度. 结果发现P. chrysosporium在氮源受限条件下以葡萄糖为碳源、HPAM的初始浓度为20 mg·L−1的培养基中生长,同时可分泌锰过氧化物酶(MnP)来解聚HPAM为水溶性代谢产物. 虽然矿化率和程度都很低,但P. chrysosporium在较长培养过程中可以将HPAM分子链上85%的放射性碳纳入其生物量. 由于14C标记位置位于HPAM的主链而不是侧链羰基碳,因此可靠地推测出该真菌可代谢HPAM的碳主链,即HPAM的分子链上碳碳键的降解. Stahl等[17]采用同样方法研究土壤微生物和P. chrysosporium对不溶性聚丙烯酸(PAA)和HPAM的降解,发现单独的土壤微生物只能很差地降解聚合物,并不能使它们矿化. 但土壤微生物与白腐真菌享有协同作用,即首先由P. chrysosporium分泌的过氧化物酶使聚合物溶解,其次土壤细菌将溶解的聚合物矿化. 因此,通过真菌和土壤细菌的共培养在聚合物增溶和矿化过程中HPAM和PAA的放射性碳分别回收了约83%和94%.
MnP首次发现于P. chrysosporium,MnP是含有亚铁血红素(Heme)的糖蛋白,Mn2+为辅助因子[18]. MnP以H2O2为电子受体,直接催化氧化降解如酚类、胺类、醛类及芳香类等富含电子的有机污染物. MnP在P. chrysosporium基因组中的位置已被识别,而且该酶的生化特性也被深入研究[19 − 20]. 在限氮的条件下,MnP对HPAM具有一定降解作用[21]. MnP通过加入H2O2将Mn2+催化生成Mn3+,并在原位形成Mn(Ⅲ)中间体而产生较高还原电位,因此具有氧化和解聚HPAM的能力[22]. 但仅通过实验无法了解MnP与HPAM的微观相互作用,而该信息对设计酶或理解酶解机理至关重要. MnP的三维晶体结构已通过实验解析出来[23 − 24],这为进一步在分子水平上研究该酶与底物相互作用提供了可能性. 目前对HPAM的降解过程研究大多集中于细菌,真菌较少,而且具体降解途径仍不清楚. 此外,经实验室筛选真菌分泌的酶活性通常较低. 分子对接是寻找酶与底物最优结合构象的有效方法,而MD模拟可探索酶与底物结合的稳定性. 因此本文采用分子对接和MD来研究和比较了MnP与HPAM结构模型相互作用,据此深入了解MnP与HPAM结合的分子机制,这将有助于理解HPAM酶降解反应,并为合理设计更高活性MnP提供理论参考.
-
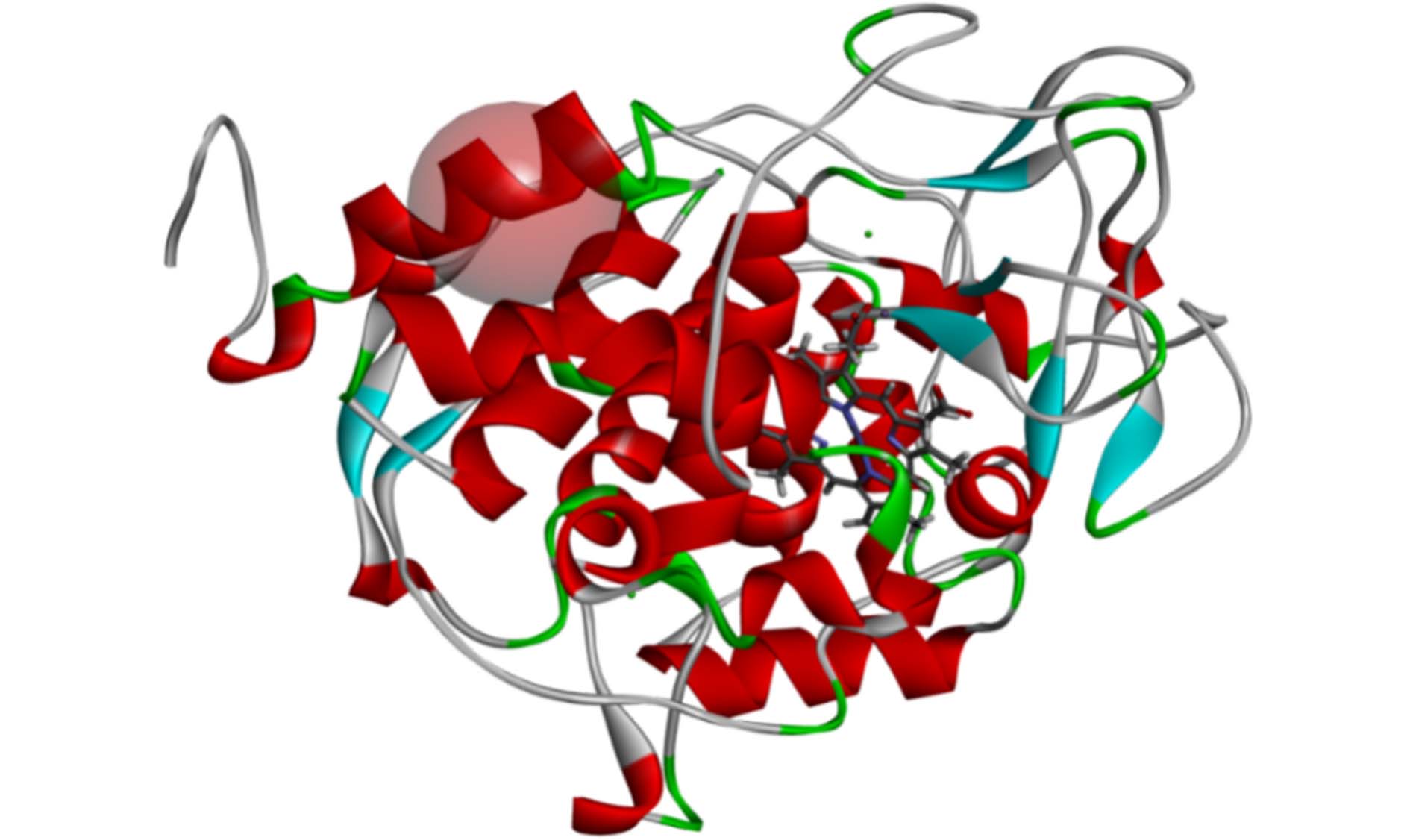
MnP的三维晶体结构下载于蛋白质数据库(http://www.rcsb.org/pdb/home/home.do),相应的PDB编号为3M5Q,分辨率为0.170 nm,A链. 该酶来源:P. chrysosporium. 采用Discovery Studio 2020的Prepare Protein工具对其进行预处理,去水加氢、去除酶结合的配体及修补缺失的氨基酸残基[25]. 图1显示预处理的MnP,其中含有2个Ca2+、1个Mn2+和Heme. 红色透明球体代表该酶进行对接的活性位点(x, 56.3; y,
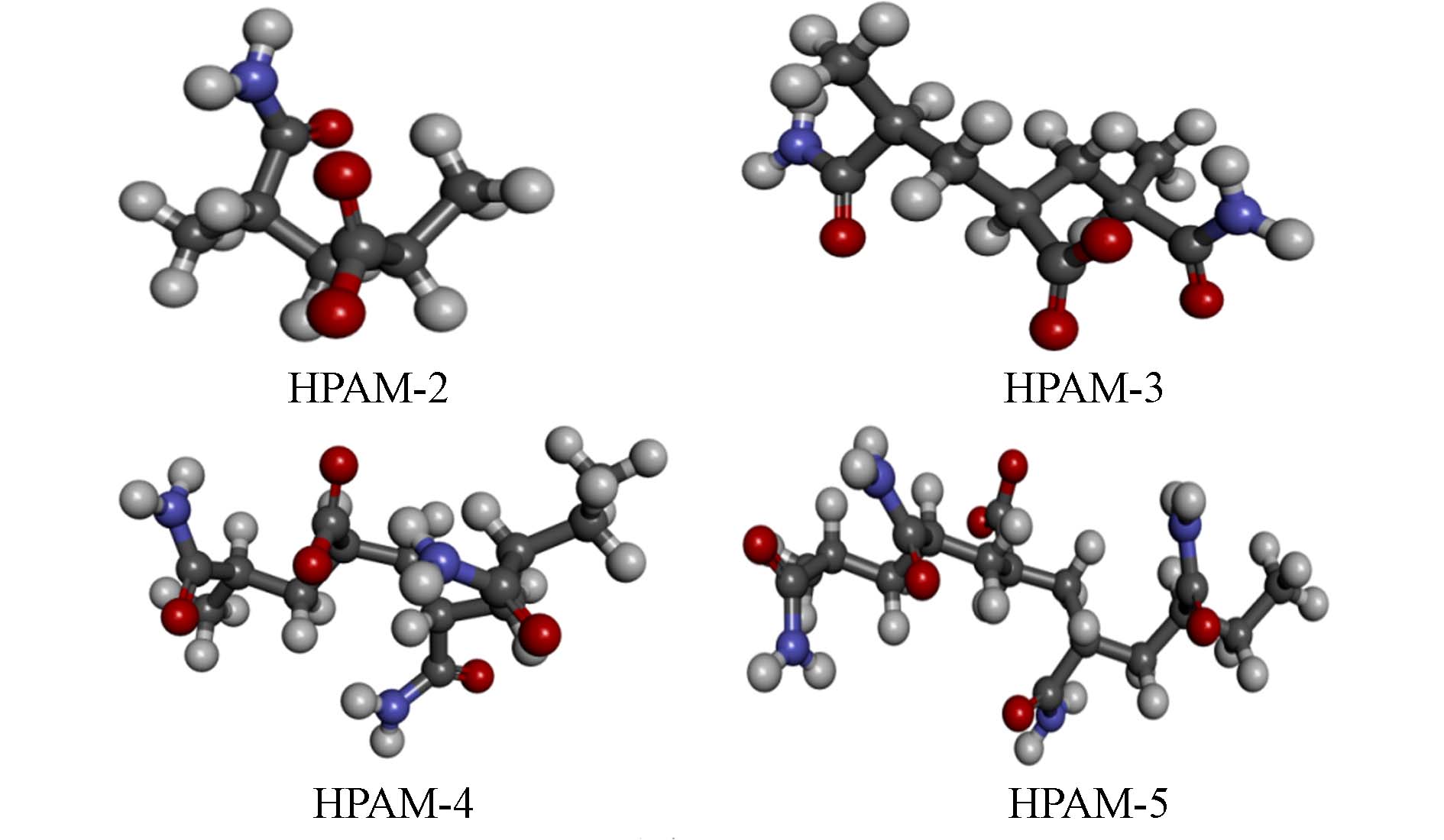
46.0903 ; z,47.0339 ; Radius, 5). HPAM球棍模型采用Materials Studio 2017软件的Build polymer工具绘制,分子链两端采用甲基进行封端[26]. 最后,采用CHARMm力场对其进行能量最小化,如图2所示. -
黄孢原毛平革菌属担子菌纲,好氧型嗜热真菌[27]. 实验所用黄孢原毛平革菌(保藏号:GIM 3.393)购买于广东省微生物菌种保藏中心,在无菌情况下,将上述菌株划线于固体基础培养基. 30 ℃培养5 d,连续活化两代后密封保存于4 ℃冰箱备用.
培养基:(1)基础培养基(L−1):马铃薯淀粉20 g、葡萄糖20 g、KH2PO4 3.0 g、MgSO4∙7H2O 1.5 g、维生素B1 8 mg、pH =6. 固体基础培养基是添加琼脂20 g于基础培养基制成;(2)降解培养基(L−1):葡萄糖0或2.0 g、酒石酸铵0.2 g、HPAM 100 mg、MgSO4 1.0 g、KH2PO4 3.0 g、维生素B1 8 mg、微量元素溶液10 mL、pH=6. 微量元素溶液(L−1):CuSO4∙5H2O 0.8 g、MnCl2∙4H2O 1.0 g、ZnSO4∙7H2O 1.2 g、Fe2(SO4)3 0.03 g. 上述所有培养基通过添加NaHCO3调节pH,且皆在121 ℃下高压蒸汽灭菌30 min后使用.
-
接种环挑取少量的P. chrysosporium菌株接种于固体基础培养基,在30 ℃培养5 d,然后向其加入10 mL无菌生理盐水,最后由接种环刮下一定量的孢子放置在盛有玻璃珠的锥形瓶,于30 ℃、200 r·min−1振荡培养10 d,每天测量菌丝的干重来计算真菌的生物量.
首先将0.5 mL的菌液接种到灭菌的含有50 mL基础培养基的150 mL锥形瓶中,在温度为30 ℃振荡富集培养2 d,然后将富集培养的0.5 mL菌液接种到含有50 mL基础培养基的150 mL锥形瓶中,在30 ℃振荡培养2 d,之后每隔3 d吸取2 mL菌液接种于一定HPAM浓度梯度的降解培养基进行驯化,其中初始浓度为100 mg·L−1,间隔为300 mg·L−1,最终浓度为
1200 mg·L−1,最后配置孢子菌悬液用于降解实验. -
取适量的降解液经6 000 r·min−1高速离心10 min后,将上清液采用淀粉-碘化镉法测定HPAM的浓度. 用UV-
5100 紫外可见分光光度计在波长585 nm处测定吸光度(A),每次试验平行进行3次,取平均值. 用A来间接表征HPAM的浓度,从而通过浓度与A之间的标准曲线得到HPAM的浓度. 计算HPAM降解率的表达式为:式中,C0为降解前HPAM的浓度,mg·L−1;Ct为降解后HPAM的浓度,mg·L−1;η为降解率.
-
将1 mL P. chrysosporium的孢子悬液转移到50 mL降解培养基中,在30 ℃下培养10 d,以不接种菌株作为空白对照. 在HPAM降解液中检测MnP的活性,以每分钟将1 μmol Mn2+转化为Mn3+的酶量作为MnP的一个酶活单位(U).
-
选用DS 2020的CDOCKER模块进行MnP与HPAM结构模型(HPAM-2、HPAM-3、HPAM-4和HPAM-5)的对接[28]. 该程序采用半柔性对接,首先,高温动力学产生配体构象集,然后,采用模拟退火对配体构象集依次在受体的活性位点进行结构优化. 默认参数设置是保存最佳的前10种配体构象,选择-CDOCKER_ Energy score打分最高的配体构象进行分析.
-
采用GROMACS软件包(版本
2020.6 )对MnP-HPAM-2、MnP-HPAM-3、MnP-HPAM-4和MnP-HPAM-5等复合物在298.15 K下的MD模拟[29],MnP和底物的力场分别选为AMBER14SB force field和general AMBER force field. 为使模拟过程更接近真实情况,体系被溶剂化在拥有1.2 nm溶剂化层的TIP3P水模型[30]周期性边界条件的立方盒子里,添加反离子Na+和Cl-. 首先,采用最速下降法和共轭梯度法进行能量最小化,并且在298.15 K下进行100 ps NPT MD使体系达到平衡. 温度和压力由Berendsen耦合算法控制[31],时间常数分别为0.1 ps和1.0 ps. 最后,模拟体系在298.15 K下进行了NPT 100 ns采样,采用Parrinello-Rahman耦合算法控制压力[32]. MD模拟过程中范德华截断半径为1.2 nm,长程静电相互作用采用PME进行处理[33],步长设置为2.0 fs. 以上MD模拟计算是由北京中大唯信科技有限公司提供的平台下完成. -
采用Visual Molecular Dynamics (VMD) 软件(版本1.9.4)对复合物的构象变化进行可视化分析及比较[34],同时对酶骨架的均方根偏差(RMSD)、酶残基的均方根涨落(RMSF)和回转半径(Rg)等动力学性质进行了分析. RMSD高度反映了轨迹相对于酶初始构象整体偏离程度,当曲线在某个高度附近平稳振动时,表明体系达到平衡,振幅反映了其振动剧烈程度.
其中,N为原子数;mi为原子i的质量,Xi t为酶骨架原子i在t时刻的矢量坐标,Yi0为酶骨架原子i在0时刻的矢量坐标,M=总质量.
RMSF计算残基相对于其平均位置的涨落,表征残基的柔性.
其中,t为时间,
$ {x}_{i}\left({t}_{j}\right) $ 为t时刻原子i的位置,$ \overline{{x}_{i}} $ 为原子i的平均位置.Rg描述体系原子沿着特定轴向分布特征,用于表征分子的紧密程度.
其中,ri—rc为原子i到全部原子质心的距离. 采用MM/PBSA方法计算结合自由能(ΔGbinding),即基于来自AmberTools20套件MMPBSA.py[35]的gmx_MMPBSA (版本1.4.3)软件[36]来计算ΔGbinding,最后10 ns平衡轨迹中每间隔100 ps保存1次.
-
本研究中选择HPAM的球棍模型用于研究MnP降解HPAM的机理. 目前还没有从分子水平上揭示MnP与HPAM之间的相互作用. CDOCKER程序通常采用两种能量打分来评价结果,即 -CDOCKER_ Energy score 是酶-底物复合物总能量打分,该值越大越说明酶同底物亲和力越大、越稳定;-CDOCKER_ Interaction_ Energy score是酶与底物相互作用能量打分,该值越大越说明酶与底物结合越好[37]. 从表1的对接结果中的打分值可以看出,无论基于 -CDOCKER_ Energy score还是-CDOCKER_ Interaction_ Energy score,MnP对HPAM的打分皆随其碳链的延长而逐渐增加,说明MnP倾向氧化降解HPAM碳链,对其碳链长度有一定耐受性. 同时发现MnP-HPAM-5拥有最高打分,说明MnP对HPAM-5拥有最高结合亲和强度[38]. 结合自由能是按酶与底物结合前后两个体系真实的能量差值计算来的,该值能可靠评估复合物的稳定性. MnP-HPAM-5拥有最高的ΔGbinding,进一步说明该复合物最稳定.
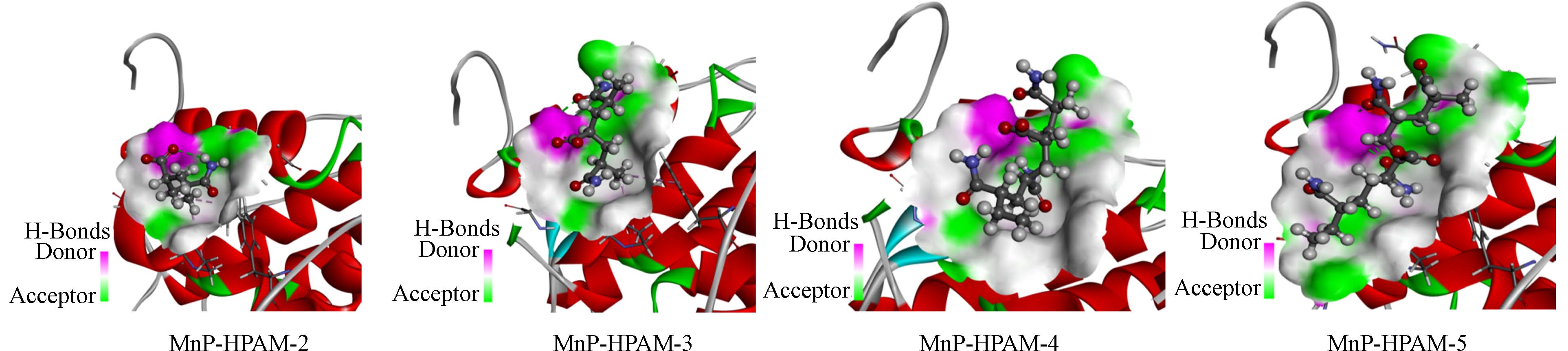
图3显示了HPAM与MnP最佳结合构象,受体表面按氢键性质显示. 从图3可以发现,HPAM-5的整个分子完全伸展,平铺在MnP受体表面上,这能很好解释MnP-HPAM-5有最高能量打分. 同时也观察到,HPAM随聚合度的增加而并没有向受体表面外偏移,说明MnP可能会降解一定分子量的HPAM.
-
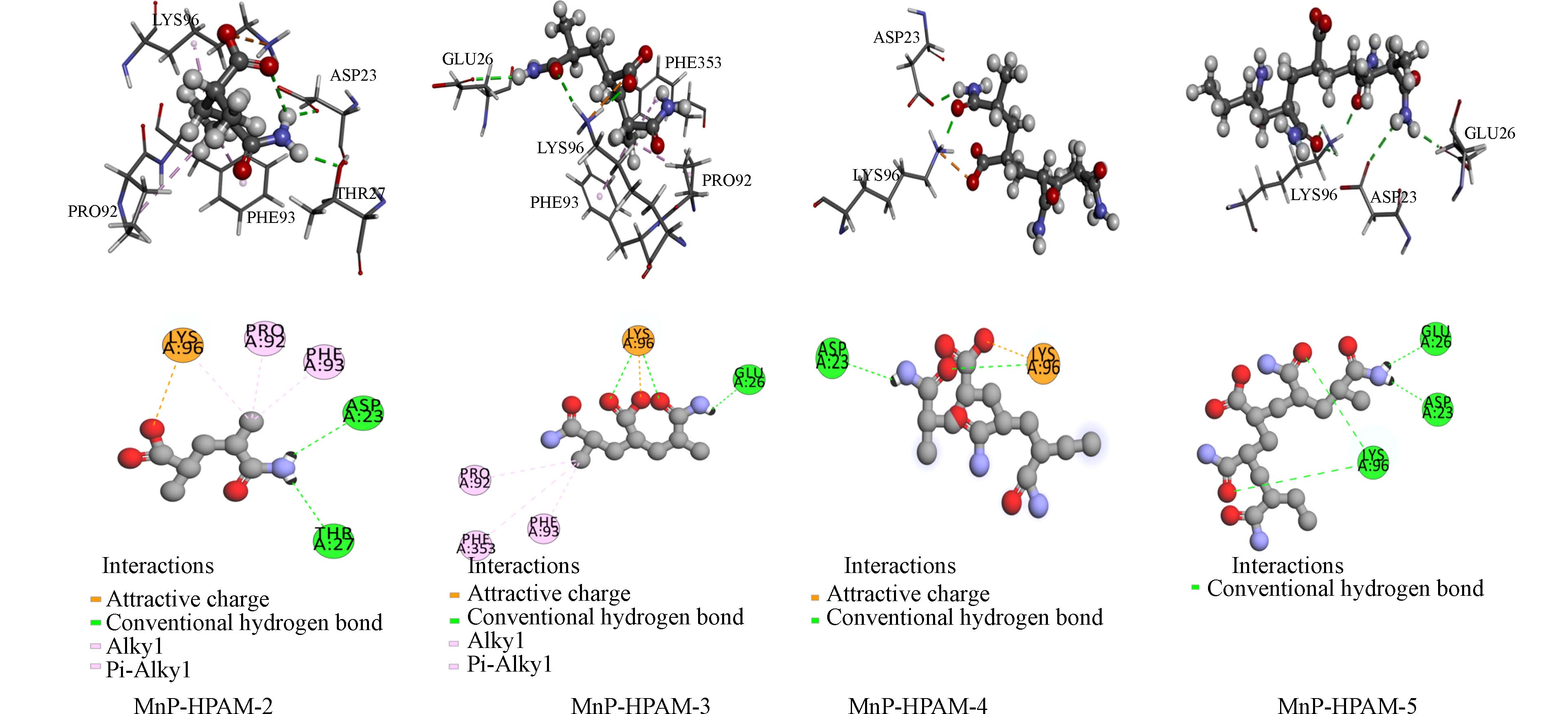
单从亲和力和对接打分很难确定酶的催化活性,相互作用分析必须考虑[39]. 在配体与蛋白结合过程中,配体周围的关键氨基酸残基对于降解底物起主要作用. 通过DS 2020对MnP与HPAM复合物进行可视化,其相互作用关系如图4.
从图4可以看出,HPAM-2的分子链一端与PHE93、PRO92和LYS96形成3个疏水键,其中LYS96的氮正离子与HPAM-2的氧负离子存在1个静电吸引. HPAM-2与ASP23和THR27形成2个氢键. HPAM-2分子内形成1个氢键,底物HPAM-2较稳定,这可能会阻碍其与MnP结合. HPAM-3分子链的一端与PHE93、PRO92和PHE353形成3个疏水键,HPAM-2的氧负离子与LYS96存在1个静电吸引,同时LYS96与HPAM-3形成2个氢键,HPAM-3与GLU26形成1个氢键. 根据上述分析,可以推测HPAM-2的分子内氢键是MnP-HPAM-3比MnP-HPAM-2更稳定的主要原因. HPAM-4与LYS96和ASP23形成2个氢键,而且LYS96与HPAM-4的氧负离子形成铵盐,说明盐桥作用可能对稳定MnP与底物起主要作用. 因此可以推测出盐桥是MnP-HPAM-4比MnP-HPAM-2和MnP-HPAM-3更稳定的主要原因. HPAM-5与GLU26和ASP23形成2个氢键,HPAM-5与LYS96形成3个氢键. 通过分析可以得到HPAM-5与MnP结合时一共形成5个氢键,形成的氢键最多,说明MnP-HPAM-5的亲和力和稳定性都最大是由于强的氢键作用.
-
在MD模拟过程中通常采用RMSD来衡量酶结构的稳定性和稳健性,因此计算了每个复合物骨架原子的RMSD,结果如图5(a)所示. 除MnP-HPAM-3外,所有分析的复合物的RMSD值在40 ns达到平衡. MnP-HPAM-3的初始RMSD值在40 ns时逐渐上升,从55 ns到65 ns达到最大值~0.38 nm,然后其缓慢下降,最终达到新的平衡. 这说明MnP在与HPAM-3结合时酶的骨架结构发生较大的振动,它们的结合稳定性是最差的. 同时,发现MnP-HPAM-4的RMSD变化范围最小,说明MnP与HPAM-4结合时酶的稳定性最佳.
RMSF被计算来分析MnP-HPAM模型化合物复合物中每个残基的动态变化,如图5(b)所示. 最大的RMSF值代表最高的残基波动,此时残基对酶结合底物的稳定性有不利影响. 所有分析复合物在350—360的残基区域(矩形黑色方框)波动最大,而MnP-HPAM-4的RMSF变化在该区域保持最小波动,这进一步说明MnP-HPAM-4的结合稳定性是最佳的.
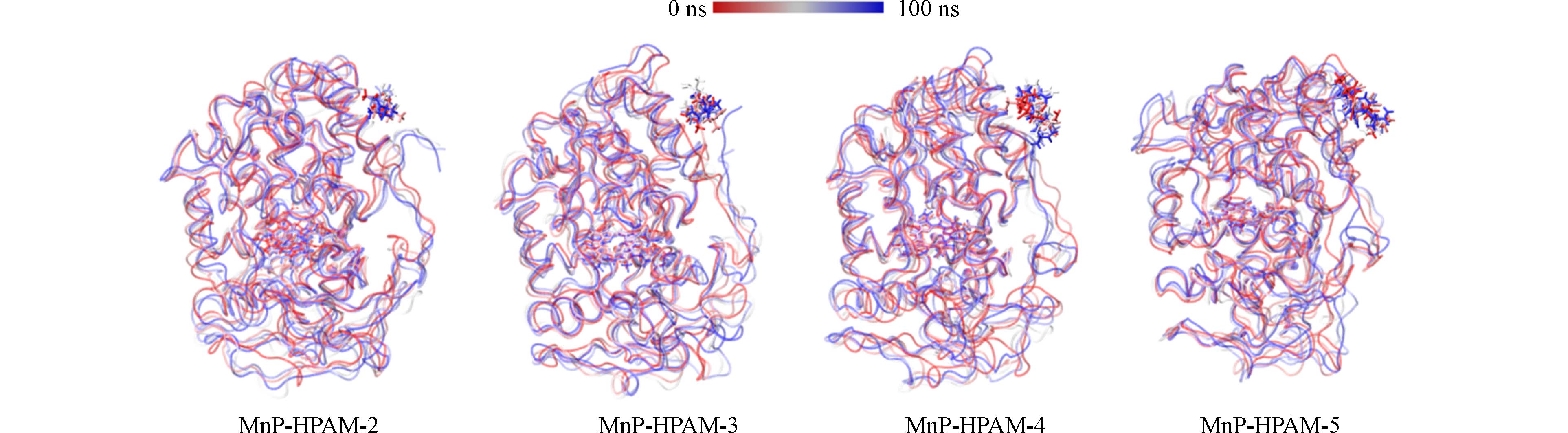
Rg反映了复合物的压实结构[40],该值波动范围越小,就越说明此时复合物的结构最稳定. 从图5(c)可以看出,MnP-HPAM-4的Rg波动最不明显,说明该复合物结构最稳定. 图6显示了MnP与底物的动力学运动轨迹. 可以发现所有分析的复合物中底物一直在MnP的活性位点附近发生振动,而酶的骨架运动较小,说明由CDOCKER程序确定MnP的结合位点是正确的.
-
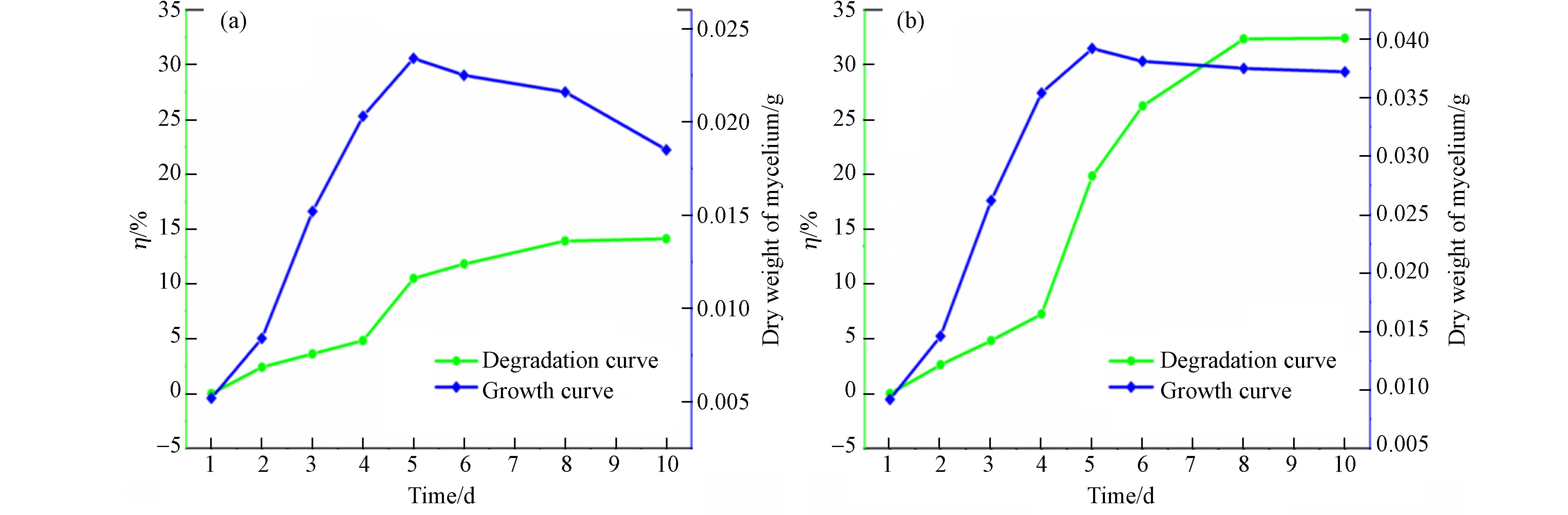
为了验证HPAM降解过程中MnP起到主导作用,采用分泌该酶的P. chrysosporium进行降解实验. 从图7(a)可以看出,在不添加葡萄糖的降解培养基中真菌生长曲线有一个下降阶段,此时的HPAM降解率较低(14.12%). 从图7(b)可以看出,真菌生长在一定时间内(0—5 d)呈增长趋势,5 d后趋于稳定,而HPAM降解率在其生长稳定期时达到最大值(32.42%). 以上结果表明葡萄糖是一种容易接近的碳源,低浓度的葡萄糖作为共基质可以促进真菌生长和HPAM的降解. 同时在降解培养基中检测到MnP,其最高活性为12.09 U·mL−1. 然而,MnP如何与底物相互作用?而且目前仅通过传统驯化获得P. chrysosporium的HPAM降解效果并不理想. 因此有必要进行分子模拟结合实验来帮助设计高活性酶突变体以及理解酶解机理.
-
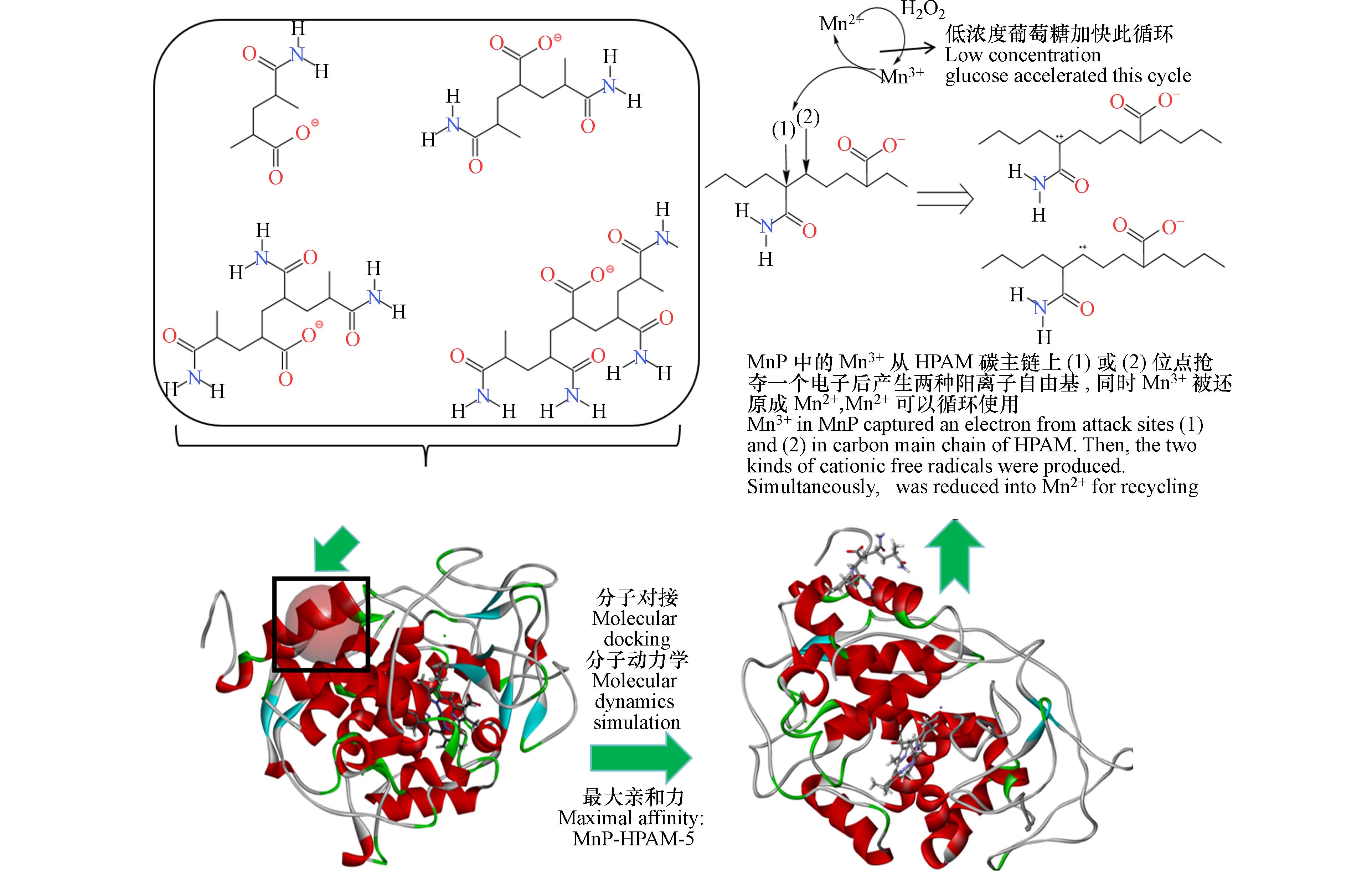
如图8所示,H2O2引发MnP中Mn2+氧化为Mn3+,而Mn3+具有很强的吸电子能力.
Mn3+能从HPAM的(1)和(2)位点上吸收一个电子而引发自由基链反应,分别产生了2种阳离子自由基而不破坏化学键,此时Mn3+被还原为Mn2+, Mn2+可循环利用. 阳离子自由基的生成是控制聚合物氧化反应速率的关键步骤. 值得注意的是,添加额外的葡萄糖可以刺激真菌生长而分泌更高活性的MnP,加快Mn3+的产生速度,最终加快锰离子的循环,产生更多的活性阳离子自由基来促进HPAM降解成小的聚合物片段.
-
采用分子对接和MD基于HPAM模型化合物比较了HPAM与MnP相互作用,而酶与底物的动态和结构信息将有助于研制新型高活性及热稳定性的MnP以降解HPAM. 然而本文仅通过实验证明MnP参与降解过程,未来需要进行大量酶降解动力学及点突变等实验来探究酶的活性和稳定性.
(1)通过结合亲和力分析表明,MnP与HPAM的亲和力随HPAM碳链的延长而逐渐增加,尤其该酶对HPAM-5拥有最高的亲和力,即HPAM-5是MnP的最适底物.
(2)通过相互作用分析可以发现,HPAM-2分子内氢键导致MnP-HPAM-2的稳定性比MnP-HPAM-3更差. HPAM-4的氧负离子与氨基酸残基LYS96形成铵盐,说明盐桥导致MnP-HPAM-4的稳定性比MnP-HPAM-2和MnP-HPAM-3都更好. MnP-HPAM-5形成的氢键最多,说明强的氢键导致MnP-HPAM-5的亲和力和稳定性都最大.
(3)通过MD模拟结果分析表明,MnP-HPAM-4享有最小的RMSD、RMSF和Rg,该复合物结合是最稳定的,同时进一步确认了对接时选取酶活性位点的位置是合理的.
(4)根据模拟以及降解实验结果提出了MnP降解HPAM机理:H2O2引发MnP中Mn2+氧化为Mn3+,而Mn3+具有很强吸电子能力;Mn3+从HPAM的(1)和(2)位点上吸到一个电子而引发自由基链反应,此时产生了2种阳离子自由基,但不破坏化学键;同时Mn3+还原成Mn2+, Mn2+可循环使用.
锰过氧化物酶与阴离子型聚丙烯酰胺计算机分子模拟
Computer molecular simulation between manganese peroxidase and anionic polyacrylamide
-
摘要: 为探究锰过氧化物酶(MnP)与阴离子型聚丙烯酰胺(HPAM)的微观相互作用,采用分子对接和分子动力学(MD)模拟研究阴离子型聚丙烯酰胺(HPAM)与MnP的结合,根据-CDOCKER Energy_ score打分最高的原则,对获得最佳结合构象进行分析. 结合亲和力分析表明,MnP对HPAM-5有最高的亲和力,而且MnP对HPAM的亲和力随其碳链的延长逐渐升高. 相互作用分析表明,MnP-HPAM-2的稳定性比MnP-HPAM-3更差是由于HPAM-2分子内氢键;MnP-HPAM-4比MnP-HPAM-2和MnP-HPAM-3更稳定是由于盐桥;强氢键是MnP-HPAM-5最稳定的主要原因. MD模拟结果分析表明,MnP-HPAM-4享有最小的RMSD、RMSF和Rg,此时复合物结合是最稳定的. 根据模拟及降解实验结果提出了MnP降解HPAM机理:H2O2引发MnP中Mn2+氧化为Mn3+; Mn3+从HPAM(1)和(2)攻击位点上抢夺一个电子而引发自由基链反应,在不破坏化学键下产生2种阳离子自由基,而且Mn3+还原成Mn2+供循环使用.
-
关键词:
- 锰过氧化物酶(MnP) /
- 阴离子型聚丙烯酰胺(HPAM) /
- 分子对接 /
- 分子动力学(MD)模拟.
Abstract: In order to explore the microscopic interaction between anionic polyacrylamide (HPAM) and manganese peroxidase (MnP), molecular docking and molecular dynamics (MD) simulations were performed to investigate the binding of anionic polyacrylamide (HPAM) with MnP. The optimal conformation was obtained according to the highest principle of -CDOCKER Energy_ score and analyzed. Analysis of binding affinity indicated that MnP-HPAM-5 acquired the highest affinity. Moreover, the affinity of MnP with HPAM was gradually risen with the extension of its carbon chain. The interaction analysis showed that the stability of MnP-HPAM-2 was worse than MnP-HPAM-3 due to intramolecular hydrogen bond of HPAM-2; MnP-HPAM-4 was more stable than MnP-HPAM-2 and MnP-HPAM-3 due to salt bridge; The strong hydrogen bond was the main reason why MnP-HPAM-5 was most stable. Results of MD simulations showed that MnP-HPAM-4 enjoyed the smallest RMSD, RMSF and Rg, and its binding was the most stable. Based on the results of simulation and degradation tests, the mechanism of MnP degrading HPAM was proposed: 1) The Mn2+ in MnP was oxidated into Mn3+ by H2O2 initiation; 2) Then, Mn3+ captured an electron from attack sites(1)and(2)in HPAM and triggered a radical chain reaction, producing two kinds of cationic free radicals without breaking the chemical bond; 3) Mn3+ was reduced into Mn2+ for recycling. -

-
表 1 所有复合物的CDOCKER能量打分和ΔGbinding
Table 1. The CDOCKER energy score and ΔGbinding for all complexes
酶与底物复合物
Enzyme-substrate complex负的总能量打分/
(kcal·mol−1)
-CDOCKER_ Energy score负的相互作用能量打分/(kcal·mol−1)
-CDOCKER_Interaction_
Energy score结合自由能/
(kcal·mol−1)
ΔGbindingMnP-HPAM-2 27.6711 26.3182 −9.58 MnP-HPAM-3 31.0265 31.4252 −10.05 MnP-HPAM-4 33.9551 32.5108 −10.18 MnP-HPAM-5 36.3184 34.6535 −11.71 -
[1] REN B, MIN F F, CHEN J, et al. Adsorption mechanism insights into CPAM structural units on kaolinite surfaces: A DFT simulation[J]. Applied Clay Science, 2020, 197: 105719. doi: 10.1016/j.clay.2020.105719 [2] MORTIMER D A. Synthetic polyelectrolytes—a review[J]. Polymer International, 1991, 25(1): 29-41. doi: 10.1002/pi.4990250107 [3] MARK H. Encyclopedia of polymer science and technology, 15 volume set[M]. New York, NY, USA: Wiley, 2014, 14: 519-520. [4] CAULFIELD M J, QIAO G G, SOLOMON D H. Some aspects of the properties and degradation of polyacrylamides[J]. Chemical Reviews, 2002, 102(9): 3067-3084. doi: 10.1021/cr010439p [5] 邹文杰, 曹亦俊, 李维娜, 等. 煤及高岭石的选择性絮凝[J]. 煤炭学报, 2013, 38(8): 1448-1453. ZOU W J, CAO Y J, LI W N, et al. Selective flocculation of coal and kaolinite[J]. Journal of China Coal Society, 2013, 38(8): 1448-1453 (in Chinese).
[6] 常自勇, 冯其明, 欧乐明. 回水中絮凝剂对铝土矿浮选影响的研究[J]. 有色金属(选矿部分), 2014(6): 88-91. CHANG Z Y, FENG Q M, OU L M. Study on the impact of flocculants in backwater on bauxite flotation[J]. Nonferrous Metals (Mineral Processing Section), 2014(6): 88-91 (in Chinese).
[7] HU H, LIU J F, LI C Y, et al. Anaerobic biodegradation of partially hydrolyzed polyacrylamide in long-term methanogenic enrichment cultures from production water of oil reservoirs[J]. Biodegradation, 2018, 29(3): 233-243. doi: 10.1007/s10532-018-9825-1 [8] ZHAO L M, BAO M T, YAN M, et al. Kinetics and thermodynamics of biodegradation of hydrolyzed polyacrylamide under anaerobic and aerobic conditions[J]. Bioresource Technology, 2016, 216: 95-104. doi: 10.1016/j.biortech.2016.05.054 [9] BAO M T, CHEN Q G, LI Y M, et al. Biodegradation of partially hydrolyzed polyacrylamide by bacteria isolated from production water after polymer flooding in an oil field[J]. Journal of Hazardous Materials, 2010, 184(1/2/3): 105-110. [10] WEN Q X, CHEN Z Q, ZHAO Y, et al. Biodegradation of polyacrylamide by bacteria isolated from activated sludge and oil-contaminated soil[J]. Journal of Hazardous Materials, 2010, 175(1/2/3): 955-959. [11] 林玉珍, 曾光明, 张娱, 等. 有机农药滴滴涕和毒死蜱生物降解机制的分子模拟研究[J]. 环境科学, 2012, 33(3): 1015-1019. LIN Y Z, ZENG G M, ZHANG Y, et al. Biodegradation mechanism of DDT and chlorpyrifos using molecular simulation[J]. Environmental Science, 2012, 33(3): 1015-1019 (in Chinese).
[12] CHEN M, ZENG G M, LAI C, et al. Molecular basis of laccase bound to lignin: Insight from comparative studies on the interaction of Trametes versicolor laccase with various lignin model compounds[J]. RSC Advances, 2015, 5(65): 52307-52313. doi: 10.1039/C5RA07916K [13] LIU Z F, SHAO B B, ZENG G M, et al. Effects of rhamnolipids on the removal of 2, 4, 2, 4-tetrabrominated biphenyl ether (BDE-47) by Phanerochaete chrysosporium analyzed with a combined approach of experiments and molecular docking[J]. Chemosphere, 2018, 210: 922-930. doi: 10.1016/j.chemosphere.2018.07.114 [14] LIU Y J, LIU Z F, ZENG G M, et al. Effect of surfactants on the interaction of phenol with laccase: Molecular docking and molecular dynamics simulation studies[J]. Journal of Hazardous Materials, 2018, 357: 10-18. doi: 10.1016/j.jhazmat.2018.05.042 [15] KÜES U. Fungal enzymes for environmental management[J]. Current Opinion in Biotechnology, 2015, 33: 268-278. doi: 10.1016/j.copbio.2015.03.006 [16] SUTHERLAND G R, HASELBACH J, AUST S D. Biodegradation of crosslinked acrylic polymers by a white-rot fungus[J]. Environmental Science and Pollution Research International, 1997, 4(1): 16-20. doi: 10.1007/BF02986258 [17] STAHL J D, CAMERON M D, HASELBACH J, et al. Biodegradation of superabsorbent polymers in soil[J]. Environmental Science and Pollution Research International, 2000, 7(2): 83-88. doi: 10.1065/espr199912.014 [18] CHEN M, ZENG G M, TAN Z Y, et al. Understanding lignin-degrading reactions of ligninolytic enzymes: Binding affinity and interactional profile[J]. PLoS One, 2011, 6(9): e25647. doi: 10.1371/journal.pone.0025647 [19] PHIL K, DAN C. Extracellular oxidative systems of the lignin-degrading Basidiomycete Phanerochaete chrysosporium[J]. Fungal Genetics and Biology: FG & B, 2007, 44(2): 77-87. [20] MARTINEZ D, LARRONDO L F, PUTNAM N, et al. Genome sequence of the lignocellulose degrading fungus Phanerochaete chrysosporium strain RP78[J]. Nature Biotechnology, 2004, 22(6): 695-700. doi: 10.1038/nbt967 [21] 韩昌福, 郑爱芳, 李大平. 聚丙烯酰胺生物降解研究[J]. 环境科学, 2006, 27(1): 151-153. HAN C F, ZHENG A F, LI D P. Study on biodegradation of polyacrylamide[J]. Environmental Science, 2006, 27(1): 151-153 (in Chinese).
[22] DING Y, CUI K P, GUO Z, et al. Manganese peroxidase mediated oxidation of sulfamethoxazole: Integrating the computational analysis to reveal the reaction kinetics, mechanistic insights, and oxidation pathway[J]. Journal of Hazardous Materials, 2021, 415: 125719. doi: 10.1016/j.jhazmat.2021.125719 [23] SUNDARAMOORTHY M, KISHI K, GOLD M H, et al. The crystal structure of manganese peroxidase from Phanerochaete chrysosporium at 2.06-a resolution[J]. Journal of Biological Chemistry, 1994, 269(52): 32759-32767. doi: 10.1016/S0021-9258(20)30056-9 [24] SUNDARAMOORTHY M, GOLD M H, POULOS T L. Ultrahigh (0.93A) resolution structure of manganese peroxidase from Phanerochaete chrysosporium: Implications for the catalytic mechanism[J]. Journal of Inorganic Biochemistry, 2010, 104(6): 683-690. doi: 10.1016/j.jinorgbio.2010.02.011 [25] TU M L, WANG C, CHEN C, et al. Identification of a novel ACE-inhibitory peptide from casein and evaluation of the inhibitory mechanisms[J]. Food Chemistry, 2018, 256: 98-104. doi: 10.1016/j.foodchem.2018.02.107 [26] ZHAO X D, SONG L Z, FU J, et al. Experimental and DFT investigation of surface degradation of polyvinylidene fluoride membrane in alkaline solution[J]. Surface Science, 2011, 605(11/12): 1005-1015. [27] 张东晨, 刘志勇, 王涛, 等. 煤炭絮凝微生物黄孢原毛平革菌光谱及电镜研究[J]. 煤炭学报, 2010, 35(5): 825-829. ZHANG D C, LIU Z Y, WANG T, et al. Experimental study on ultraviolet spectrum and FTIR and SEM of coal bio-flocculant phanerochaete chrysosporium[J]. Journal of China Coal Society, 2010, 35(5): 825-829 (in Chinese).
[28] WANG X K, JI G X, HAN X Y, et al. Thiazolidinedione derivatives as novel GPR120 agonists for the treatment of type 2 diabetes[J]. RSC Advances, 2022, 12(10): 5732-5742. doi: 10.1039/D1RA08925K [29] ABRAHAM M J, MURTOLA T, SCHULZ R, et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J]. SoftwareX, 2015, 1/2: 19-25. doi: 10.1016/j.softx.2015.06.001 [30] JORGENSEN W L, CHANDRASEKHAR J, MADURA J D, et al. Comparison of simple potential functions for simulating liquid water[J]. The Journal of Chemical Physics, 1983, 79(2): 926-935. doi: 10.1063/1.445869 [31] BERENDSEN H J C, POSTMA J P M, van GUNSTEREN W F, et al. Molecular dynamics with coupling to an external bath[J]. The Journal of Chemical Physics, 1984, 81(8): 3684-3690. doi: 10.1063/1.448118 [32] PARRINELLO M, RAHMAN A. Polymorphic transitions in single crystals: A new molecular dynamics method[J]. Journal of Applied Physics, 1981, 52(12): 7182-7190. doi: 10.1063/1.328693 [33] ESSMANN U, PERERA L, BERKOWITZ M L, et al. A smooth particle mesh Ewald method[J]. The Journal of Chemical Physics, 1995, 103(19): 8577-8593. doi: 10.1063/1.470117 [34] HUMPHREY W, DALKE A, SCHULTEN K. VMD: Visual molecular dynamics[J]. Journal of Molecular Graphics, 1996, 14(1): 33-38. doi: 10.1016/0263-7855(96)00018-5 [35] MILLER B R, McGEE T D, SWAILS J M, et al. MMPBSA. py: An efficient program for end-state free energy calculations[J]. Journal of Chemical Theory and Computation, 2012, 8(9): 3314-3321. doi: 10.1021/ct300418h [36] VALDÉS-TRESANCO M S, VALDÉS-TRESANCO M E, VALIENTE P A, et al. gmx_MMPBSA: A new tool to perform end-state free energy calculations with GROMACS[J]. Journal of Chemical Theory and Computation, 2021, 17(10): 6281-6291. doi: 10.1021/acs.jctc.1c00645 [37] 王方略, 张东晨, 吴学凤, 等. 红球菌酰胺酶降解阴离子型聚丙烯酰胺的亲和力分析[J]. 环境化学, 2023, 42(1): 319-326. doi: 10.7524/j.issn.0254-6108.2021083103 WANG F L, ZHANG D C, WU X F, et al. Affinity analysis of anionic polyacrylamide degraded by amidase from Rhodococcus sp. N-771[J]. Environmental Chemistry, 2023, 42(1): 319-326 (in Chinese). doi: 10.7524/j.issn.0254-6108.2021083103
[38] TU M L, LIU H X, ZHANG R Y, et al. Analysis and evaluation of the inhibitory mechanism of a novel angiotensin-I-converting enzyme inhibitory peptide derived from casein hydrolysate[J]. Journal of Agricultural and Food Chemistry, 2018, 66(16): 4139-4144. doi: 10.1021/acs.jafc.8b00732 [39] CASSIDY C E, SETZER W N. Cancer-relevant biochemical targets of cytotoxic Lonchocarpus flavonoids: A molecular docking analysis[J]. Journal of Molecular Modeling, 2010, 16(2): 311-326. doi: 10.1007/s00894-009-0547-5 [40] JALKUTE C B, BARAGE S H, SONAWANE K D. Insight into molecular interactions of Aβ peptide and gelatinase from Enterococcus faecalis: A molecular modeling approach[J]. RSC Advances, 2015, 5(14): 10488-10496. doi: 10.1039/C4RA09354B -



 下载:
下载: