-
微生物是土壤生态系统的重要组成部分. 自然条件下,微生物对土壤环境的长期适应使得土壤微生物群落组成相对稳定[1 − 2]. 在石油烃等污染物胁迫条件下,微生物群落结构和组成发生改变,一些特异性微生物生长为土壤中的优势菌群[3 − 4]. 例如,Gao等[5]的研究发现,石油胁迫条件下变形菌门(Proteobacteria)和放线菌门(Actinomycetes)成为污染土壤中的优势菌门,土壤中微生物群落的均匀度和多样性减低,表现出石油污染对土壤菌群落结构和组成的胁迫性. 但是,也有研究认为土壤中的石油烃可作为碳源被微生物利用,从而刺激土著微生物的生长,使得微生物多样性指数和活性均高于洁净土壤[6 − 7]. 蔡萍萍等[8]的研究发现油污土壤中的微生物群落结构经过长时间演变后与洁净土壤相似. 土著微生物在对石油烃产生应激响应的同时,对土壤中石油烃的降解起到促进作用[8 − 10]. Polyak等[10]发现,经过9年的自然降解土壤中总石油烃的去除率可以达到91.2%. 尽管目前已有大量文献报道了石油污染对土著菌群的影响作用以及石油烃在土壤中的自然降解过程,但是对于石油污染土壤中不同微生物类群随时间的变化特征、以及各微生物类群对石油烃利用的差异性尚不清楚.
磷脂脂肪酸(phospholipid fatty acids,PLFA)广泛存在于活体微生物的细胞膜中,当细胞凋亡后能在短时间内完全降解[11 − 12]. 由于不同微生物类群的PLFA组成和含量具有种属特异性,因此通过测定PLFA可以估算微生物群落结构活性变化[13 − 14]. 目前,稳定同位素(stable isotope probing,SIP)技术已经广泛应用于环境科学领域,在污染物溯源、识别转化历程和指示反应程度等过程中均发挥了重要作用 [15]. 将SIP技术和PLFA分析结合,可以揭示土壤微生物群落对污染物的利用特征[16 − 17]. 目前磷脂脂肪酸-稳定同位素联用(PLFA-SIP)技术广泛应用于研究农业土壤中微生物对植物残体、肥料等的利用情况[18 − 20],但较少用于研究土壤中微生物类群对污染物胁迫的应激响应.
该论文以陕北地区的洁净黄绵土为研究对象,以13C标记的十六烷作为石油中烷烃组分的模式化合物对土壤进行人工污染,通过培养试验,结合13C-PLFA-SIP技术研究土壤微生物组成变化及不同微生物类群对烷烃污染的响应情况,研究可为探明土壤微生物类群对石油烃污染响应的时效性及不同微生物类群对污染物的利用特征提供科学依据.
-
土壤样品取自陕北的洁净黄绵土. 采集表层土壤(0—20 cm),剔除杂质、风干研磨、过筛(100 目),充分混匀后装袋,储存于−80 ℃以备实验所用[21]. 土壤基本理化性质见表1.
供试烷烃为13C标记的正十六烷. 将
0.1000 g丰度为99 %atom的标记十六烷(Sigma)与1.8909 g丰度为1.108 %atom的分析纯十六烷(Sigma)充分混合,经计算后其13C丰度为5.984% atom. -
取6份质量为100 g的黄绵土,分成两组,每组3个平行. 将第一组在自然条件下放置作为控制实验(标记为“C”),向第二组土壤中加入0.5000 g 13C标记的十六烷进行人工污染(标记为“W”). 置于室温下培养30 d,在培养的第0、3、7、15、30 天采集各土壤样品,利用磷脂脂肪酸—稳定同位素技术分析土壤中的PLFA和13C-PLFA含量[16 − 18]. 取0、3、30 d土壤利用GC-MS测定十六烷含量.
-
土壤的理化性质测定参考《土壤农化分析》常规的分析方法 [22]. 土壤含水率采用烘干法测定,pH采用电极法测定,利用总有机碳测定仪(Vario TOC,Elementar)测定总有机碳,氨氮、硝氮采用紫外分光光度法测定,总氮和全硫含量采用元素分析仪(Vario MACRO,Elementar)测定. 十六烷含量采用超声萃取—GC-MS(7000B,Agilent Technologies)测定,具体操作详见文献[23 − 24]. 每个土壤样品进行3次平行测定.
-
PLFA的提取和测定参考文献 [14,20,25],具体测定方法如下:
称取3.0000 g冷冻干燥的土样加入到15 mL提取剂中(氯仿:甲醇:柠檬酸=1:2:0.8),室温振荡2 h后于
3000 r·min−1离心10 min,收集上清液. 向剩下土样中加入7 mL提取剂,重复上述步骤,合并两次提取的上清液,并加入柠檬酸缓冲液和氯仿,静置过夜,分层后用氮气吹干. 再用甲醇冲洗两次后用氮气吹干.采用12 mL硅胶SPE小柱(Supelco公司,KLJ-
57116 )分离磷脂、糖脂和中性脂. 用氯仿(3次,每次1.0 mL)转移脂类至SPE柱中. 分别用10 mL氯仿和10 mL丙酮依次洗去中性脂和糖脂,最后用10 mL甲醇洗脱磷脂并收集,用氮气吹干. 加入内标(十九脂肪酸甲酯,C19:0)200 μL,用氮气吹干.在含磷脂的试管中依次加入1 mL甲醇甲苯混合液(甲醇:甲苯=1:1)和KOH甲醇溶液(0.2 mol·L−1),于37 ℃的水浴锅中保温15 min,加入2 mL超纯水和0.3 mL醋酸溶液,再用正己烷振荡摇匀提取上层的磷脂脂肪酸甲酯,重复上述步骤,合并两次提取液,并用氮气吹干. 将该样品重新溶解于正己烷中后进行样品分析测定.
样品的测定工作由中国科学院城市环境研究所宁波观测研究站完成(http://www.iue.cas.cn/nb/). 具体步骤为:用气相色谱仪(Trace GC
1310 +GC IsoLink II连ConFlo IV)进行测定. 用峰面积和内标曲线法对脂肪酸进行定量,对微生物的鉴定由MIDI SHERLOCKS微生物鉴定系统完成. 利用气相色谱-燃烧法-稳定同位素比质谱仪(GC-C-IRMS)对单体PLFA的δ13C值进行测定.本研究中主要得到18个PLFA单体种类,表征微生物的PLFA标志物见表2.
-
磷脂脂肪酸含量[25]:
式中,AF和AS为样品和内标物(C19:0)的峰面积;WS为内标物(C19:0)的质量(μg);W为烘干土的重量(g);M为磷脂脂肪酸单体的相对分子质量(g·mol−1).
δ13C值为[20]:
式中,Rsample为样品13C/12C比值;Rstandard为标准品(PDB)13C/12C比值.
PLFA单体中13C的量为:
式中,(atom%13C) lable和(atom%13C) unlable分别为添加十六烷样品和对照样品中PLFA单体的atom%13C;Mi为标记样品中PLFA单体的碳含量(ng·g−1).
每种单体PLFA中利用十六烷碳所占比例为[18]:
式中,Fi为每种单体PLFA中利用十六烷碳所占比例;δ13CPLFA,total和δ13CPLFA,control分别为添加十六烷样品和对照样品中单体PLFA的δ13C值;δ13Chexadecane和δ13Csoil分别为十六烷和土壤的δ13C值.
-
利用SPSS 27. 0 软件进行数据的方差分析,不同处理间的显著性差异水平采用Duncan 法进行多重比较,显著水平为P<0.05. 采用 Origin 2022 软件绘制图表. 数据表示为平均值±标准差.
-
经过30 d的自然消减作用,土壤中的十六烷
5000 mg·kg−1降低至4693 mg·kg−1,十六烷的自然降解率为6.14%,说明土著微生物具有降解烷烃的能力. 在0—3 d时十六烷的降解速率为69.00 mg·(kg·d)−1;4—30 d十六烷的降解速率为3.70 mg·(kg·d)−1,说明污染初期十六烷的自然消减速率高于后期(表3).有文献报道土壤中的十六烷可通过生物降解、土壤吸附和化学氧化去除,但是由于其性质稳定属于难挥发有机物,因此较难通过挥发作用去除土壤中十六烷[27]. 化学氧化修复石油污染土壤需要氧化剂或通过改变反应条件驱动[28],有研究发现石油烃的去除主要通过生物降解进行[29]. 在本研究中没有添加氧化剂或改变反应条件等措施,因此污染初期十六烷的降解速率是后期的18.6倍,可能是由于添加十六烷使微生物获得充足的碳源,在污染初期十六烷经过土著微生物的降解作用得以快速去除. 后期可能是因为微生物间相互竞争、营养失衡,导致十六烷的降解速率较低.
-
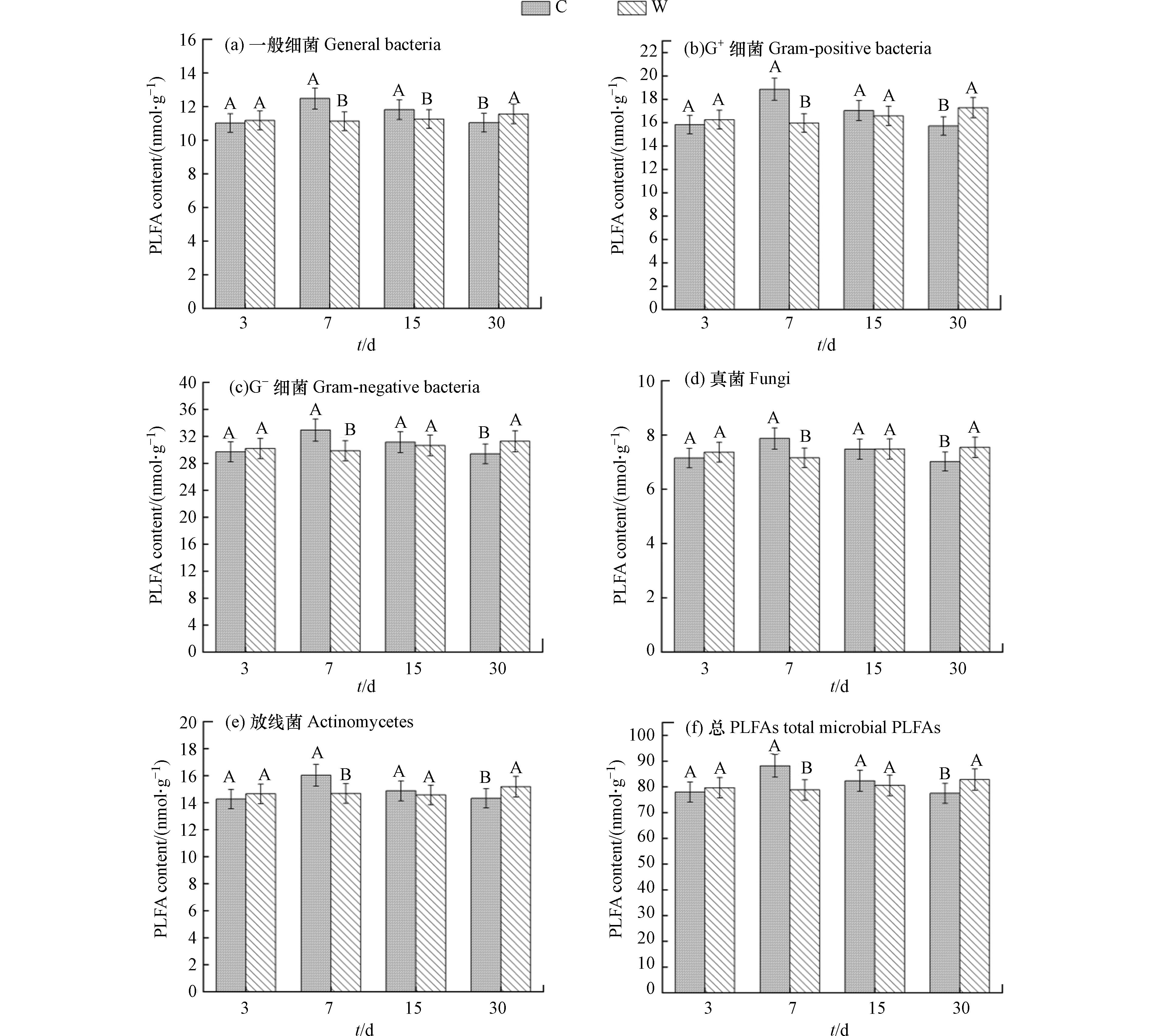
污染土壤中各微生物类群(一般细菌、G+细菌、G—细菌、真菌和放线菌)组成及变化见图1,在土壤受到烷烃污染3 d时(初期),与未受污染的土壤中各微生物类群相比,污染土壤中各PLFAs含量无显著变化;在污染的第7 天(前期),污染土壤中各微生物类群的PLFAs含量显著降低.
在未受污染的土壤中,一般细菌、G+细菌、G—细菌、真菌和放线菌的PLFAs含量分别为12.48、18.86、32.92、7.87、16.02 nmol·g−1;在污染土壤中各微生物类群的PLFAs含量分别为11.13、15.97、29.86、7.16、14.68 nmol·g−1;污染15 d时(中期),除一般细菌外,污染土壤中的其他各微生物类群包括G+细菌、G—细菌、真菌、放线菌的PLFAs含量与洁净土壤无显著性差异;污染30 d时(后期),土壤中各微生物类群的PLFAs含量显著高于未污染土壤(一般细菌、G+细菌、G—细菌、真菌和放线菌的PLFAs含量在土壤中分别为11.05、15.72、29.40、7.02、14.32 nmol·g−1,在污染土壤中分别为11.56、17.29、31.26、7.54、15.18 nmol·g−1). 总体上,与洁净土壤相比,土壤受到
5000 mg·kg−1的十六烷污染后,各微生物类群活性随时间变化的趋势为:污染初期(3 d)微生物活性无明显变化——污染前期(7 d)其活性显著降低——污染中期(15 d)微生物适应烷烃胁迫,开始利用十六烷作为底物碳源进行生长代谢——污染后期(30 d)微生物生长代谢能力增强,其活性显著高于洁净土壤.污染土壤中PLFA单体含量的变化见图2. 总体上,土壤样品中各微生物类群的磷脂脂肪酸含量依次为:G—细菌>G+细菌>放线菌>一般细菌>真菌(图1),污染土壤和洁净土壤中的PLFA单体含量分布具有相似性(图2),一般细菌16:0、G—细菌18:1ω7c、真菌18:1ω9c和放线菌10Me16:0单体的PLFA含量最高,分别为9.48、11.04、7.54、9.91 nmol·g−1. 与未受污染土壤(C)相比,重度烷烃胁迫条件下土壤(W)中PLFA单体含量总体上呈增加趋势(图2). 在污染土壤中显著增加的单体主要有8种,分别是一般细菌16:0,G+细菌i15:0、a17:0,G—细菌16:1ω7c、18:1ω7c、cy19:0ω7c,真菌18:1ω9c和放线菌10Me16:0.
表4为洁净土壤和污染土壤中细菌/真菌、革兰氏阳性菌/革兰氏阴性菌、革兰氏阳性菌异构/反异构的PLFA比值变化情况. 洁净土壤和污染土壤的G+/G—的PLFA比值约为0.55,且随时间无显著变化. 洁净土壤中的G+异构/G+反异构PLFA比值在整个培养期间无显著变化(比值约为1.96);污染土壤中的G+异构/G+反异构PLFA比值在培养前期和中期无显著变化(比值约为1.96),但在培养后期出现明显降低(比值为1.75). 培养3 d时,细菌/真菌的PLFA比值无明显差异;在培养7 d时,比值均有明显增加,且洁净土壤中的增幅更大;在培养15 d时,比值均有明显降低,且洁净土壤中的降幅更大;在培养30 d时,洁净土壤中该比值无显著变化,而污染土壤中显著增加,此时两个土壤样品的细菌/真菌的PLFA比值无明显差异.
污染土壤中不同微生物类群对十六烷的响应情况存在差异. 其中G+菌PLFAs的增加幅度最大(图1),但G+/G—的PLFAs比值无显著变化(表4). G+ / G—能够反映土壤的养分状况,可以揭示土壤微生物对有机碳的获取,比值越高则表示可利用的有机碳越少,在农业土壤中该比值多为大于1[30 − 31]. 本文中未受污染土壤与受污染土壤中的G+/G—的PLFAs比值约为0.55,且不随污染时间的增加而变化. 可能是由于土壤采自陕北黄土生态脆弱区,土壤贫瘠养分含量低,而G—细菌更能适应不利条件[30],因此其数量更占优势. 受烷烃污染的土壤由于添加了外源碳,G—细菌比G+细菌抵抗烷烃胁迫条件的能力更强,对有机碳的获取更多[31 − 32],故在数量上G—细菌也比G+细菌更多. 与前人研究结果一致,在石油污染土壤中,石油烃的降解菌群主要是革兰氏阴性菌中的变形菌[33 − 35],该研究认为G—细菌在石油胁迫条件下能够更快的适应并增长繁殖.
袁庆叶等[36]的研究发现石油污染显著增加了细菌的PLFAs含量,对真菌PLFAs无显著影响. 该研究中,土壤受到十六烷污染后,细菌和真菌的PLFAs含量均增加,可能是由于细菌和真菌均可利用烷烃为底物进行生长代谢所致[37 − 38]. 在污染后期细菌/真菌的PLFAs比值有所增加(表4),说明细菌数量增幅大于真菌. 细菌/真菌的PLFAs比值的增加说明土壤生态系统的稳定性增强[39]. 土壤中G+异构/G+反异构(iN:0∶aN:0)的PLFAs比值可以揭示土壤中的营养情况及微生物面对的环境压力[12]. 受到十六烷污染后,土壤中G+异构/G+反异构的PLFAs比值显著降低(表4),可能由于土壤微生物受烷烃胁迫,且外加碳源使土壤的碳氮比失衡,营养利用率较低[38].
-
污染土壤中各微生物类群的13C含量见图3. 根据图3,除G—细菌的13C-PLFA含量在培养后期(30 d)减少外,G+细菌、真菌、一般细菌的13C-PLFA含量在培养期内表现为随时间变化增加趋势. 其中G+细菌的13C-PLFA含量最多,放线菌的13C-PLFA含量最少,G—细菌、真菌、一般细菌的13C-PLFA含量相差不大. 总体上,各微生物类群13C-PLFA的含量呈现G+细菌>真菌>一般细菌>G—细菌>放线菌的变化趋势,依次为27.42、18.61、15.88、12.32、6.88 ng·kg−1. 烷烃污染土壤中由这五种微生物类群共同协作以促进烷烃的同化代谢,其中能够利用十六烷的主要微生物类群为革兰氏阳性菌,其次是真菌,而放线菌对十六烷的利用较少.
在烷烃污染土壤中,可以利用13C—十六烷的微生物主要有真菌18:1ω9c,G+细菌a15:0、i15:0,G—细菌16:1ω7c、16:1ω5c,一般细菌16:0和放线菌10Me16:0(图4). 重度烷烃胁迫条件下土壤中的十六烷同化为微生物各类群13C-PLFA单体的含量总体呈增加趋势. 培养前期(7 d),各单体13C含量较低且增长缓慢,但到培养中后期时(15—30 d),各单体的13C含量迅速增加. 由此可见,整个培养期间,土壤受到十六烷污染后,土壤中的真菌、G—细菌、一般细菌和放线菌表现出逐渐适应十六烷的毒性,到中后期利用十六烷为底物碳源进行生长代谢的过程.
通常,土壤有机质和外源碳为微生物生长提供碳源[20],该研究中外加的碳源为正十六烷,尽管在烷烃胁迫条件下G—细菌的数量更占优势(图1-c;图2),但13C-PLFA 的测定结果表明烷烃污染土壤中利用正十六烷碳的主要微生物类群为G+细菌,其次为真菌(图4和图5). 然而,在培养初前期(0—7 d),G—细菌对烷烃的代谢发挥了主要作用,随着培养时间的延长,G+细菌和真菌逐渐成为代谢十六烷的主要类群. Nicolas等[31]发现G—细菌容易利用简单的碳化合物(如烷基化合物)进行生长代谢,而G+细菌更擅于利用较复杂的碳化合物(如羰基、芳基和酮类化合物). Stefano等[40]发现由于真菌的生长方式,其菌丝网络能够到达其他菌群难以到达的土壤区域,所以真菌是降解长链脂肪烃的主要成员. 有研究认为,r—策略微生物(主要是变形菌)生长迅速,多利用不稳定、易分解的碳组分进行生长代谢,而k—策略微生物多利用不易分解的碳组分[41 − 42]. 在外源碳输入后的短时间内改变土壤中原有的有机碳矿化过程的现象[42]. r—策略微生物的激发效应主要表现为表观激发效应,是由于土壤微生物利用外源碳为底物提高了生长代谢水平而形成;k—策略微生物则主导真实激发效应,其反映了土壤有机质分解的实际变化过程[42 − 43]. 但也有研究发现,添加单独的外源碳使得真菌数量显著增加,而总微生物量和细菌数量降低,形成正向激发效应[44]. 这可能是由于输入外源碳的碳氮比更适于k—策略微生物(主要是真菌)的生长[45].
该研究与文献[18,33,36]报道的结果一致,受烷烃污染前期(7 d),G—细菌作为r—策略微生物占主导(占13C-PLFA总量的43.40%),表现为表观激发效应;而在污染后期(30 d)对烷烃的降解出现真实激发效应,真菌和多数G+细菌作为k—策略微生物发挥主要作用(分别占13C-PLFA总量的22.95%和33.81%). 此外,一般细菌对十六烷碳利用的贡献随时间变化而逐渐增加(图5),由培养开始时(0 d)占13C-PLFA总量的0.41%到培养后期(30 d)增至19.57%. 而放线菌对十六烷碳利用的贡献最小,除培养中期外,其他时期在13C-PLFA总量占比无显著变化(8.48%—8.93%). 出现该现象的原因可能是在培养中期,各微生物类群均已适应了烷烃胁迫条件,竞争较大,使得本对十六烷利用不高的放线菌更不占优势[46]. 总体而言,一般细菌和真菌随培养时间的增加逐渐适应十六烷的毒性[38],G—细菌在培养前期对十六烷有一定耐受性[47],而十六烷对放线菌无显著影响. 不同微生物类群对来自十六烷碳利用的贡献率占比有所差异,说明土壤中不同微生物类群对十六烷碳的偏好程度和同化能力不同.
-
土壤受到十六烷的污染后,一方面,土壤微生物类群对于烃类污染物发生“应激响应”,导致其活性发生变化[1,48]. 另一方面,一些微生物类群对十六烷具有降解代谢作用[47,49]. 利用13C-PLFA-SIP对不同微生物类群的活性和对十六烷的代谢能力进行测定,微生物的PLFA含量越高,表示其活性越大[13 − 14]. 微生物的13C-PLFA含量越高,表示对十六烷的代谢能力越强[19]. 本文中,土壤受到十六烷污染后,各微生物类群PLFA含量由高到低依次为G—细菌>G+细菌>放线菌>一般细菌>真菌(图1);各微生物类群13C-PLFA含量由高到低依次为G+细菌>真菌>一般细菌>G—细菌>放线菌(图3和图5). 各微生物类群的PLFA含量与13C-PLFA含量顺序不一致,说明土壤中不同微生物类群对十六烷代谢能力的大小不仅是由其活性决定的,还可能受到微生物类群数量、菌群结构多样性等其他因素的影响作用[3,50],因此通过测定微生物的活性预测其对污染物的代谢能力存在较大偏差.
-
与未受烷烃污染的土壤相比,各微生物类群对十六烷毒性的响应表现为迟滞效应,在污染初期时各类群无明显变化,随污染时长的增加,各微生物类群表现出“毒性响应(污染前期)—毒性适应(污染中期)—生长代谢(污染后期)”的应激过程. 重度烷烃胁迫条件下,土壤中的土著微生物有降解烷烃的能力,但各微生物类群对十六烷的利用程度差异明显. 在污染前期G—细菌对十六烷利用的贡献最大,污染后期G+细菌和真菌占主导地位. 土壤中的G+细菌对十六烷的污染响应最为敏感,且对十六烷的同化代谢能力最强. 土壤中不同微生物类群对十六烷的同化能力和利用程度的不同,对于后续优化污染物的功能降解菌生境以修复污染土壤具有重要的指示作用.
烷烃胁迫下土壤微生物类群的应激响应及其降解特性
The response of soil microbial groups to alkane stress and hydrocarbon biodegradation characteristics
-
摘要: 污染物胁迫条件下土壤微生物会发生应激响应,然而目前对于烷烃污染土壤中微生物类群应激响应的时效性尚不清楚. 论文以人工配制的十六烷污染黄绵土为对象,利用磷脂脂肪酸-稳定同位素技术(phospholipid fatty acid technology-stable isotope probing,PLFA-SIP)探究了烷烃污染对黄绵土中生物群落组成和结构影响的时效性及烷烃降解情况. 结果表明,土壤土著微生物对十六烷有一定的降解作用,培养30 d时,十六烷含量由
5000 mg·kg−1降低至4693 mg·kg−1,降解率为6.14%. 土壤中各微生物类群活性在受到十六烷污染3 d时无明显变化,随污染时长的增加,各微生物类群表现出“毒性响应(污染7 d)—毒性适应(污染15 d)—生长代谢(污染30 d)”的应激过程. 在污染前期(污染7 d时),G—细菌的13C-PLFA含量为14.52 ng·kg−1,占总细菌13C-PLFA总量的43.40%,说明在污染前期G—细菌是土壤中存在的可利用十六烷的主要优势类群;污染中后期(污染15—30 d),G+细菌和真菌的13C-PLFA含量分别占13C总量的33.81%和22.95%,说明污染后期G+细菌和真菌对十六烷的降解代谢起到主要作用. 研究表明土壤中十六烷的降解需要各微生物类群的协同代谢作用,污染前期主要由r—策略微生物(G—细菌)降解十六烷,污染后期则主要由k—策略微生物(G+细菌和真菌)执行降解功能. 研究可为探明土壤微生物类群对烷烃污染响应的时效性及不同微生物类群对污染物的利用特征提供科学依据.Abstract: Soil microorganisms present the stress response to the contaminants. However, the timeliness response of the microbial community under hydrocarbon stress is unclear. In this study, the loessal soil collecting from the northern of Shaanxi province of China was used to study the effects of hexadecane pollution on the compositions and structures of microbial communities using phospholipid fatty acid technology-stable isotope probing (PLFA-SIP) techniques. Results showed that the indigenous microorganisms in the polluted soil had certain degradation capacity toward hexadecane through natural attenuation. After 30 days of incubation, the hexadecane content decreased from5000 mg·kg−1 to4693 mg·kg−1, with a degradation rate of 6.14%. Soil microbial community had no significant changes at the 3th days of pollution and the toxic effects appeared at the 7th days. Then soil microorganisms was adapted to alkane stress and finally exhibited growth after 15th days of incubation. In the early stage of hexadecane pollution (7 days of pollution periods), the 13C-PLFA content of G− bacteria was 14.52 ng·kg−1 which accounting for 43.40% of the total 13C-PLFA. In the medium and later stages of pollution (15—30 days of pollution durations), the 13C-PLFA content of G+ bacteria and fungi accounted for 33.81% and 22.95% of the total 13C-PLFA, respectively. Result indicated that the G− bacteria were the dominant microorganisms which could utilize hexadecane in the soil during the early stage, but the G+ bacteria and fungi played a major role for hexadecane degradation in the later stages of pollution. Studies suggested that hexadecane biodegradation in soil involved the synergistic metabolism of various microbiomes. In the early stage of incubation, r-strategic microorganisms (G—bacteria) mainly utilized easily degradable carbon components, and k-strategic microorganisms (composed of G+ bacteria and fungi) utilized the refractory carbon components in the later stage. This study provided a theoretical basis for the pollution timeliness and the utilization characteristics towards alkane pollution by different microbial communities. -

-
表 1 供试土壤的基本理化性质
Table 1. Basic physical and chemical properties of the soil
指标
Index含水率/%
Moisture contentpH 总有机碳/(mg·kg−1)
Total organic carbon总氮/(mg·kg−1)
Total nitrogen氨氮/(mg·kg−1)
Ammonia nitrogen硝氮/(mg·kg−1)
Nitrate nitrogen全硫/(mg·kg−1)
Total sulphur测定值 12.3±0.8 8.30±0.10 5275 ±127400.1±12.5 4.11±0.16 4.01±0.15 120±10 表 2 表征不同种群微生物的磷脂脂肪酸标志物
Table 2. Phospholipid fatty acid (PLFA) biomarkers of different microbial community biomass
微生物类型
Microbial communityPLFA标志物
Phospholipid fatty acid biomarkers参考文献
References一般细菌
General Bacteria16:0、18:0 [18, 20] 革兰氏阳性菌
Gram-positive bacteriai15:0、a15:0、i16:0、i17:0、a17:0 [20, 25] 革兰氏阴性菌
Gram-negative bacteria16:1ω5c、16:1ω7c、cy17:0ω7c、18:1ω5c、18:1ω7c、cy17:0ω7c、cy19:0ω7c [18, 25 − 26] 真菌
Fungi18:1ω9c [25 − 26] 放线菌
Actinomycetes10Me16: 0 、10Me17: 0 、10Me18: 0、10Me17:1ω7c [18, 20, 25] 表 3 十六烷在土壤中的自然消减
Table 3. The hexadecane contents and degradation rates in hexadecane-added soil
样品
Sample十六烷含量/(mg·kg−1)
Hexadecane content降解率/%
Degradation efficiency降解速率/(mg·(kg·d)−1)
Degradation rate0 d 3 d 30 d 3 d 30 d 0—3 d 4—30 d 污染土壤 5000 ±504793 ±884693 ±974.14 6.14 69.00 3.70 表 4 不同处理土壤细菌/真菌、革兰氏阳性菌/革兰氏阴性菌、革兰氏阳性菌异构/反异构的变化
Table 4. Ratios of bacteria to fungi, gram-positive to -negative bacteria and isomeric to reverse isomeric gram-positive bacteria under different soils
处理
Treat-ments细菌/真菌
(Bacteria : Fungi)G+/G— G+异构/G+反异构
(iN:0∶aN:0)3d 7d 15d 30d 3d 7d 15d 30d 3d 7d 15d 30d 洁净
土壤7.91±0.05Ab 8.17±0.05Aa 8.02±0.04Ab 8.00±0.06Ab 0.53±0.03Aa 0.57±0.03Aa 0.55±0.02Aa 0.53±0.03Aa 1.96±0.05Aa 1.97±0.05Aa 1.98±0.04Aa 1.94±0.04Aa 污染
土壤7.82±0.10Ab 7.96±0.05Ba 7.81±0.09Bb 7.97±0.06Aa 0.54±0.02Aa 0.53±0.03Aa 0.54±0.02Aa 0.55±0.03Aa 1.95±0.04Aa 1.94±0.03Aa 1.98±0.04Aa 1.75±0.03Bb 注:同行不同小写字母表示同一处理不同培养时间差异显著(P<0.05);同列不同大写字母表示同一培养时间不同处理差异显著(P<0.05).
Note: the lowercase letters in the same treatments indicated significant differences of incubation time for the same treatment (P<0.05); the capital letters in the same tandem indicated significant differences of treatment at the same incubation time (P<0.05) -
[1] 杨萌青, 李立明, 李川, 等. 石油污染土壤微生物群落结构与分布特性研究[J]. 环境科学, 2013, 34(2): 789-794. YANG M Q, LI L M, LI C, et al. Microbial community structure and distribution characteristics in oil contaminated soil[J]. Environmental Science, 2013, 34(2): 789-794(in Chinese).
[2] PACWA-PŁOCINICZAK M, PŁAZA G A, PIOTROWSKA-SEGET Z. Monitoring the changes in a bacterial community in petroleum-polluted soil bioaugmented with hydrocarbon-degrading strains[J]. Applied Soil Ecology, 2016, 105: 76-85. doi: 10.1016/j.apsoil.2016.04.005 [3] WU M L, DICK W A, LI W, et al. Bioaugmentation and biostimulation of hydrocarbon degradation and the microbial community in a petroleum-contaminated soil[J]. International Biodeterioration & Biodegradation, 2016, 107: 158-164. [4] SAFDARI M S, KARIMINIA H R, RAHMATI M, et al. Development of bioreactors for comparative study of natural attenuation, biostimulation, and bioaugmentation of petroleum-hydrocarbon contaminated soil[J]. Journal of Hazardous Materials, 2018, 342: 270-278. doi: 10.1016/j.jhazmat.2017.08.044 [5] GAO Y C, DU J H, BAHAR M M, et al. Metagenomics analysis identifies nitrogen metabolic pathway in bioremediation of diesel contaminated soil[J]. Chemosphere, 2021, 271: 129566. doi: 10.1016/j.chemosphere.2021.129566 [6] 刘五星, 骆永明, 滕应, 等. 石油污染土壤的生态风险评价和生物修复 Ⅱ. 石油污染土壤的理化性质和微生物生态变化研究[J]. 土壤学报, 2007, 44(5): 848-853. doi: 10.3321/j.issn:0564-3929.2007.05.011 LIU W X, LUO Y M, TENG Y, et al. Eco-risk assessment and bioremediation of petroleum contaminated soil ⅱ. changes in physico-chemical properties and microbial ecology of petroleum contaminated soil[J]. Acta Pedologica Sinica, 2007, 44(5): 848-853(in Chinese). doi: 10.3321/j.issn:0564-3929.2007.05.011
[7] BUNDY J G, PATON G I, CAMPBELL C D. Combined microbial community level and single species biosensor responses to monitor recovery of oil polluted soil[J]. Soil Biology and Biochemistry, 2004, 36(7): 1149-1159. doi: 10.1016/j.soilbio.2004.02.025 [8] 蔡萍萍, 宁卓, 何泽, 等. 采油井场土壤微生物群落结构分布[J]. 环境科学, 2018, 39(7): 3329-3338. CAI P P, NING Z, HE Z, et al. Microbial community distributions in soils of an oil exploitation site[J]. Environmental Science, 2018, 39(7): 3329-3338(in Chinese).
[9] 贾建丽, 李广贺, 钟毅. 石油污染土壤生物修复中试系统对微生物特性的影响[J]. 环境科学研究, 2007, 20(5): 115-118 JIA J L, LI G H, ZHONG Y. The influence of the microbial properties of oil contaminated soils in the bioremediation pilot systems[J]. Research of Environmental Sciences, 2007, 20(5): 115-118(in Chinese)
[10] POLYAK Y M, BAKINA L G, CHUGUNOVA M V, et al. Effect of remediation strategies on biological activity of oil-contaminated soil - A field study[J]. International Biodeterioration & Biodegradation, 2018, 126: 57-68. [11] ZHANG Y Y, ZHENG N G, WANG J, et al. High turnover rate of free phospholipids in soil confirms the classic hypothesis of PLFA methodology[J]. Soil Biology and Biochemistry, 2019, 135: 323-330. doi: 10.1016/j.soilbio.2019.05.023 [12] 赵美玲, 张一鸣, 张志麒, 等. 神农架大九湖不同生境表土磷脂脂肪酸揭示的微生物群落结构差异[J]. 地球科学, 2020, 45(6): 1877-1886. ZHAO M L, ZHANG Y M, ZHANG Z Q, et al. Comparison of microbial community in topsoil among different habitats in Dajiuhu, Hubei Province: Evidence from phospholipid fatty acids[J]. Earth Science, 2020, 45(6): 1877-1886 (in Chinese).
[13] WANG J, CHAPMAN S J, YAO H Y. Incorporation of 13C-labelled rice rhizodeposition into soil microbial communities under different fertilizer applications[J]. Applied Soil Ecology, 2016, 101: 11-19. doi: 10.1016/j.apsoil.2016.01.010 [14] 左易灵, 贺学礼, 王少杰, 等. 磷脂脂肪酸(PLFA)法检测蒙古沙冬青根围土壤微生物群落结构[J]. 环境科学, 2016, 37(7): 2705-2713 ZUO Y L, HE X L, WANG S J, et al. Characteristics of soil microbial community structure in the rhizospheric soil of ammopiptanthus mongolicus by phospholipid fatty acid (PLFA)[J]. Environmental Science, 2016, 37(7): 2705-2713(in Chinese)
[15] 王万洁, 侯兴旺, 刘稷燕, 等. 传统稳定同位素技术在环境科学领域的应用及研究进展[J]. 环境化学, 2021, 40(12): 3640-3650. doi: 10.7524/j.issn.0254-6108.2021041601 WANG W J, HOU X W, LIU J Y, et al. Application and research progress of traditional stable isotope technology in environmental science[J]. Environmental Chemistry, 2021, 40(12): 3640-3650(in Chinese). doi: 10.7524/j.issn.0254-6108.2021041601
[16] SILES J A, CAJTHAML T, FROUZ J, et al. Assessment of soil microbial communities involved in cellulose utilization at two contrasting Alpine forest sites[J]. Soil Biology and Biochemistry, 2019, 129: 13-16. doi: 10.1016/j.soilbio.2018.11.004 [17] MARTINEZ-CRUZ K, LEEWIS M C, HERRIOTT I C, et al. Anaerobic oxidation of methane by aerobic methanotrophs in sub-Arctic lake sediments[J]. Science of the Total Environment, 2017, 607/608: 23-31. doi: 10.1016/j.scitotenv.2017.06.187 [18] 邓少虹, 郑小东, 毛婉琼, 等. 稻田与旱地土壤中真菌和细菌对秸秆碳的利用特征[J]. 环境科学, 2022, 43(2): 1069-1076. DENG S H, ZHENG X D, MAO W Q, et al. Characteristics of microbial utilization for crop residue-derived C in paddy and upland soils[J]. Environmental Science, 2022, 43(2): 1069-1076(in Chinese).
[19] XU Y D, SUN L J, LAL R, et al. Microbial assimilation dynamics differs but total mineralization from added root and shoot residues is similar in agricultural Alfisols[J]. Soil Biology and Biochemistry, 2020, 148: 107901. doi: 10.1016/j.soilbio.2020.107901 [20] 徐英德, 孙良杰, 王阳, 等. 土壤微生物群落对玉米根茬和茎叶残体碳的利用特征[J]. 中国环境科学, 2020, 40(10): 4504-4513. XU Y D, SUN L J, WANG Y, et al. Characteristics of microbial utilization of maize root- and straw derived carbon[J]. China Environmental Science, 2020, 40(10): 4504-4513 (in Chinese).
[21] 许殷瑞, 吴蔓莉, 王丽, 等. 陕北石油污染土壤微生物种群变化及影响因素[J]. 中国环境科学, 2021, 41(9): 4349-4359 doi: 10.3969/j.issn.1000-6923.2021.09.042 XU Y R, WU M L, WANG L, et al. The influences of petroleum pollution on the microbial population distribution in Northern Shaanxi Province of China[J]. China Environmental Science, 2021, 41(9): 4349-4359(in Chinese) doi: 10.3969/j.issn.1000-6923.2021.09.042
[22] 鲍士旦. 土壤农化分析[M]. 3版. 北京: 中国农业出版社, 2000. BAO S D. Soil and agricultural chemistry analysis[M]. 3rd ed. Beijing: China Agriculture Press, 2000(in Chinese).
[23] 邢玥, 吴蔓莉, 刘泽梁, 等. 高浓度含油废水中不同组分烃的生物强化去除特性[J]. 环境化学, 2023, 42(2): 567-574. doi: 10.7524/j.issn.0254-6108.2021102505 XING Y, WU M L, LIU Z L, et al. Removal efficiencies of different components of crude oil by bioaugmentation[J]. Environmental Chemistry, 2023, 42(2): 567-574 (in Chinese). doi: 10.7524/j.issn.0254-6108.2021102505
[24] 叶茜琼, 吴蔓莉, 陈凯丽, 等. 微生物修复油污土壤过程中氮素的变化及菌群生态效应[J]. 环境科学, 2017, 38(2): 728-734. YE X Q, WU M L, CHEN K L, et al. Impacts of bioremediation on microbial communities and different forms of nitrogen in petroleum contaminated soil[J]. Environmental Science, 2017, 38(2): 728-734(in Chinese).
[25] 许殷瑞. 陕北采油区土壤微生物群落结构及对石油烃组分的利用机制[D]. 西安: 西安建筑科技大学 . XU Y R. Soil microbial community structure and utilization mechanism of petroleum hydrocarbon components in northern Shaanxi oil production area [D]. Xi'an: Xi 'an University of Architecture and Technology (in Chinese).
[26] 于颖超, 张心昱, 戴晓琴, 等. 亚热带红壤区森林土壤剖面微生物残体碳分布及影响因素[J]. 生态学报, 2022, 42(3): 1108-1117 YU Y C, ZHANG X Y, DAI X Q, et al. Distributions and influencing factors of microbial residue carbon contents in forest soil profiles in subtropical red soil region[J]. Acta Ecologica Sinica, 2022, 42(3): 1108-1117(in Chinese)
[27] SAMAEI M R, MORTAZAVI S B, BAKHSHI B, et al. Investigating the effects of combined bio-enhancement and bio-stimulation on the cleaning of hexadecane-contaminated soils[J]. Journal of Environmental Chemical Engineering, 2022, 10(1): 106914. doi: 10.1016/j.jece.2021.106914 [28] 郑嘉如, 方战强, 易云强, 等. 基于化学氧化法修复石油烃污染土壤研究进展[J]. 环境化学, 2023, 42(2): 608-626. doi: 10.7524/j.issn.0254-6108.2021092701 ZHENG J R, FANG Z Q, YI Y Q, et al. Research progress on remediation of petroleum hydrocarbon contaminated soil using chemical oxidation[J]. Environmental Chemistry, 2023, 42(2): 608-626(in Chinese). doi: 10.7524/j.issn.0254-6108.2021092701
[29] LIU H, WU M L, GAO H, et al. Crude oil removal by Meyerozyma consortium and nitrogen supplement: Hydrocarbon transformation, nitrogen fate, and enhancement mechanism[J]. Journal of Environmental Chemical Engineering, 2023, 11(1): 109034. doi: 10.1016/j.jece.2022.109034 [30] KOURTEV P S, EHRENFELD J G, HÄGGBLOM M. Experimental analysis of the effect of exotic and native plant species on the structure and function of soil microbial communities[J]. Soil Biology and Biochemistry, 2003, 35(7): 895-905. doi: 10.1016/S0038-0717(03)00120-2 [31] FANIN N, KARDOL P, FARRELL M, et al. The ratio of Gram-positive to Gram-negative bacterial PLFA markers as an indicator of carbon availability in organic soils[J]. Soil Biology and Biochemistry, 2019, 128: 111-114. doi: 10.1016/j.soilbio.2018.10.010 [32] SUN R, THATER B, SHI P, et al. The effect of cowpea (Vigna unguiculata) with root mucilage on phenanthrene (PHE) dissipation and microbial community composition using phospholipid fatty acid (PLFA) analysis and artificial neural network (ANN) modeling[J]. International Biodeterioration & Biodegradation, 2015, 100: 29-37. [33] 吴蔓莉, 李可欣, 侯爽爽, 等. 贫养分低有机质黄绵土中石油烃的生物去除特性及菌群结构变化[J]. 环境科学研究, 2021, 34(8): 1961-1970 doi: 10.13198/j.issn.1001-6929.2021.04.13 WU M L, LI K X, HOU S S, et al. Petroleum hydrocarbon degradation characteristics and microbial community shift by bioremediation in oligotrophic and low organic matter soil[J]. Research of Environmental Sciences, 2021, 34(8): 1961-1970(in Chinese) doi: 10.13198/j.issn.1001-6929.2021.04.13
[34] WU M L, WU J L, ZHANG X H, et al. Effect of bioaugmentation and biostimulation on hydrocarbon degradation and microbial community composition in petroleum-contaminated loessal soil[J]. Chemosphere, 2019, 237: 124456. doi: 10.1016/j.chemosphere.2019.124456 [35] SUJA F, RAHIM F, TAHA M R, et al. Effects of local microbial bioaugmentation and biostimulation on the bioremediation of total petroleum hydrocarbons (TPH) in crude oil contaminated soil based on laboratory and field observations[J]. International Biodeterioration & Biodegradation, 2014, 90: 115-122. [36] 袁庆叶, 安菁, 高俊琴, 等. 芦苇克隆整合对石油污染湿地土壤微生物群落结构和生物量的影响[J]. 生态学报, 2018, 38(1): 215-225. YUAN Q Y, AN J, GAO J Q, et al. Effects of clonal integration of Phragmites australis on the composition and biomass of soil microbial communities in a wetland contaminated by crude oil[J]. Acta Ecologica Sinica, 2018, 38(1): 215-225(in Chinese).
[37] de VRIES F T, HOFFLAND E, van EEKEREN N, et al. Fungal/bacterial ratios in grasslands with contrasting nitrogen management[J]. Soil Biology and Biochemistry, 2006, 38(8): 2092-2103. doi: 10.1016/j.soilbio.2006.01.008 [38] MARSCHNER P, UMAR S, BAUMANN K. The microbial community composition changes rapidly in the early stages of decomposition of wheat residue[J]. Soil Biology and Biochemistry, 2011, 43(2): 445-451. doi: 10.1016/j.soilbio.2010.11.015 [39] 谷晓楠, 贺红士, 陶岩, 等. 长白山土壤微生物群落结构及酶活性随海拔的分布特征与影响因子[J]. 生态学报, 2017, 37(24): 8374-8384. GU X N, HE H S, TAO Y, et al. Soil microbial community structure, enzyme activities, and their influencing factors along different altitudes of Changbai Mountain[J]. Acta Ecologica Sinica, 2017, 37(24): 8374-8384(in Chinese).
[40] COVINO S, D'ANNIBALE A, STAZI S R, et al. Assessment of degradation potential of aliphatic hydrocarbons by autochthonous filamentous fungi from a historically polluted clay soil[J]. Science of the Total Environment, 2015, 505: 545-554. doi: 10.1016/j.scitotenv.2014.10.027 [41] FABIAN J, ZLATANOVIC S, MUTZ M, et al. Fungal–bacterial dynamics and their contribution to terrigenous carbon turnover in relation to organic matter quality[J]. The ISME Journal, 2017, 11(2): 415-425. doi: 10.1038/ismej.2016.131 [42] 魏圆云, 崔丽娟, 张曼胤, 等. 土壤有机碳矿化激发效应的微生物机制研究进展[J]. 生态学杂志, 2019, 38(4): 1202-1211. doi: 10.13292/j.1000-4890.201904.008 WEI Y Y, CUI L J, ZHANG M Y, et al. Research advances in microbial mechanisms underlying priming effect of soil organic carbon mineralization[J]. Chinese Journal of Ecology, 2019, 38(4): 1202-1211(in Chinese). doi: 10.13292/j.1000-4890.201904.008
[43] FONTAINE S, MARIOTTI A, ABBADIE L. The priming effect of organic matter: A question of microbial competition?[J]. Soil Biology and Biochemistry, 2003, 35(6): 837-843. doi: 10.1016/S0038-0717(03)00123-8 [44] BLAGODATSKAYA Е, KUZYAKOV Y. Mechanisms of real and apparent priming effects and their dependence on soil microbial biomass and community structure: Critical review[J]. Biology and Fertility of Soils, 2008, 45(2): 115-131. doi: 10.1007/s00374-008-0334-y [45] MAZZILLI S R, KEMANIAN A R, ERNST O R, et al. Priming of soil organic carbon decomposition induced by corn compared to soybean crops[J]. Soil Biology and Biochemistry, 2014, 75: 273-281. doi: 10.1016/j.soilbio.2014.04.005 [46] GAO H, WU M L, LIU H, et al. Effect of petroleum hydrocarbon pollution levels on the soil microecosystem and ecological function[J]. Environmental Pollution, 2022, 293: 118511. doi: 10.1016/j.envpol.2021.118511 [47] WU M L, LIU Z L, GAO H, et al. Assessment of bioremediation potential of petroleum-contaminated soils from the Shanbei oilfield of China revealed by qPCR and high throughput sequencing[J]. Chemosphere, 2022, 308: 136446. doi: 10.1016/j.chemosphere.2022.136446 [48] 李海兰. 低渗透油藏定向激活石油烃降解菌及其采油机理研究[D]. 北京: 中国石油大学(北京) . LI H L. Study on directional activation of petroleum hydrocarbon degrading bacteria and its oil recovery mechanism in low permeability reservoirs[D]. Beijing: China University of Petroleum(Beiiing) (in Chinese).
[49] ZHEN L S, HU T, LV R, et al. Succession of microbial communities and synergetic effects during bioremediation of petroleum hydrocarbon-contaminated soil enhanced by chemical oxidation[J]. Journal of Hazardous Materials, 2021, 410: 124869. doi: 10.1016/j.jhazmat.2020.124869 [50] GU D C, XIANG X J, WU Y C, et al. Synergy between fungi and bacteria promotes polycyclic aromatic hydrocarbon cometabolism in lignin-amended soil[J]. Journal of Hazardous Materials, 2022, 425: 127958. doi: 10.1016/j.jhazmat.2021.127958 -



 下载:
下载:





